郭鑫瑶, 季晶焱, 程敏, 赵亮, 曹荷清, 王丰, 李小兵, 廖万清, 李文均, 康颖倩*
(1.贵州医科大学 基础医学院 微生物学教研室, 贵州 贵阳 550025; 2.贵州省微生物与人类健康关系研究人才基地 & 贵州省普通高校病原生物学特色重点实验室, 贵州 贵阳 550025; 3.贵州医科大学 环境污染与疾病监控教育部重点实验室, 贵州 贵阳 550025; 4.贵州省烟草科学研究院 贵州省微生物与健康院士工作站, 贵州 贵阳 550000; 5.贵州省赤水河畔酱酒研究中心, 贵州 贵阳 550003; 6.上海市医学真菌分子生物学重点实验室, 上海 200003; 7.海军军医大学长征医院 皮肤科, 上海 200003; 8.中山大学 生命科学学院, 广东 广州 510275)
戈登菌属(Gordonia)由Tsukamura[1]于1971年首次提出,是从肺部疾病患者的痰液或土壤中分离出的棒状细菌,具有好氧、革兰阳性至可变、过氧化氢酶阳性、轻微抗酸、不形成芽孢及非运动性等特点[2]。大多数戈登菌从环境中分离出来,尤其是在红树林、废水处理厂、生物滤器及石油污染过的土壤等地,可以合成多种化合物(如抗菌剂、表面活性剂、碳水化合物活性酶及次级代谢产物等),在石油降解、生物修复和临床医学中有重要的作用[3-6]。有研究表明,大多数细菌处于极端环境时可通过碳水化合物活性酶(carbohydrate-active enzymes,CAZymes)对糖原物质进行代谢,以满足其生存的条件,如处于极冷环境中的节杆菌属(Arthrobacter)可利用CAZymes相关的糖原和海藻糖代谢途径快速的适应环境,还可以通过CAZymes的种类和数量了解总体酶功能和碳水化合物代谢情况[7]。另外,放线菌是产生新的次级代谢产物最重要来源之一,尤其是聚酮合酶(polyketides synthase,PKS)、非核糖体肽合成酶(non-ribosomal peptide synthetase,NRPS)、萜烯(terpene)及翻译后修饰肽类(post-translationally modified peptides,RiPP)等[8]。虽然基因测序和生物信息学分析水平的发展迅速,但是对戈登菌属基因组层面的研究于2010年才开始[9],CAZymes和次级代谢产物方面的研究较缺乏,因此本研究对美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)下载的41株戈登菌的全基因组进行全基因组特征分析、功能注释、泛基因组分析,并挖掘戈登菌属产生CAZymes、生物合成基因簇以及产次级代谢产物的能力,以期探究戈登菌属的应用潜力和环境适应性等。
1 材料与方法
1.1 实验菌株与戈登菌基因组数据来源
菌株来源于千叶大学病原真菌与微生物毒理研究中心(Institute of Food Microbiology, IFM),分别为G.bronchialis3株IFM10331、IFM150及B0002、G.terraeIFM161、G.amaraceIFM210、G.effuseIFM10200、G.araiiIFM10211、G.rubripertinctaIFM10321、G.iteransIFM10348、G.polyisoprenivoransIFM10351、G.alkanivoransIFM10352、G.amicalisIFM10353、G.desulfuricansIFM10355、G.namibiensisIFM10356、G.rhizospheraIFM10357、G.sihwensisIFM10619、G.westfalicaIFM10629、G.paraffinivoransIFM10631及G.sptuiB0003,菌株保存在病原生物学特色重点实验室;戈登菌全基因组数据主要是从 NCBI下载,优先下载全基因组组装完整的数据,共下载41个戈登菌的全基因组数据,其中包括40个已明确分类地位的戈登菌和1株尚未明确的Gordoniasp.,5株来源于人体,16株来源于土壤,4株来源于污泥,5株来源于水,4株来源于生物滤器,其余6株分别来自动物粪便、动物直肠、水稻、空气或未明确来源,具体情况见表1。
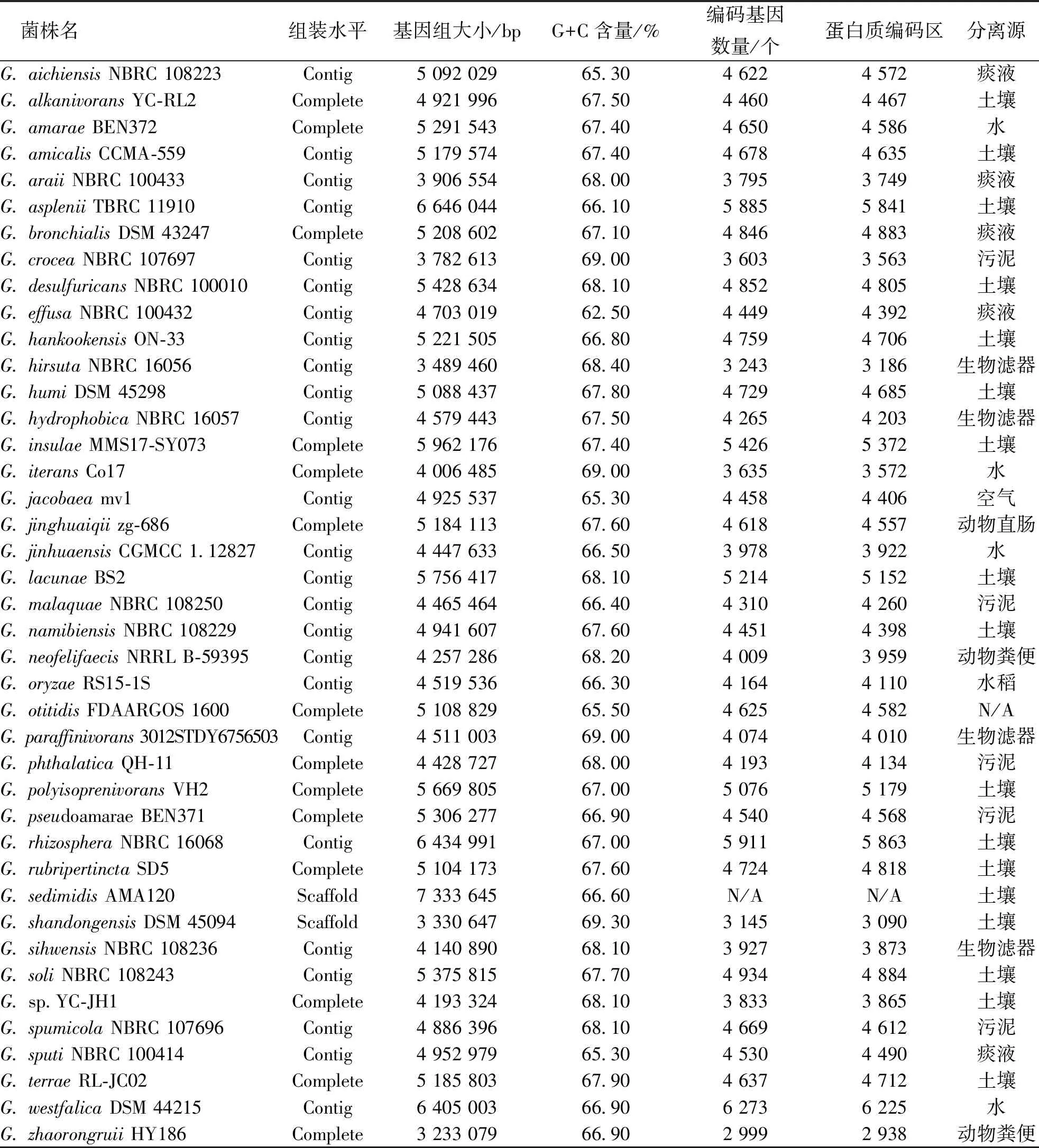
表1 41株戈登菌全基因组基本信息
1.2 研究方法
1.2.1系统发育学分析 19株实验室保存的戈登菌接种于脑心浸出液琼脂培养基,37 ℃培养48~72 h,采用3区划线法分离单个菌落,挑取单个菌落进行传代培养;根据参考文献[10]方法对培养的戈登菌进行基因组DNA提取,采用16S小亚基核糖体RNA(16S ribosomal RNA,16S rRNA)通用引物进行聚合酶链反应(polymerase chain reaction,PCR)扩增[11],PCR产物送上海生工生物工程股份有限公司进行测序;采用barrnap软件(https://github.com/tseemann/barrnap)提取从NCBI数据库中下载的部分戈登菌全基因组中的16S rRNA基因序列;所有序列经过分子进化遗传学分析软件(molecular evolutionary genetics analysis,MEGA)剪切处理后,利用邻位相接法(neighbor-joining,NJ)构建系统发育树,以诺卡菌标准菌株Nocardiaasteroides作为外群,自展检验(bootstrap)重复抽样1 000次;系统发育树美化在iTOL平台[12]完成。采用FastANI(https://github.com/ParBLiSS/FastANI)对41株戈登菌的基因组两两比较,计算对应的平均核苷酸多态性(average nucleotide identity,ANI),采用在线分析软件(http://ggdc.dsmz.de/distcalc2.php)计算戈登菌属之间DNA-DNA杂交值(DNA-DNA hybridization,DDH),使用HeatMap绘制热图。
1.2.2泛基因组学特征分析 采用软件Mugssy对戈登菌基因组序列进行比对,结合原核生物泛基因组学分析的自动化软件(pan-genomes analysis pipeline,PGAP)对戈登菌进行全基因组和核心基因组分析,通过Gene Family(GF)方法对基因组进行聚类,将结果导入PanGP软件,根据希普斯定律(Heap's law)和指数定律(exponential law)分别拟合泛基因组和核心基因组特征曲线[13]。
1.2.3基因组功能分析 利用直系同源蛋白分组比对数据库(evolutionary genealogy of genes: non-supervised orthologous groups,eggNOG)对戈登菌预测到的基因序列进行直系同源基因簇(clusters of orthologous groups,COG)功能注释;CAZymes的注释利用CAZymes数据库(http://www.cazy.org/);使用基因组次级代谢产物分析工具(antibiotics and secondary metabolite analysis shell,antiSMASH;https://antismash.secondarymetabolites.org/)进行生物合成基因簇预测,再通过生物合成基因簇的标准化信息库(minimum information about a biosynthetic gene cluster,MIBiG)[14]预测检测到的次级代谢产物合成基因簇与已知或新的生物活性化合物的产生之间的关联。
1.3 统计学分析

2 结果
2.1 41株戈登菌全基因组特征分析
整体而言,戈登菌基因组大小为3 233 079(G.zhaorongruiiHY186)~6 646 044 bp(G.aspleniiTBRC 11910),均数为4 941 636.42 bp;土壤来源组基因组均数最大(5 398 505.47±886 760.99) bp,生物滤器来源组基因组最小(4 180 199.00±432 288.28) bp,且2组间差异有统计学意义(P<0.05),其他各菌株组间的基因组大小比较、差异无统计学意义(P>0.05);戈登菌基因组G+C含量为62.50%(G.effuseNBRC 100432)~69.30%(G.shandongensisDSM 45094),均数67.25%,是属于高GC含量的一类微生物;预测到的基因数量为2 999(G.zhaorongruiiHY186)~6 237(G.aspleniiTBRC 11910),均数为4 479.25,蛋白质编码区(coding sequence,CDS)均数为4 445.60,具有丰富的基因组多样性(表1)。
2.2 系统发育学分析
41株戈登菌的ANI值 和DDH值评估结果显示,40株已知种的戈登菌两两之间ANI值介于70%~90%,分析软件 GGDC结果均显示为该40株戈登菌两两之间的DDH值为20%~30%,然而未进行准确分类的1株Gordoniasp.YC-JH1与G.sihwensisNBRC 108236之间的ANI值和DDH值均为99.9%(图1);通过16S rRNA基因序列构建的系统发育树可发现(图2),戈登菌属分类地位明确,且Gordoniasp.YC-JH1与G.sihwensis处于同一个分支之中,进一步说明Gordoniasp.YC-JH1是属于G.sihwensis的一个菌株。戈登菌系统发育树分为5个分支,在第5支中的菌株主要来源于土壤,其他分支中的菌株来源较为丰富。
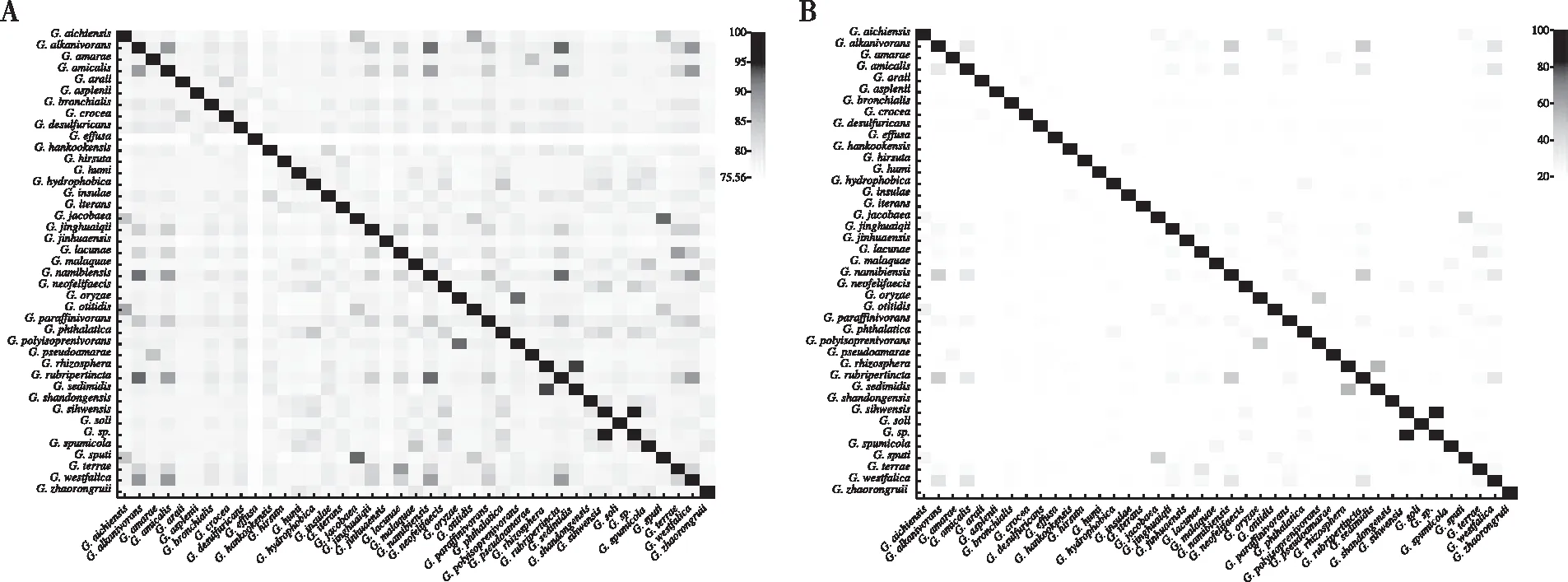
注:A、B分别为ANI和DDH分析结果;浅灰至深灰表示基因组之间相似性越来越大。
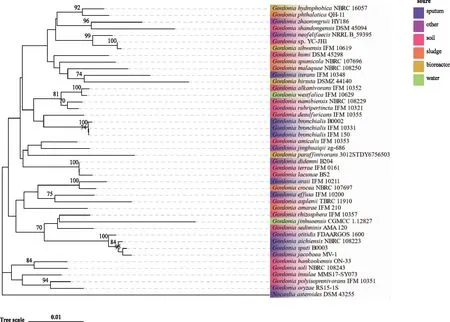
注:节点上数字表示自展值(仅显示自展值大于50%的值),标尺表示该长度的分支遗传变异度为0.01;蓝色、粉色、橙色、黄色、绿色及紫色分别表示来源于痰液、土壤、污泥、生物滤器、水及其他。
2.3 泛基因组特征分析
通过泛基因组特征曲线可得到戈登菌泛基因组、核心基因组与菌株数目之间的关系(图3),泛基因组特征方程y=3 686.37x0.55+343.9,R2=0.999 9,其中y为泛基因组大小,x为基因组数,即平均每增加1个新的基因组、就会有343.9个新基因会添加到泛基因组。与之相反的核心基因组的数量随菌株数量增加逐渐减少,直至趋于稳定,特征方程为y=3 560.92e-0.33x+1 036.39,当添加第12个基因组后缓慢稳定,可以推断出戈登菌的核心基因组基因数目会稳定在1 036个左右。
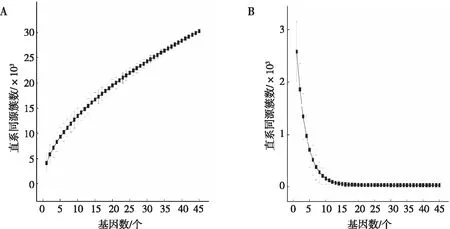
注:A和B分别为泛基因组特征曲线和核心基因组特征曲线。
2.4 戈登菌属基因功能分析
2.4.1COG功能注释分析 根据COG功能注释,戈登菌基因组几乎80%的基因被分为21个直系同源功能类别中,其中[S]未知功能的基因数量最多,其次是[C]能量的产生和转换、[E]氨基酸转运和代谢、[I]脂肪运输和代谢和[K]转录(图4),并且COG丰度与菌株来源之间差异无统计学意义(P>0.05)。

注:A表示RNA加工和修饰,B表示染色质结构和动力学,C表示能量产生和转换,D表示细胞周期控制、细胞分裂和染色体分离,E表示氨基酸转运和新陈代谢,F表示核酸转运和代谢;G表示碳运输和新陈代谢,H表示辅酶运输和代谢,I表示脂类转运和代谢,J表示翻译、核糖体结构和合成,K表示转录,L表示复制、重组及修复,M表示细胞壁膜核膜的合成,N表示细胞机动性,O表示翻译后修饰、蛋白翻转及分子伴侣,P表示无机离子转运和代谢,Q表示二级代谢生物加工、转运及分解代谢,S表示未知功能,T表示信号传导机制,U表示胞内的交换、分泌和膜泡输送,V表示防御机制。
2.4.2CAZymes在戈登菌中的分布 经过CAZymes数据库的注释,戈登菌属糖基转移酶(glycosyl transferases,GTs)含量最多,其次是糖苷水解酶(glycoside hydrolases,GHs),含少量的碳水化合物酯酶(carbohydrate esterases,CEs)和碳水化合物结合模块(carbohydrate-binding modules,CBMs;表2)。在不同来源的戈登菌属中,污泥来源菌株有(34.00±5.50)个基因簇与合成碳水化合物活性酶有关,含量较其他来源菌株少(P<0.05),来源于红树林泥泞土壤的G.rhizospheraNBRC 16068 CAZyme基因数量最多(55个),包含26个GT、19个GH、6个CE和4个CBM/GH,该菌株生存于养分低、重金属浓度及盐度较高地方,丰富的CAZymes为其生存提供了更大的可能。GH注释结果显示戈登菌基因组包含了参与不同糖代谢的基因,如β-葡萄糖苷酶、磷酸α-麦芽糖转移酶、分支酶和海藻糖磷酸化酶等。GT参与蛋白、核酸、各种杂环化合物和其它糖类的生物合成,戈登菌基因组中普遍存在1,6-半乳呋喃糖基转移酶、ADP依赖性α-麦芽糖-1-磷酸合酶、α-海藻糖-磷酸合酶等与能量储存有关的基因。
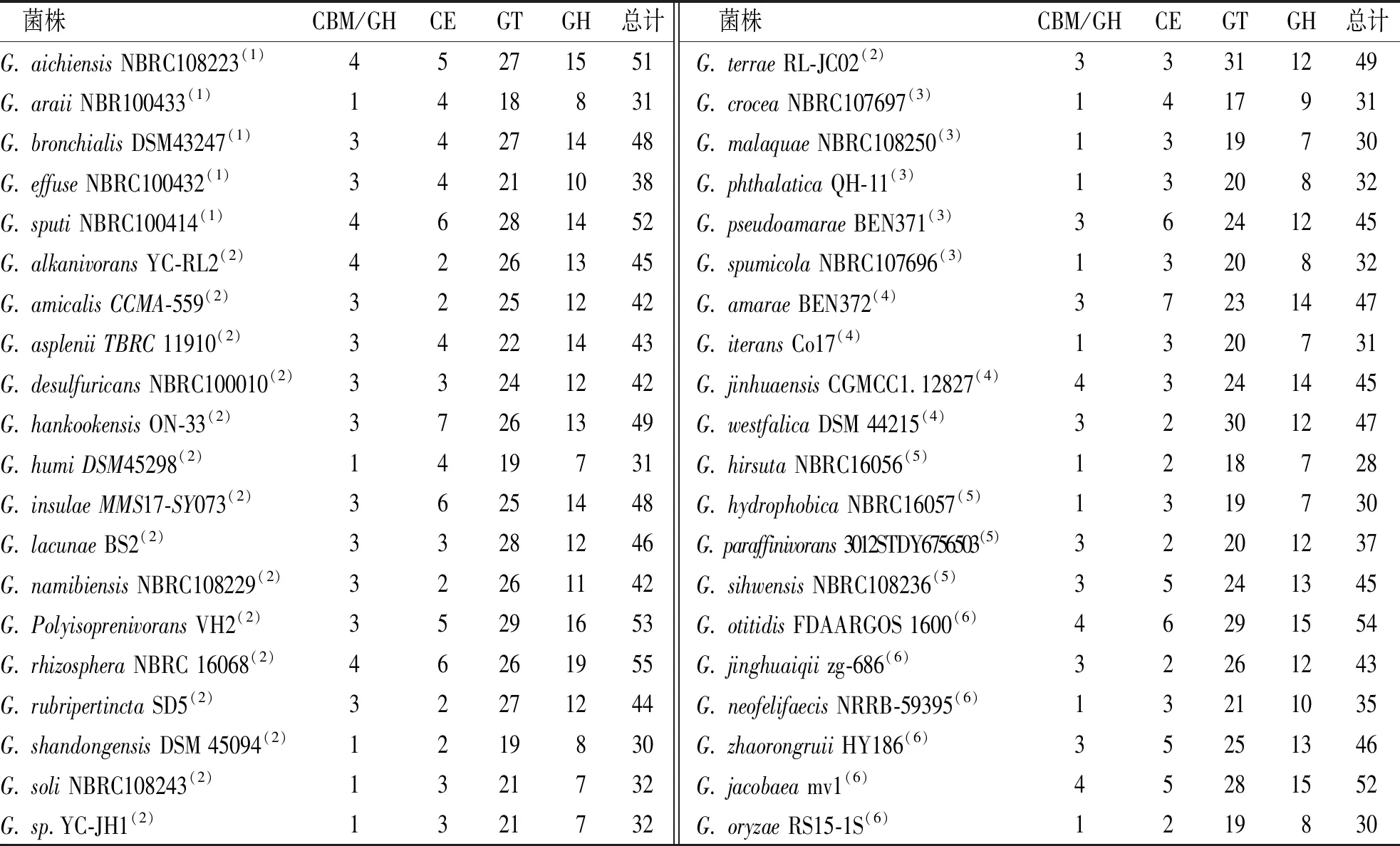
表2 戈登菌基因组中CAZyme种类及数目
2.4.3戈登菌次级代谢产物类型分布情况 通过AntiSMASH分析,被注释的戈登菌属含有PKS、NRPS、萜烯、四氢嘧啶(ectoine)以及RiPP次级代谢产物生物合成基因簇,其中PKS、NRPS和萜烯在该菌属所含基因簇中占比较高。其中,土壤来源的菌株含有的生物合成基因簇最长(565 044.18±145 911.85) bp,痰液和生物滤器来源的菌株含有的生物合成基因簇较短[(410 160.60±55 322.45) bp和(390 754.25±39 338.59) bp],3组之间差异有统计学意义(P<0.05);其他组别之间所含生物合成基因簇大小差异无统计学意义(P>0.05,表3);病原菌株基因簇的总长度在整个基因组中所占的比例低于土壤来源菌株,说明病原菌株合成次级代谢产物的能力比来源于土壤的菌株能力差(图5)。与MIBiG数据库比对以预测可能由戈登菌合成的已知或新的次级代谢产物,其中四氢嘧啶、匹马霉素(pimaricin)、类胡萝卜素(carotenoid)和SF2575的生物合成簇在戈登菌属中广泛存在,四氢嘧啶存在于40株戈登菌中,除G.otitidisFDAARGOS 1600有33.00%的基因与已知基因簇相似,其他的菌株至少有50%的基因与已知基因簇一致。相反,用于合成nocobactin NA、scabichelin、fuscachelin的生物合成基因簇只在少数菌株中存在超过50%的基因与已知基因簇一致,存在产生新的化合物的可能。
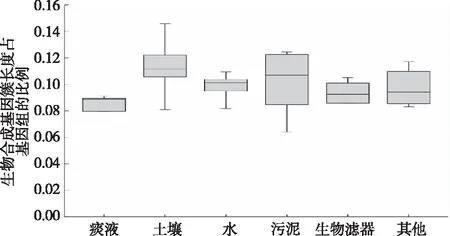
图5 不同来源戈登菌属基因组中生物合成基因簇长度占整个基因组的比例
3 讨论
通过对戈登菌属基因组的比较分析发现,戈登菌的基因组中最大基因组为G.aspleniiTBRC 11910和最小基因组为G.zhaorongruiiHY186;通过COG功能注释发现,G.aspleniiTBRC 11910拥有[I]脂质运输和代谢、[K]转录功能和[S]未知功能蛋白的数量是GordoniazhaorongruiiHY186的2倍,这表明为适应环境等因素的变化,不同的戈登菌的基因组也在进行演变,因此会出现不同菌株之间的差异性。同时,戈登菌属开放性的泛基因组,进一步说明戈登菌的基因组具有强大的可塑性和遗传多样性。
通过戈登菌属系统发育学分析可知40株已明确种的戈登菌之间ANI值<95%,DDH值<70%,均符合为同一属内的相关种的标准[15],结果表明40株已明确种的戈登菌分类准确。然而剩余的1株未进行准确分类的Gordoniasp.YC-JH1与G.sihwensisNBRC 108236之间的ANI值和DDH值均超过阈值范围,说明G.sp.YC-JH1为G.sihwensis的一个菌株,并且通过16S rRNA基因序列分析也证实了这一观点。
此前有学者对放线菌的CAZymes进行预测,如Ghimire等[16]通过比对红球菌属、分支杆菌属和棒状杆菌属部分菌株之间的CAZymes,发现它们含有淀粉、糖原和纤维素等化合物降解的CAZyme基因,并且红球菌属存在的角质酶可能是潜在的植物致病物质;Shin等[17]学者成功在大肠杆菌中克隆并表达G.terraeDSM 43249的β-葡萄糖苷酶,从而完成生物转化,提高人参皂苷中Rg3、Rg2和Rh1的转化率,将常量皂苷变为具有抗肿瘤、抗过敏、抗炎、缓解动脉硬化特性的稀有皂苷[18]。β-葡萄糖苷酶在生物代谢反应过程中起关键作用,能催化水解芳基或烃基与糖基原子团之间的糖苷键生成葡萄糖,在农业、食品行业、生物能源以及医药行业具有不可取代的作用。另外,本研究选取的戈登菌属菌株均注释到了海藻糖相关酶类,海藻糖具有稳定的化学性质,可参与细菌对高温、寒冷、高渗透和和脱水抗性的适应[19]。因此,戈登菌中丰富的CAZyme基因可在降解天然聚合物、生物技术等方面起着重要作用。
本研究还着重分析了戈登菌属产生次级代谢产物的能力,与其他放线菌相比戈登菌属显示出较强且多样的产次级代谢的能力[20]。所有的戈登菌菌株都含有能够生产萜烯的基因片段,萜烯和它的衍生物萜类化合物是一类主要的天然芳香化合物,具有多种生物活性,如抗氧化、抗微生物和抗炎特性等[21],有研究验证了链霉菌中存在合成戊内酯型萜烯的生物合成簇,对革兰阳性菌和革兰阴性菌表现出中等的抗菌活性[22]。有些类型的次级代谢产物只由1株或某几株戈登菌产生,如套索肽(lassopeptide)的相关基因只存在于G.croceaNBRC 107697,套索肽属于RiPP,其结构非常稳定,95 ℃~120 ℃高温条件下也不会被降解,具有抗菌、抗病毒、抗肿瘤和受体拮抗活性,基于其生物活性的多样性,套索肽可能在治疗胃肠疾病、结核病、阿尔茨海默病、心血管疾病、真菌感染和癌症等方面发挥很大的作用[23];呋喃的相关基因只存在于G.aspleniiTBRC 11910和G.spumicolaNBRC 107696中,呋喃是很多天然产物、药物和有机化合物的重要结构,可形成具有生物活性的物质,如能够抗氧化、降低心血管疾病风险的呋喃脂肪酸等[24]。通过与MIBiG数据库比对发现,戈登菌属中存在较多可产生PKS、NRPS及PKS-NRPS混合型的基因簇,可产生相关物质64种,PKS和NRPS参与大量生物活性化合物的生物合成,在临床上具有抗菌剂、抗寄生虫剂、抗肿瘤剂和免疫抑制剂等作用;Florez等[25]通过对链霉菌、诺卡菌等海洋放线菌的次级代谢产物研究发现,PKS和NRPS相关的生物活性化合物在宿主防御系统起重要作用。除此之外,部分基因簇存在于多数戈登菌菌株中,但与已知的生物合成簇相似性较低,如胡萝卜素、匹马菌素、康乐霉素及伯尔尼霉素A等,它们可能编码一些与己知次级代谢产物的结构或合成机制相似的天然产物,在临床和生物技术应用相关的活性化合物合成方面具有广阔前景。
综上所述,本研究通过比较不同来源的戈登菌属菌株之间在基因组大小、COG功能、碳水化合物酶类和次级代谢产物合成基因簇之间的差异性,发现该属菌株可产生非常具有生物和医学价值的酶类或次级代谢产物,并且还存在部分未明确的化合物,这为以后挖掘该属相关天然产物的研究奠定基础。



