郭云涛,张秀秀,苗向阳
(中国农业科学院北京畜牧兽医研究所,北京 100193)
牛miR-320a的靶基因预测及生物信息学分析
郭云涛,张秀秀,苗向阳*
(中国农业科学院北京畜牧兽医研究所,北京 100193)
旨在对bta-miR-320a的靶基因进行生物信息学预测和分析,探索其影响牛脂肪沉积的作用机制。本试验利用Promoter Scan、TargetScan、DAVID、Cytoscape等生物信息学软件和miRBase、Ensembl、NCBI、miRWalk等数据库对bta-miR-320a进行转录因子结合位点预测、保守性分析、靶基因预测、基因本体论富集分析和信号通路富集分析。结果表明,miR-320(a)在各物种间非常保守,bta-miR-320a启动子区域有SP1等多个转录因子结合位点,获得的84个靶基因主要参与负调控细胞分化、细胞周期、负调控生长等多个生物学过程,涉及p53、细胞周期和MAPK等信号转导通路。由此推测,bta-miR-320a受到SP1等多种转录因子调控,它可能通过对MAPK、细胞周期和p53信号通路中靶基因TP53、MAPK1等的抑制作用调控牛脂肪细胞分化,进而影响了牛的脂肪沉积。
牛;bta-miR-320a;靶基因;生物信息学
microRNA(miRNA,微RNA)是真核生物中广泛存在的一类内源性非编码RNA,长约21~22 nt,它可以通过特异性碱基互补的方式与靶基因mRNA的3′-UTR结合,抑制靶mRNA翻译或诱导其降解,从而在转录后水平调控基因的表达[1-3]。自从第一个miRNAlin-4在线虫中被发现以来[4],越来越多的miRNA被鉴定出来,目前最权威的miRbase(http://www.mirbase.org/)数据库收录的miRNA条目已达28 645条,并且数目在逐年增长,库中收录了牛的808条miRNAs前体,793条成熟体。作为近年来研究的热门,miRNA参与了各种生命过程的调控,包括细胞的增殖、分化、凋亡和新陈代谢等,在生物体生长、发育和疾病发生等过程中扮演着重要角色[5]。在肉牛生产中,胴体中的皮下和肌内脂肪含量是肉品质的一个重要指标,肌内脂肪不足和皮下脂肪过多是对牛肉品质的最大挑战,研究肉牛脂肪沉积过程的分子机制对于生产优质牛肉至关重要。脂肪沉积伴随着脂肪细胞体积增大和数目增加,脂肪细胞数目增加与脂肪细胞分化密切相关。已有研究证实,miRNA参与了脂肪细胞分化调控[6],故研究脂肪细胞分化中的miRNA对于肉牛脂肪沉积研究有重要的理论和实际意义。
miR-320a属于miR-320家族,家族内有许多成员,如人的miR-320有5个高度同源的种类:hsa-miR-320a/b/c/d/e,牛的miR-320(Bta-miR-320)有两种高度同源的类型:bta-miR-320a(MIMAT0003534)和bta-miR-320b(MIMAT0011991)。目前,关于miR-320家族的研究多集中在癌症方面,miR-320可以抑制癌细胞增殖,在结肠直肠癌、肝癌、胃癌、乳腺癌、前列腺癌和肝癌骨肉瘤等多种恶性肿瘤侵袭和迁移中,miR-320都出现了下调表达。H.Y.Ling等[7]研究发现,miR-320在3T3-L1脂肪细胞中通过调控胰岛素-PI3-K信号通路调控了胰岛素抵抗,miR-320可能参与了脂肪组织中的生物过程调控。D.Hamam等[8]在研究人骨骼肌多功能干细胞(Human mesenchymal stem cells,hMSCs)向脂肪细胞分化时发现,miR-320家族发生了显着上调,进一步试验证实hsa-miR-320c通过靶向抑制RUNT相关转录因子2(Runt related transcription factor 2,RUNX2)影响了脂肪细胞分化。本实验室前期对不同脂肪沉积能力的两个品种牛的皮下脂肪组织小RNA进行了高通量测序,在分析结果时发现,bta-miR-320a在脂肪沉积能力强的牛种中发生了显着的下调表达(|log2FC|>1,P<0.05,FDR<0.05),且在两个牛种中表达丰度都非常高(RPKM>500),推测bta-miR-320a可能在脂肪沉积中发挥了重要作用。目前,最权威的miRBase数据库中对bta-miR-320a的功能没有注释,miR-320a对牛脂肪细胞分化的影响机制尚不清楚,因此,本试验旨在利用生物信息学的方法对bta-miR-320a进行转录因子结合位点(Transcription factor binding site,TFBS)预测、同源性和进化分析、靶基因预测、靶基因的基因本体论(Gene ontology,GO)和KEGG信号通路(KEGG Pathway)富集分析,以了解bta-miR-320a可能的转录因子调控位点、进化关系及其参与调控的生物过程和信号通路,进而对其在牛脂肪细胞分化中的功能进行探索,为bta-miR-320a在牛脂肪沉积中的研究提供理论指导。
1 材料与方法
1.1 转录因子结合位点(TFBS)预测
在Ensemble数据库中检索出bta-miR-320a的两条前体,取其上游10 kb的序列,利用Promoter Scan Version 1.7(http://www-bimas.cit.nih.gov/molbio/proscan/)、JASPAR CORE Vertebrate(http://jaspardev.genereg.net/)和PATCH public 1.0(http://www.gene-regulation.com/cgi-bin/pub/programs/patch/bin/patch.cgi)3个在线预测软件做TFBS预测。
1.2 miR-320(a)序列保守性分析
从miRBase中检索出牛(Bostaurus,bta)、大猩猩(Gorillagorilla,ggo)、人(Homosapiens,hsa)、猕猴(Macacamulatta,mml)、黑猩猩(Pantroglodytes,ptr)、婆罗州猩猩(Pongopygmaeus,ppy)、灰仓鼠(Cricetulusgriseus,cgr)、大鼠(Rattusnorvegicus,rno)、小鼠(Musmusculus,mmu)、山羊(Caprahircus,chi)、狗(Canisfamiliaris,cfa)的miR-320(a)序列,分析其保守性。
1.3 Bta-miR-320a的靶基因预测和后续分析靶基因的获得
选择TargetScan 6.2(http://www.targetscan.org/vert_61/)、PicTar(http://pictar.mdc-berlin.de/)和miRanda(阈值mirSVR score<-1,http://www.microrna.org/microrna/getDownloads.do)3种计算方法预测bta-miR-320a的靶基因,取3者预测结果的交集,然后结合miRwalk(http://www.umm.uni-heidelberg.de/apps/zmf/mirwalk/mirnatargetpub.html)上miR-320a的已验证靶标以及文献中已报道miR-320a靶基因,共同组成bta-miR-320a的靶基因集合。
1.4 靶基因的GO富集分析
将bta-miR-320a的预测靶基因集合用Gene Ontology进行富集分类,选择所有蛋白编码基因作为背景基因,用DAVID[9-10]工具(http://david.abcc.ncifcrf.gov/)对bta-miR-320a预测的靶基因集合进行基于生物学过程(Biological process,BP)、细胞组成(Cellular component,CC)和分子功能(Molecular function,MF)3个层面的Gene Ontology注释层次分类及富集分析,通过Fsiher Exact Test计算P值,以P<0.05为显着性阈值得到基因集合相对于背景具有统计意义的GO条目,并对P值进行校正,标准是Benjamini<0.05。
1.5 靶基因的KEGG Pathway富集分析
用DAVID工具对bta-miR-320a预测的靶基因集合进行基于KEGG的生物通路富集分析,通过Fsiher Exact Test计算P值,以P<0.05为显着性阈值得到基因集合相对于背景具有统计意义的信号转导通路,并对P进行校正,标准是Benjamini<0.05。
1.6 TF-miRNA-靶基因作用网络
根据转录因子、靶基因和通路富集分析结果,使用Cytoscape3.2.0软件对预测的结果进行作用关系网络图绘制。
2 结 果
2.1 TFBS预测结果
使用3种在线软件分别对两条bta-miR-320a前体(表1)的上游10 kb区域TFBS进行预测,发现它们启动子区域有致癌基因JUN(Jun proto-oncogene,JUN或AP-1)、增强子激活结合蛋白2(Activating enhancer binding protein 2 alpha,AP-2)、转录因子SP1(Sp1 transcription factor,SP1)、八聚体结合蛋白(Major octamer-binding protein,OCT)、过氧化物酶体增殖物活化受体γ(Peroxisome proliferator-activated receptor gamma,PPARG)、成肌分化因子1(Myogenic differentiation 1,MYOD1)、cAMP反应元件结合蛋白(cAMP responsive element binding protein,CREB)等多个转录因子结合位点,其中SP1、AP-2、PPARG、MYOD1和CREB参与了脂肪细胞增殖分化和脂代谢调控。
表1 bta-miR-320a的两条前体信息
Table 1 Information of 2 bta-miR-320a precursors

名称NameEnsembl登录号EnsemblaccessionNo.染色体Chromosome位置/bpLocation长度/bpLengthbta⁃mir⁃320a⁃1ENSBTAG00000029761870060384~7006046582bta⁃mir⁃320a⁃2ENSBTAG000000297602015213924~1521400582
2.2 保守性分析
从牛、大猩猩、人等11个物种的miR-320(a)序列(表2)可以看出,miR-320a在进化中非常保守,成熟体序列完全一致。
表2 不同物种的miR-320a序列
Table 2 Sequences of miR-320a from different species

名称Name登录号AccessionNo.序列(5′⁃3′)Sequencebta⁃miR⁃320aMIMAT0003534AAAAGCUGGGUUGAGAGGGCGAggo⁃miR⁃320aMIMAT0024080AAAAGCUGGGUUGAGAGGGCGAhsa⁃miR⁃320aMIMAT0000510AAAAGCUGGGUUGAGAGGGCGAmml⁃miR⁃320aMIMAT0006263AAAAGCUGGGUUGAGAGGGCGAptr⁃miR⁃320aMIMAT0008093AAAAGCUGGGUUGAGAGGGCGAppy⁃miR⁃320aMIMAT0015821AAAAGCUGGGUUGAGAGGGCGAcgr⁃miR⁃320aMIMAT0023906AAAAGCUGGGUUGAGAGGGCGArno⁃miR⁃320⁃3pMIMAT0000903AAAAGCUGGGUUGAGAGGGCGAmmu⁃miR⁃320⁃3pMIMAT0000666AAAAGCUGGGUUGAGAGGGCGAchi⁃miR⁃320⁃3pMIMAT0036132AAAAGCUGGGUUGAGAGGGCGAcfa⁃miR⁃320MIMAT0006658AAAAGCUGGGUUGAGAGGGCGA
2.3 靶基因预测
选择TargetScan、PicTar和miRanda 3种计算方法预测bta-miR-320a的靶基因,取3者的交集共得40个基因,合并已经证实为miR-320a的 30个来自miRwalk数据库的靶基因和已发表研究证实的19个靶基因,除去5个重复,获得共计84个靶基因作为后续GO和KEGG pathway分析的靶基因集合(图1,表3)。
2.4 GO富集分析
通过对靶基因集合进行GO富集分析(表4~表6)可知bta-miR-320a的靶基因集富集在负调控细胞分化、细胞周期、负调控生长、细胞发育、神经发育和细胞凋亡等多个生物学过程中,作用的位置主要在细胞核内和细胞器内腔中,发挥转录调控活动和酶结合的分子作用。其中生物学过程中的细胞周期、细胞增殖和分化条目下的基因可能会对脂肪细胞的增殖、分化产生影响。
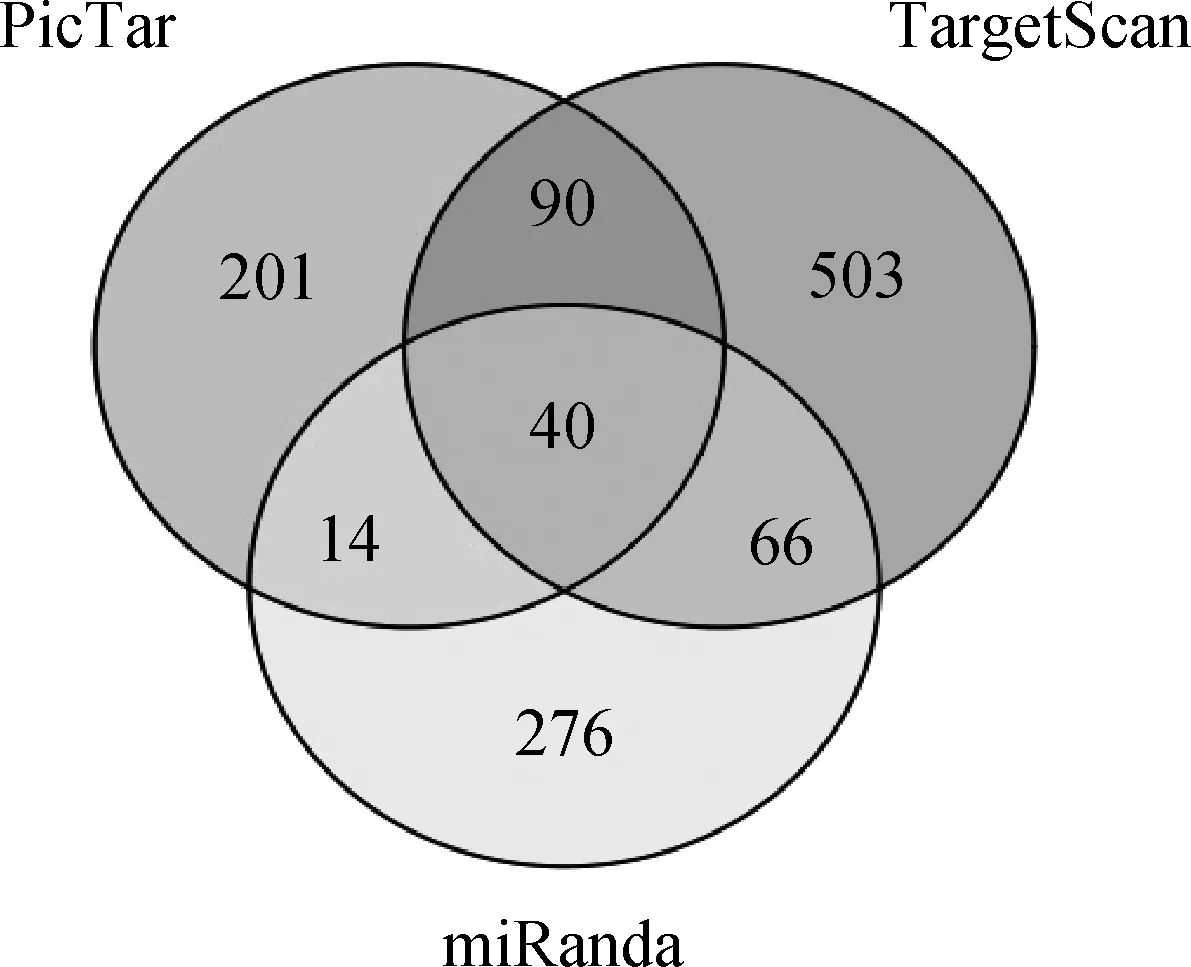
图1 3种靶基因预测软件对bta-miR-320a的靶基因预测情况Fig.1 The prediction results of bta-miR-320a target genes using 3 kinds of bioinformatics tools
表3 bta-miR-320a的靶基因统计
Table 3 Statistics of target genes of bta-miR-320a

来源Source数目Number靶基因列表ListoftargetgenesPicTar(P)345TargetScan(T)669miRanda(M)396Research(R)19miRWalk(W)30P∩T∩M40(P∩T∩M)∪R∪W84ARF1,ARL8B,BANP,C14orf147,CDH20,CDK13,COPS2,DAZAP1,DBN1,DHX15,DNER,ELL2,FAM49B,FLRT3,GABPB1,GPBP1,KCNS3,KLHL36,LMO3,LPPR1,MLLT3,MN1,MSI2,PBX3,PCDHA1,PCDHA4,PLXNC1,PPP2R2C,RAI2,RAP1A,RASA1,SLITRK3,SPOPL,STAG2,TMEM108,TPM3,XPO1,YWHAZ,ZIC3,ZNF281,IL1A,BBC3,BRCA1,TAC1,MCL1,COX8A,TFRC,TP53,AQP1,TP73,AQP4,EZH2,ITCH,ETV6,EIF2C1,RCOR1,DICER1,LIN9,BCL2,BIRC5,POLR3D,REST,RUNX1,HSPB6,E2F1,HNF4A,PIK3R1,GNAI1,TFRC,RAC1,MAPK1,ITGB3,HSP20,FAS,ARPP19,RUNX2,NRP1,ETS2,CDKN1C,CDKN1A,MIB1,PAX6,YWHAH,ZWILCH,HSC70
P∩T∩M表示3种不同靶基因预测软件PicTar、TargetScan和miRanda预测结果的交集,(P∩T∩M)∪R∪W表示将3个预测软件获得的交集与已发表文献和miRWalk已验证靶标取并集作为后续分析的靶基因集合
P∩T∩M means the results of prediction using 3 different kinds of target gene prediction sofwares,(P∩T∩M)∪R∪W means the group of target genes used for the following analysis which come from the intersection of 3 target gene prediction softwares,target genes in papers published and those verified in miRWalk database
2.5 KEGG Pathway富集分析
为了获取更多关于miR-320a在功能方面的信息,本试验在GO 注释分类的基础上,利用已有生物通路数据对基因集合中的84个基因进行生物通路富集分析。结果显示,在经典通路数据库KEGG中bta-miR-320a预测靶基因集合显着富集于神经营养因子、细胞周期、p53信号通路、细胞凋亡、蛋白激酶(MAPK)5个信号转导通路和10个疾病相关信号通路(图2),其中与生长发育关系密切的MAPK信号通路也发生了显着性富集(P<0.05,Benjamini=0.062),可能具有假阳性。
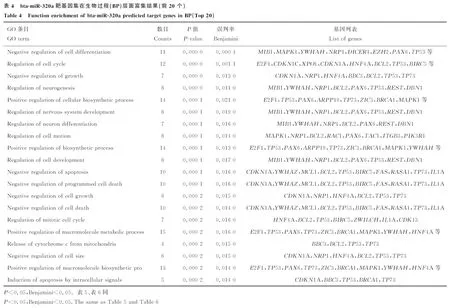
表5 bta-miR-320a靶基因集在细胞组分(CC)层面富集结果
Table 5 Function enrichment of bta-miR-320a predicted target genes in CC

GO条目GOterm数目CountsP值P⁃value误判率Benjamini基因列表ListofgenesNuclearlumen190.00010.0110E2F1,COPS2,XPO1,MCL1,LIN9,EZH2,TP53,BIRC5,BRCA1,POLR3D等Nucleoplasm140.00020.0140E2F1,XPO1,MCL1,LIN9,EZH2,TP53,BRCA1,POLR3D,ELL2,MAPK1等Intracellularorganellelumen190.00080.0490E2F1,COPS2,XPO1,MCL1,LIN9,EZH2,TP53,BIRC5,BRCA1,POLR3D等Organellelumen190.00100.0480E2F1,COPS2,XPO1,MCL1,LIN9,EZH2,TP53,BIRC5,BRCA1,POLR3D等Membrane⁃enclosedlumen190.00130.0480E2F1,COPS2,XPO1,MCL1,LIN9,EZH2,TP53,BIRC5,BRCA1,POLR3D等
表6 bta-miR-320a靶基因集在分子功能(MF)层面富集结果
Table 6 Function enrichment of bta-miR-320a predicted target genes in MF

GO条目GOterm数目CountsP值P⁃value误判率Benjamini基因列表ListofgenesTranscriptionregulatoractivity200.00010.0230E2F1,ZNF281,COPS2,EZH2,TP53,PAX6,REST,BRCA1,TP73,ELL2,CDKN1C等Enzymebinding110.00030.0280MAPK1,YWHAH,BCL2,RAC1,TP53,RAP1A,BIRC5,FAS,RASA1,BRCA1,PIK3R1

Y轴为-log2P-value,X轴为富集到的信号通路条目,Y越大,富集越显着(P<0.05,Benjamini<0.05)The Y-axis means -log2P-value,X-axis means the pathway terms enriched(P<0.05,Benjamini<0.05)图2 bta-miR-320a靶基因的KEGG 通路分析结果Fig.2 KEGG enrichment result of bta-miR-320a predicted target genes
2.6 转录因子-miRNA-靶基因作用网络
转录因子SP1、AP-2、PPARG、MYOD1和CREB调控了bta-miR-320a基因的转录,bta-miR-320a通过TP53、MAPK1等靶基因参与了MAPK、p53和细胞周期信号通路(图3)。

三角代表转录因子,圆形代表bta-miR-320a,方块表示靶基因The triangles represent transcription factors,the center is bta-miR-320a,the squares means target genes图3 转录因子-miRNA-靶基因作用关系图Fig.3 TF-miRNA-target gene network
3 讨 论
在生物体内,1个miRNA可能作用于多个靶基因,也可能是多个miRNA调控1个靶基因,这种作用构成一个调控网络,在某些信号的刺激下,从整体上调控有机体的生命活动。 因此,miRNA的作用是通过其复杂的靶基因协同调节实现的,仅仅靠试验手段来研究miRNA已变得相当困难。靶基因数量和种类较多,生物信息学在miRNA的研究中扮演的角色越来越重要。生物信息学方法可以对海量和复杂信息进行分析和处理,为下一步试验提供指导,包括编码miRNA基因上游的TFBS预测、miRNA的靶基因预测及靶基因的GO分析、信号通路分析等。miRanda、TargetScan和PicTar是目前较流行的3种miRNA靶基因预测计算方法。miRanda是最早的一种预测算法,有较好的检出率,预测的靶基因数量较多,但假阳性率也较高;TargetScan和PicTar作为第二代预测算法,其预测靶基因的数量相对减少,但同时也增加了预测的精确度,降低了假阳性率。虽然这3种不同的软件预测的结果不一样,但同时采用这3种预测算法进行预测,然后取其交集部分,就能提高预测结果的可靠性,减少假阳性率。本试验在利用miRanda对bta-miR-320a的靶基因进行预测时将阈值设定为mirSVR score<-1,减少了假阳性率,然后采用上述3种靶基因预测软件预测结果的交集部分,使靶基因的预测更加准确。在进行后续功能分析之前,综合了miRWalk数据库(2011)和近几年来关于miR-320家族的研究中所确定的靶标作为后续分析的靶基因集合,靶基因预测是进行miRNA功能研究的基础,采用上述策略鉴定出的靶基因保证了后续功能分析的全面和可靠性。
关于miR-320家族的功能研究多集中在癌症方面。C.Shang等[11]研究发现miR-320a通过靶向抑制整合素β3(Integrin,beta 3,ITGB3)介导了膀胱癌的侵袭;miR-320a/c/d可靶向抑制G蛋白α抑制性多肽1(G protein alpha inhibiting activity polypeptide 1,GNAI1)表达,进而抑制肝癌细胞的迁移和迁徙[12];miR-320a亦可通过靶向抑制Ras相关的C3肉毒杆菌毒素底物1(Ras-related C3 botulinum toxin substrate 1,RAC1)影响结肠直肠癌的发展[13];在重症肌无力病人中miR-320a下调表达,通过靶作用于丝裂原活化蛋白激酶(Mitogen-activated protein kinase 1,MAPK1)调节炎性细胞因子产生[14];在血红素介导的红细胞分化研究[15]中,miR-320a通过抑制其靶基因BTG3相关核蛋白(BTG3 associated nuclear protein,BANP)发挥了调控作用。由这些研究可以得知miR-320a是癌细胞和红细胞分化中的抑制因子,推测它在牛脂肪细胞分化中可能也起到了抑制作用。
Bta-miR-320a靶基因集的GO注释富集在负调控细胞分化、细胞周期等多个生物学过程条目中,由此推测,bta-miR-320a对牛脂肪细胞分化产生了负调控作用,从而抑制脂肪沉积。脂肪沉积能力强的牛种中bta-miR-320a下调表达,减弱了这种抑制作用,这与人们的预期相符。bta-miR-320a对牛脂肪细胞分化的负调控作用是通过对其靶基因如抑癌基因53(Tumor suppressor53,TP53或p53)、MAPK1等的抑制作用实现的。TP53编码了一种具有转录激活、DNA结合功能的肿瘤抑制蛋白,介导了细胞周期停滞、细胞凋亡、衰老、DNA损伤修复等生物过程,参与了MAPK、细胞周期和p53等多个信号通路的信号转导。MAPK1属于蛋白激酶家族,是MAPK信号通路中的重要成员,参与了细胞的增殖分化、转录调控和发育等广泛的生物学过程。牛脂肪细胞由前体脂肪细胞增殖分化而来,前体脂肪细胞经历有丝分裂克隆增殖、生长停滞和终末分化形成成熟的脂肪细胞,在这一过程中pRB-E2F、MAPK、SMAD/TGFβ、Wnt等信号通路发挥了重要的调控作用。本试验中,bta-miR-320a的靶基因富集在细胞周期、p53信号通路、MAPK等信号转导通路和10个疾病相关信号通路,其中细胞周期、p53信号通路在真核生物有丝分裂细胞周期调控中发挥重要功能,MAPK级联信号通路在真核生物中是一个高度保守的通路,它调控了包括细胞增殖、分化和迁移等各种功能,MAPK作为上游的通路也会对细胞周期产生作用。靶基因之一的TP53参与了上述3条通路,在信号传导中占据着枢纽位置,它在G1、S和 G2时相转换中发挥调控作用。Q.Wang等[16]研究发现,在小鼠3T3L1前体脂肪细胞分化的有丝分裂克隆增殖阶段,miR-17-92簇上调表达,激素诱导的miR-17-92过表达加速了3T3L1前体脂肪细胞分化,增加了甘油三脂积聚。针对这些现象,他们提出了一个模型:在激素诱导后3T3L1前体脂肪细胞中,miR-17-92表达量升高,抑制了其靶基因成视网膜细胞瘤样2(Retinoblastoma-like 2,RB2/P130)表达,导致没有足够的RB2/P130与转录因子E2F(E2F transcription factor,E2F)形成二聚体来抑制E2F,活化状态的E2F4和E2F5增加,激活了成视网膜瘤蛋白pRB-E2F信号通路,启动细胞进入下一个周期。因此,miR-17-92簇在克隆增殖阶段的上调表达促进了前体脂肪细胞分化。L.Chen等[17]用芯片技术研究大鼠脂肪组织来源基质细胞(Adipose tissue-derived stromal cells,ADSCs)向成熟脂肪细胞分化时miRNA的表达谱,发现miR-363发生了最显着的下调,ADSCs中过表达miR-363会引起克隆增殖和终末分化抑制,进而确定miR-363是脂肪生成中的一个负调控者,可以靶向抑制E2F3的转录后翻译水平,通过激活pRB-E2F抑制周期蛋白E(Cyclin E,CYCE)的表达,从而抑制细胞从G1向S期的转变也就抑制了前体脂肪细胞克隆增殖,从而抑制了细胞终末分化。C.Esau和L.Chen等[6,18]研究发现,miR-143能通过对其在MAPK信号通路中靶基因丝裂原活化蛋白激酶5(Mitogen-activated protein kinase 5,MAPK5)的抑制作用调控脂肪细胞的分化。miR-17-92、miR-363和miR-143等通过pRB-E2F和MAPK信号通路调节了脂肪细胞分化,而pRB-E2F信号通路中的RB、E2F也是细胞周期通路的作用元件,因此推测,在牛脂肪细胞分化中,miR-320a通过对细胞周期和p53信号通路中相关靶基因如TP53的靶向抑制,影响了这些通路信号的传递,进而导致前体脂肪细胞有丝分裂克隆扩增中G1-S-G2相位转变的时序发生变化,从而影响了前体脂肪细胞分化。同时,它也对MAPK级联信号通路中相关的分子组件进行靶向抑制,对MAPK下游信号通路产生影响,调控脂肪细胞分化。bta-miR-320a基因启动子区域具有SP1、PPARG和MYOD1等转录因子结合位点,这些转录因子与脂肪细胞增殖分化、脂代谢关系密切,可能会对miR-320a调控牛脂肪细胞分化产生影响。
本试验筛选出的靶基因还需要进一步的试验验证。另外,在进行靶基因预测和功能分析时使用的条件比较严格,可以有效减少假阳性率,提高预测准确度,但同时会有一些漏筛率,还需找到更加准确全面的方法对bta-miR-320a进行后续分析。总之,本试验对miR-320a在牛脂肪沉积中的功能进行了探索,挖掘出了SP1等多种转录因子、TP53和MAPK1等关键靶基因以及p53等重要信号通路,为后续的试验验证提供参考。
4 结 论
Bta-miR-320a受到SP1等多种转录因子调控,它可能通过对MAPK、细胞周期和p53信号通路中靶基因TP53、MAPK1等的抑制调控了牛脂肪细胞分化,进而影响了牛的脂肪沉积。
[1] HE L,HANNON G J.MicroRNAs:small RNAs with a big role in gene regulation[J].NatRevGenet,2004,5(7):522-531.
[2] LEE Y,AHN C,HAN J,et al.The nuclear RNase III Drosha initiates microRNA processing[J].Nature,2003,425(6956):415-419.
[3] PASQUINELLI A E,REINHART B J,SLACK F,et al.Conservation of the sequence and temporal expression of let-7 heterochronic regulatory RNA[J].Nature,2000,408(6808):86-89.
[4] LEE R C,FEINBAUM R L,AMBROS V.The C.elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14[J].Cell,1993,75(5):843-854.
[5] AMBROS V.MicroRNA pathways in flies and worms:growth,death,fat,stress,and timing[J].Cell,2003,113(6):673-676.
[6] ESAU C,KANG X L,PERALTA E,et al.MicroRNA-143 regulates adipocyte differentiation[J].JBiolChem,2004,279(50):52361-52365.
[7] LING H Y,OU H S,FENG S D,et al.Changes in microRNA(miR) profile and effects of miR-320 in insulin-resistant 3T3-L1 adipocytes[J].ClinExpPharmacolPhysiol,2009,36(9):e32-39.
[8] HAMAM D,ALI D,VISHNUBALAJI R,et al.microRNA-320/RUNX2 axis regulates adipocytic differentiation of human mesenchymal(skeletal) stem cells[J].CellDeathDis,2014,5(10):e1499.
[9] HUANG DA W,SHERMAN B T,LEMPICKI R A.Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources[J].NatProtoc,2009,4(1):44-57.
[10] DENNIS G JR,SHERMAN B T,HOSACK D A,et al.DAVID:Database for annotation,visualization,and integrated discovery[J].GenomeBiol,2003,4(5):P3.
[11] SHANG C,ZHANG H,GUO Y,et al.MiR-320a down-regulation mediates bladder carcinoma invasion by targeting ITGB3[J].MolBiolRep,2014,41(4):2521-2527.
[12] YAO J,LIANG L H,ZHANG Y,et al.GNAI1 suppresses tumor cell migration and invasion and is post-transcriptionally regulated by miR-320a/c/d in hepatocellular carcinoma[J].CancerBiolMed,2012,9(4):234-241.
[13] ZHAO H,DONG T,ZHOU H,et al.miR-320a suppresses colorectal cancer progression by targeting Rac1[J].Carcinogenesis,2014,35(4):886-895.
[14] CHENG Z,QIU S,JIANG L,et al.MiR-320a is downregulated in patients with myasthenia gravis and modulates inflammatory cytokines production by targeting mitogen-activated protein kinase 1[J].JClinImmunol,2013,33(3):567-576.
[15] MITTAL S P,MATHAI J,KULKARNI A P,et al.miR-320a regulates erythroid differentiation through MAR binding protein SMAR1[J].IntJBiochemCellBiol,2013,45(11):2519-2529.
[16] WANG Q,LI Y C,WANG J,et al.miR-17-92 cluster accelerates adipocyte differentiation by negatively regulating tumor-suppressor Rb2/p130[J].ProcNatlAcadSciUSA,2008,105(8):2889-2894.
[17] CHEN L,CUI J,HOU J,et al.A novel negative regulator of adipogenesis:microRNA-363[J].StemCells,2014,32(2):510-520.
[18] CHEN L,HOU J,YE L F,et al.MicroRNA-143 regulates adipogenesis by modulating the MAP2K5-ERK5 signaling[J].SciRep,2014(4):3819.
(编辑 郭云雁)
Target Gene Prediction and Bioinformatics Analysis of bta-miR-320a
GUO Yun-tao,ZHANG Xiu-xiu,MIAO Xiang-yang*
(InstituteofAnimalScience,ChineseAcademyofAgriculturalSciences,Beijing100193,China)
The prediction and analysis of bta-miR-320a target genes by bioinformatics were performed to explore its effect on the fat deposition in bovine.In this study,bioinformatics tools such as Promoter Scan,TargetScan,DAVID,Cytoscape and databases for example miRBase,Ensemble,NCBI,miRWalk,et al were used to perform the prediction of the transcription factor binding site and the analysis of its target genes,involved Gene ontology and KEGG pathway.As a result,miR-320(a) was very conservative among various species.There were many different transcription factor binding sites in the bta-miR-320a promoter area.Eighty-four target genes were obtained in total which involved in multiple biological processes such as negative regulation of cell differentiation,regulation of cell cycle and negative regulation of growth and participated in p53,cell cycle and MAPK signal pathways.We assumed that bta-miR-320a is regulated by different transcription factors,and it can also regulate the adipocyte differentiation and fat deposition in bovine by post transcriptional inhibiting of its target genes involved in p53,cell cycle and MAPK signal pathways.
bovine;bta-miR-320a;target gene;bioinformatics
10.11843/j.issn.0366-6964.2015.11.006
2014-12-24
中央级公益性科研院所基本科研业务费专项资金项目(2013ywf-yb-5;2013ywf-zd-2);中国农业科学院农业科技创新项目(ASTIP-IAS05);转基因生物新品种培育科技重大专项(2009ZX08008-004B;2008ZX08008-003);国家“863”计划项目(2008AA10Z140);国家自然科学基金项目(30571339);中国农业科学院创新基金项目(2004-院-1)
郭云涛(1988-),男,河南南阳人,硕士,主要从事转基因与细胞工程研究,E-mail:yuntao008@126.com
*通信作者:苗向阳,研究员,博士,博士生导师,主要从事基因工程与功能基因组学及转基因动物研究,E-mail:mxy32@sohu.com
S823;S813.3
A
0366-6964(2015)11-1952-09



