邓 娟,张红平,巴 贵,次仁德吉,宋天增*,李 利*
(1.四川农业大学动物遗传育种研究所,成都 611130; 2.西藏农牧科学院畜牧兽医研究所,拉萨 850009)
藏山羊作为我国独特的种质资源,其体型矮小,被毛富有光泽,颜色较杂,除白色和黑色较多外,还有青色、褐色等,在青藏高原的农业、经济和文化,甚至宗教等方面都扮演了重要角色[1],品种主要分布在西藏自治区全境、四川省甘孜和阿坝2个自治州、青海省玉树和果洛藏族自治州、甘肃甘南藏族自治州及新疆部分地区[2]。高原环境的长期阻隔以及地理分布的差异,在长期的自然选择和人工选择下,藏山羊群体间缺乏足够的基因交流,逐步形成了不同的生态类群。王杰等[3]、王永等[1]先后采用SSR与ISSR分子标记对藏山羊的遗传多态性进行了分析,结果显示,藏山羊多样性较丰富,但群体间存在差异。在此次采样过程中,笔者发现当地牧民大多以单户养殖为主,一些产区(如城关镇、林周县、萨嘎县等)的藏山羊群体数量已经极其稀少。加上近年来,在经济利益的驱动下,各地也持续引入外来山羊品种,如辽宁绒山羊[4]、南江黄羊[5-6]、内蒙古白绒山羊[7]等对本地山羊品种进行遗传改良,提高其综合生产性能,导致藏山羊这一遗传资源正在面临危机。
线粒体基因组(Mitochondrial DNA,mtDNA)上的细胞色素b(Cytb)是蛋白编码基因之一,其进化速度适中,序列片段包含种内到种间的进化遗传信息,已被用来研究多个物种的系统发育关系和遗传多样性[8-9]。然而,利用Cytb基因对藏山羊的遗传多样性和分子进化方面的研究还未见报道。本研究对西藏地区8个群体共157个藏山羊个体的Cytb基因全序列进行扩增和测序,在此基础上分析群体遗传多样性,揭示不同地域藏山羊群体间的遗传结构及系统发育关系,为藏山羊品种资源的保护与利用提供理论参考。
1 材料与方法
1.1 样本采集
根据藏山羊的主产地区,从西藏8个地区采集藏山羊血液样本共计157个,每个群体内的个体之间无亲缘关系。采用颈静脉采血,3.9% ACD抗凝,-20 ℃冰箱储存备用。样本详细信息见表1。
表1藏山羊样品采集信息
Table1SampleinformationofTibetangoats
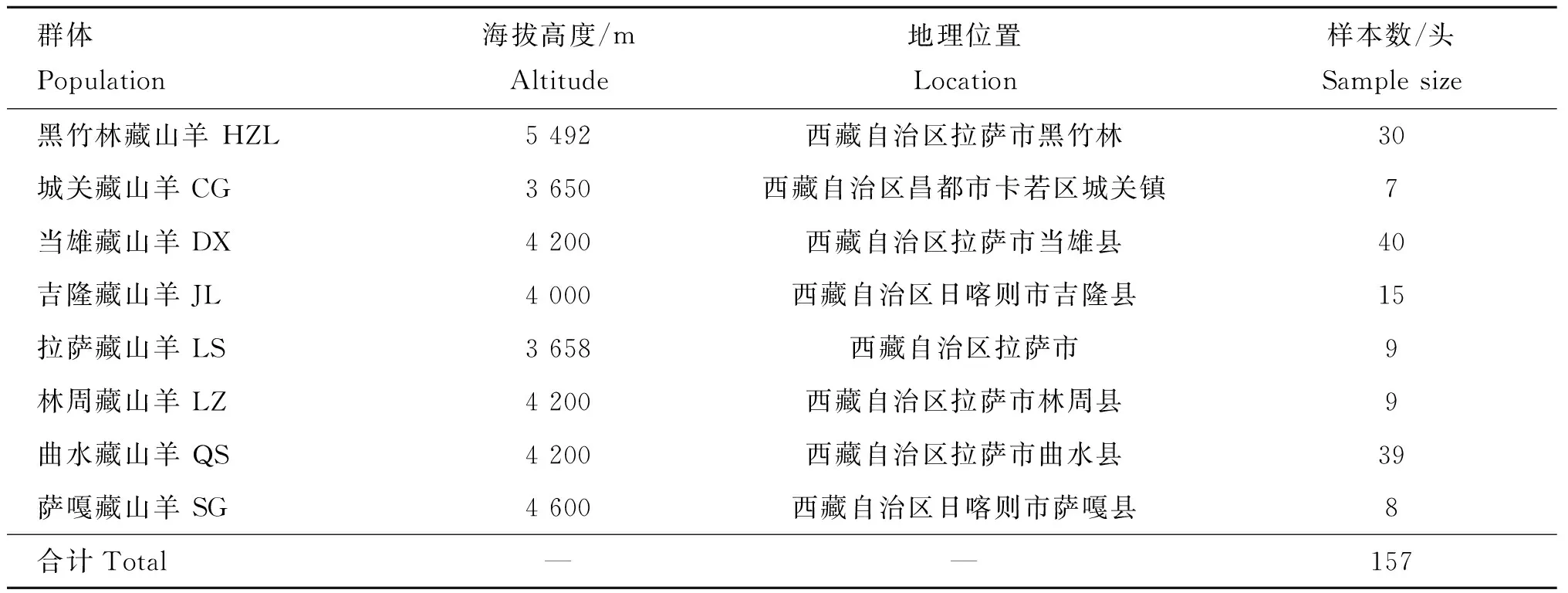
群体Population海拔高度/mAltitude地理位置Location样本数/头Samplesize黑竹林藏山羊HZL5492西藏自治区拉萨市黑竹林30城关藏山羊CG3650西藏自治区昌都市卡若区城关镇7当雄藏山羊DX4200西藏自治区拉萨市当雄县40吉隆藏山羊JL4000西藏自治区日喀则市吉隆县15拉萨藏山羊LS3658西藏自治区拉萨市9林周藏山羊LZ4200西藏自治区拉萨市林周县9曲水藏山羊QS4200西藏自治区拉萨市曲水县39萨嘎藏山羊SG4600西藏自治区日喀则市萨嘎县8合计Total——157
1.2 试验方法
1.2.1 序列扩增及测序 采用DNA提取试剂盒(TIANamp Genomic DNA Kit;北京)提取藏山羊血液基因组DNA。扩增Cytb全序列所用引物参照藏山羊mtDNA全序列(GenBank No.:KJ940969)设计(正向引物F1: 5′-AATAGGCGAAGGTTTTGAA-3′,反向引物R1:5′-GCTTTGGGTGCTGATAGTG-3′),并由成都擎科生物公司合成。本研究中,PCR为30 μL反应体系:10×缓冲液15 μL,DNA(2.5 ng·μL-1)1 μL,正反向引物(10 pmol·μL-1)各1 μL,超纯水12 μL。PCR扩增条件:94 ℃ 预变性5 min,35个循环(95 ℃ 30 s,55.3 ℃ 30 s,72 ℃ 90 s),72 ℃ 延伸10 min,之后于4 ℃保存。PCR产物在1.5% 的琼脂糖凝胶电泳及核酸检测仪上检测纯度及浓度后,送至成都擎科生物有限公司进行测序。为确保序列的准确性,每条序列均经过正反双向测定。
1.2.2 数据处理及分析 所有测得并经拼接的DNA序列用DNASTAR软件中的SeqMan 5.01(DNAstar Inc;Madison. WI)程序排列同源序列,并进行人工校正。用DnaSP 5.0软件[10]统计单倍型种类,计算核苷酸多样度(Nucleotide diversity,Pi)和单倍型多样性(Haplotypic diversity,Hd)。用MEGA 4.0 软件[11]计算变异位点、简约信息位点、遗传距离等,构建系统发育邻接树(Neighbor-Joining,NJ),以Bootstrap 1 000次重复抽样检验分支置信度[12]。利用Network 4.6.1.0软件[13]构建单倍型网络图,以直观揭示单倍型之间的亲缘关系以及基因流。Arlequin 3.5 软件[14]计算群体间遗传分化指数(FST),并根据Nm≈(1-FST)/(2FST)计算群体间的基因流值;将种群进行群体划分模式检测,用分子变异分析方法(AMOVA)估测遗传变异在种群内和种群间的分布、分化指数和对应的P值。
2 结 果
2.1 藏山羊的Cytb序列特征及遗传多样性
序列扩增及测序后经软件比对并人工校正后得到157条全长1 140 bp的Cytb基因序列(图略),共检测到33个变异位点,包含21个简约信息位点和12个单碱基替换,其中错义突变9个,无义突变24个,所有序列没有出现插入或缺失的变异位点。基于此定义了30种单倍型(H1~H30),其中CG(H1、H2)与 LS(H15、H16)为群体独享单倍型。遗传多样性结果表明(表2),LZ(Hd=0.722±0.097,Pi=0.001 8±0.000 6)与CG(Hd=0.625±0.093,Pi=0.001 8±0.000 4)的多样性指数接近,且高于其他群体;JL多样性指数(Hd=0.133±0.112,Pi=0.000 4±0.000 3)在8个群体中最低。但总的Hd(0.736±0.035)和Pi(0.001 8±0.000 2)值显示出较高水平,表明所研究西藏地区的8个藏山羊群体具有较丰富的遗传多样性。
表2藏山羊群体间遗传多样性指数及中性检验值
Table2GeneticdiversityandneutralitytestsamongTibetangoatpopulations
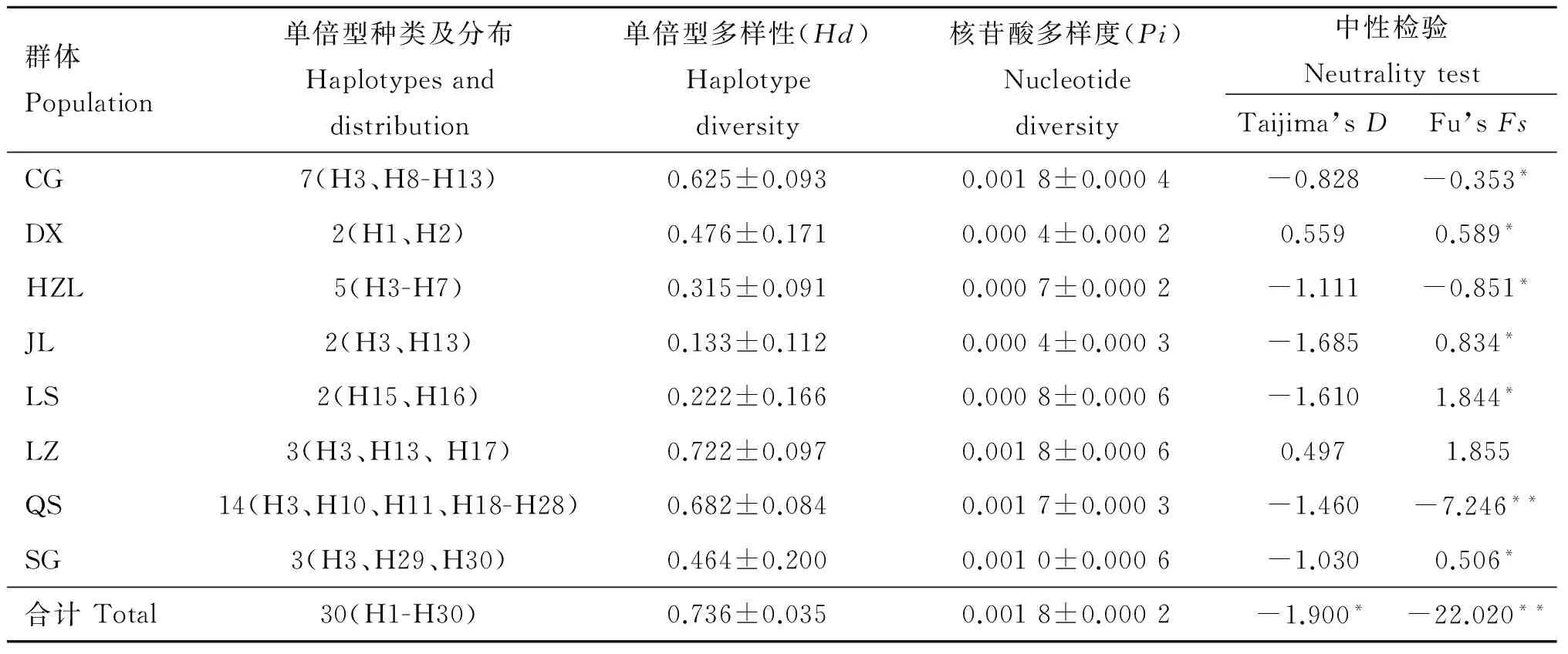
群体Population单倍型种类及分布Haplotypesanddistribution单倍型多样性(Hd)Haplotypediversity核苷酸多样度(Pi)Nucleotidediversity中性检验NeutralitytestTaijima’sDFu’sFsCG7(H3、H8-H13)0.625±0.0930.0018±0.0004-0.828-0.353*DX2(H1、H2)0.476±0.1710.0004±0.00020.5590.589*HZL5(H3-H7)0.315±0.0910.0007±0.0002-1.111-0.851*JL2(H3、H13)0.133±0.1120.0004±0.0003-1.6850.834*LS2(H15、H16)0.222±0.1660.0008±0.0006-1.6101.844*LZ3(H3、H13、H17)0.722±0.0970.0018±0.00060.4971.855QS14(H3、H10、H11、H18-H28)0.682±0.0840.0017±0.0003-1.460-7.246**SG3(H3、H29、H30)0.464±0.2000.0010±0.0006-1.0300.506*合计Total30(H1-H30)0.736±0.0350.0018±0.0002-1.900*-22.020**
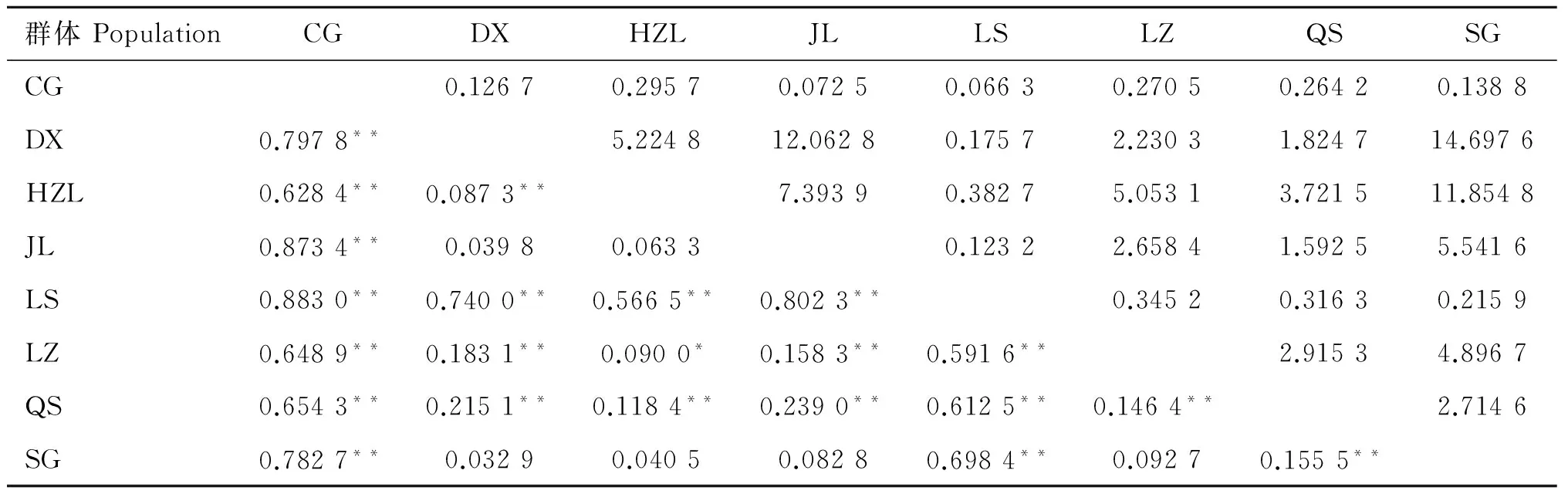
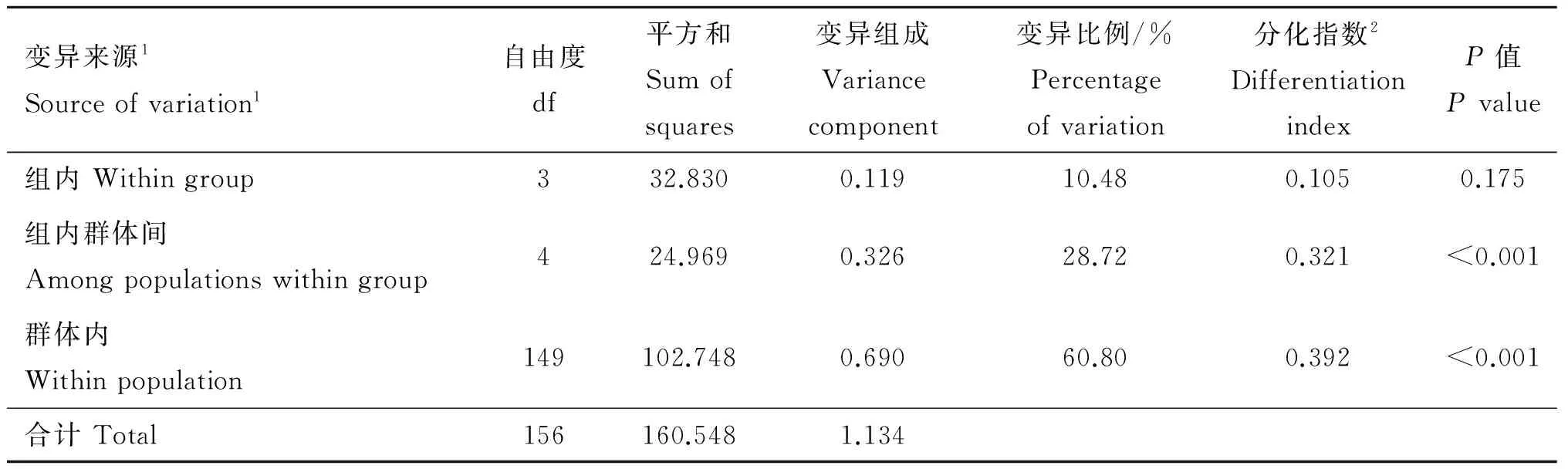
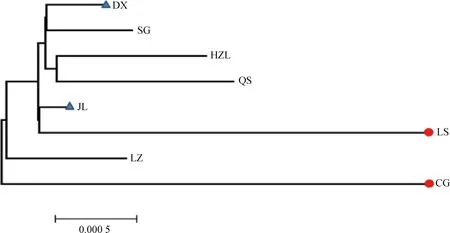
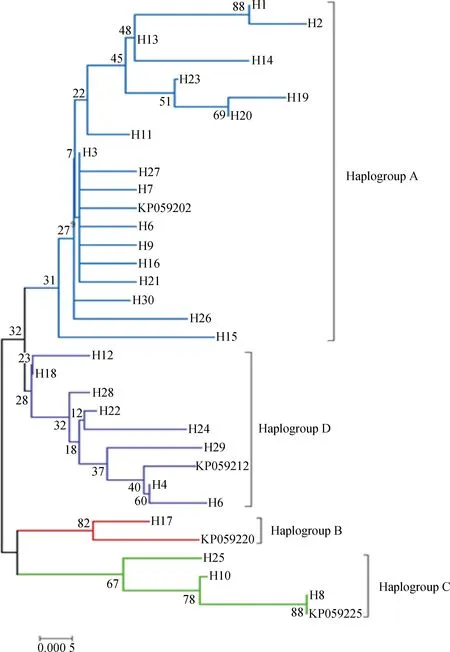
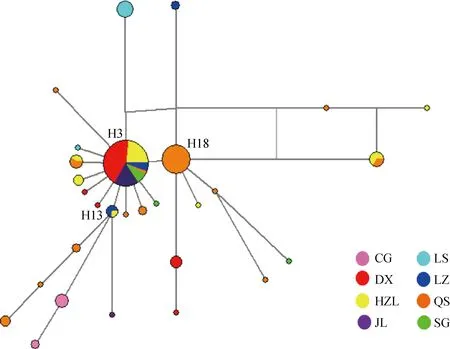
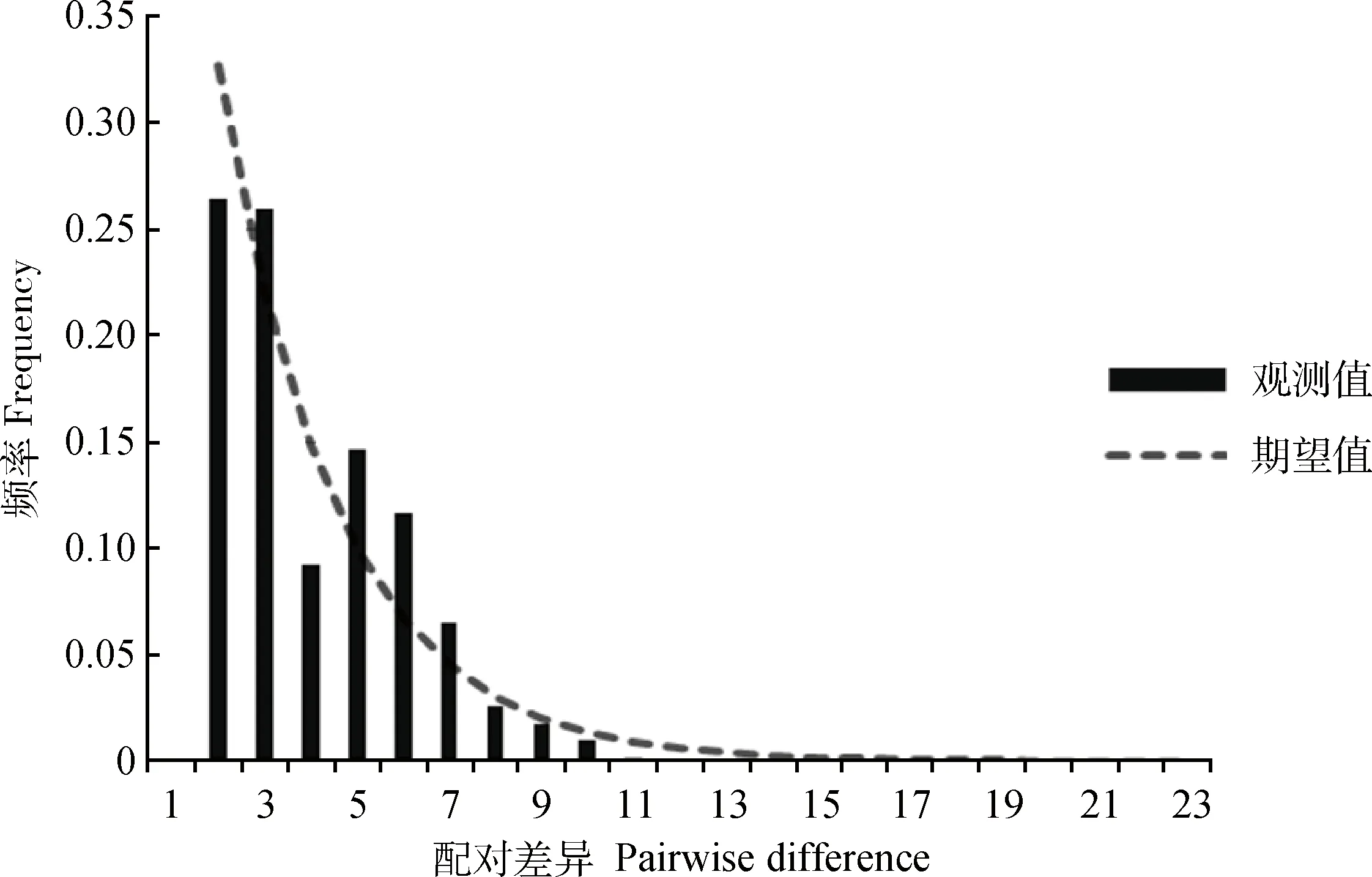
*. 0.01 群体间遗传分化指数(PairwiseFST)结果显示(表3,下三角),78.6 % 群体间的FST值达到了显着水平(P<0.05),表明大部分群体间(22/28,78.6%)发生了显着或极显着的遗传分化。其中CG与LS间差异最大(FST=0.883 0,P<0.01),表明两组群体间的遗传分化程度最高;反之DX和SG群体间分化程度最低(FST=0.032 9,P>0.05)。基因流参数值(Nm)区间为0.066 3~14.697 6(表3,上三角),表明各群体之间的基因交流水平差异较大,其中CG、LS与其他群体间的基因交流受阻(Nm< 1),表明群体间高度分离;SG和DX间基因交流较多(Nm>1),群体分化程度低,与遗传分化指数分析结果相符。AMOVA分析结果(FSC=0.392,P<0.001,表4 )表明,组内群体间遗传分化差异极显着,但变异来源所占比例较低,变异主要发生在种群内部,提示群体遗传结构差异在缩小。整体群体间基因流Nm=0.776<1,表明藏山羊整体基因流水平较低,存在一定的遗传分化。 表3藏山羊群体间的遗传分化指数FST(下三角)和基因流值Nm(上三角) Table3ThepairwiseFST(belowthediagonal)andgeneflowNm(abovethediagonal)amongtheTibetangoatpopulations 群体PopulationCGDXHZLJLLSLZQSSGCG0.12670.29570.07250.06630.27050.26420.1388DX0.7978**5.224812.06280.17572.23031.824714.6976HZL0.6284**0.0873**7.39390.38275.05313.721511.8548JL0.8734**0.03980.06330.12322.65841.59255.5416LS0.8830**0.7400**0.5665**0.8023**0.34520.31630.2159LZ0.6489**0.1831**0.0900*0.1583**0.5916**2.91534.8967QS0.6543**0.2151**0.1184**0.2390**0.6125**0.1464**2.7146SG0.7827**0.03290.04050.08280.6984**0.09270.1555** 表4藏山羊群体分子变异方差分析 Table4Analysisofmolecularvariance(AMOVA)ofTibetangoatpopulations 变异来源1Sourceofvariation1自由度df平方和Sumofsquares变异组成Variancecomponent变异比例/%Percentageofvariation分化指数2DifferentiationindexP值Pvalue组内Withingroup332.8300.11910.480.1050.175组内群体间Amongpopulationswithingroup424.9690.32628.720.321<0.001群体内Withinpopulation149102.7480.69060.800.392<0.001合计Total156160.5481.134 1.分组依据:来自同一个市区的样本被分在同组。2.组内分化指数为FCT;组内群体间分化指数为FSC;群体间分化指数为FST 1. The samples from the same city are divided into one group.2.FCT,FSCandFSTrepresent differentiation indexes within group, among populations within group and within population, respectively 图1是基于群体间遗传距离构建的群体间NJ树,结果显示,DX与SG首先聚在一起,之后依次与HZL、QS、JL、LS、LZ、CG聚类。总体上看,CG与LS的遗传关系最远,DX与JL遗传关系最近。另外,将NCBI下载的山羊Cytb4个单倍型组(Haplogroup A-D,登录号:KP059202、KP059212、KP059220和KP059225)作为参考序列,构建了30种单倍型系统发育NJ树(图2),藏山羊单倍型聚为4个支系,呈现出较强的谱系结构,不同地理来源的个体没有完全形成各自的分支,而是混杂分布部分交错,没有形成明显的地理分隔格局。其中,Haplogroup A 是主要单倍型组,由8个群体单倍型组成,其中LS、CG和JL的单倍型只在A系;Haplogroup B构成最简单,只有1种单倍型(H17);Haplogroup C 由来自QS和HZL的部分单倍型组成;Haplogroup D由HZL、QS、SG和DX的部分单倍型组成。 图1 基于藏山羊群体间遗传距离构建的亲缘关系NJ树Fig.1 An neighbor-joining (NJ) tree based on genetic distance of the Tibetan goat populations 图2 藏山羊单倍型系统发育NJ树Fig.2 An neighbor-joining (NJ) tree based on Tibetan goat haplotypes 根据30种单倍型序列数据及其出现的频率,绘制出藏山羊群体单倍型的中介网络图。图3显示,以H3为中心,呈星状向周围单倍型发散,表明群体可能发生过群体扩张;其次,H3与H18和H13连接,逐渐形成大大小小的网络分支,推断H3是主导单倍型,主要形成H3-H18和H3-H13的2个不重叠分布区的历史基因流,通过扩散或基因交流,衍生出其余单倍型,分布于不同群体中;其中QS经过基因流的扩散,分布最广。部分来自不同地区的群体的个体分享同种单倍型,但地理位置相同或相近的个体不完全聚类到同一个分支,表现了藏山羊群体地理关联性较弱。 图3 藏山羊单倍型中介网络图Fig.3 Network analysis of Tibetan goat haplotypes 中性检验值分析显示(表2),从单个地理群体看,JL、SG、LS的Tajima’sD和Fu’sFs值为一正一负,说明3个群体符合中性假说,没有发生过群体扩张事件。DX、LZ 的2个值均为正,提示在历史发展中,群体数量有过缩减;CG、HZL、QS的2个值均为负数,表明群体数量可能发生过急速扩增。从整体藏山羊种群来看,其中性检验结果均为负值(Tajima’sD=-1.900,P<0.05;Fu’sFs=-22.020,P<0.01),且错配分布曲线显示为多峰现象(图4),表明藏山羊在历史驯养过程中,可能因为部分种群的急速扩增而导致整体有种群扩增的迹象,曲线的多峰现象同时也验证了部分群体数量的缩减事件。 普遍认为,遗传多样性的高低反映了群体的环境适应能力和进化潜能[15]。本研究中,157只藏山羊共有30种单倍型,整体单倍型多样性显示出较高水平(0.736),表明整个藏山羊群体较丰富的遗传多样性。但各群体的单倍型多样性水平参差不齐,低至0.133,高达0.722,核苷酸多样度均为较低水平,表明群体间多样性水平差异较大,然而最高多样性参数值(林周藏山羊群体LZ)低于中国家养山羊地方品种的平均水平[16-17]。此外,中性检验结果和错配分析证明,在藏山羊历史驯养过程中经历过群体扩张事件,表明群体遗传变异得到了积累,整体表现出较高的多样性。然而值得注意的是,如果一个群体长期处于低水平的变异率或遗传多样性的状态下,这将成为一个重要的濒危信号[18]。吉隆县位于西藏自治区西南部,是西藏同纬度地带自然条件比较独特的地区之一,自然生态环境结构复杂,交通相对闭塞,这些因素可能导致吉隆藏山羊群体(JL)与外部群体基因交流受限,有效群体数量逐渐缩小,处于多样性快速丢失的过程中。因此,保护藏山羊群体的遗传多样性已刻不容缓。 图4 基于Cytb基因分析藏山羊群体的错配分布曲线Fig.4 Mismatch distribution curves of the Tibetan goats based on Cytb sequences FST值的大小反映了给定群体间的遗传分化水平,0~0.05表示轻度分化,0.05~0.25表示中度分化,大于0.25表示重度分化。本研究中,8个群体间遗传结构大部分有了明显分化,但分化程度有所不同。AMOVA结果显示了群体间极显着的变异程度,但变异来源所占的比例较低,提示群体间的遗传结构差异正在缩小。Nm值大小表明一个群体向另一个群体的基因流动,城关群体(CG)与拉萨群体(LS)的单倍型种类完全独立,不与其他群体共享,且基因流值均小于1,表明城关地区和拉萨地区的藏山羊群体分离程度最高。这与我们之前基于mtDNA D-loop 序列所研究的藏山羊遗传结构结果是相符的[19]。产生此结果的一个重要原因可能是,所研究群体的有效群体数量较低(如LZ、SG、CG等),且生活在封闭的高山环境中,导致当地藏山羊与邻近藏山羊群体之间的基因交流受限,进而长期保持低水平种群规模。由于有效群体数量的衰退,核苷酸替换数也极大可能被固定[20-21]。近年来,随着山羊养殖业的迅速发展,牧民为了短期效益,盲目地引入外来品种与藏山羊进行无序杂交,进一步导致藏山羊优良基因逐渐丢失,多样性降低,遗传结构差异缩小,地理分隔格局变弱。结合J.C.Avise等[22]提出的亲缘地理格局的模式分析藏山羊8个群体的遗传结构,LS和CG符合“岛屿模型”,出现独享单倍型;QS、DX、HZL、LZ、JL和SG地理屏障作用较小,使群体未彼此隔开,个体发生过相互迁移,有较多的基因交流。其中,当雄县位于西藏自治区中部,为拉萨市的纯牧业县,加上现代畜牧科技(如人工授精技术)的利用与推广,使得当雄产区的藏山羊与外群基因交流更为充分,群体间遗传分化变弱。 西藏卡若遗址出土的野生动物骨骼里发现了藏原羊的骨骼[23],说明藏族先民驯养家羊的历史可追溯至4 000年前的新石器时期。迄今为止,关于藏山羊起源的观点并不一致。在以往的研究中,B. Fan等[24]分析了13个中国山羊品种的mtDNA D-loop序列多样性,王杰等[3]利用微卫星标记分析了藏山羊群体系统发育关系,其结果均表明,藏山羊可能有多个母系起源;此外,蔡欣等[25]得到了不一致的结果,提出了藏山羊单个起源的观点,可能是由于样本采集地点、数目差异等因素导致。目前,对家养山羊的起源研究大部分是通过对mtDNA的研究将山羊大致分为了以下几个单倍型组:Haplogroup A、B、C[26]、D[27]、F[28]和G[29]。M.B.Joshi等[30]命名了一个新的单倍型组(Haplogroup E),但在加大样本量研究后,Haplogroup E被认为是A的附系[31],现今我国家养山羊共发现了4个支系[32],在本研究中也证实了这点。本研究加入NCBI下载的4种Cytb单倍型作为参考序列构建藏山羊30个单倍型系统发育树,形成4个主要的单倍型组(Haplogroup A-D),其中Haplogroup A为主要单倍型组,Haplogroup B只由LZ一种单倍型构成,结果提示,藏山羊有4个母系起源,这与已发表的藏山羊起源研究的结果是一致的[19],进一步支撑了藏山羊多个起源的假说。Haplogroup A广泛分布于全世界家养山羊品种中,有报道称Haplogroup B起源于中国[33],Haplogroup C现在主要分布在欧洲地区,K.Nomura等[34]研究表明,Haplogroup D主要分布在亚洲南部和中部地区。本研究结果表明,藏山羊可能起源于欧亚大陆,有4个母系起源。此外,藏山羊个体没有完全依据采样区域形成独立分支,而是呈现出地理上的融合,这一点在Haplogroup A中有明显体现,8个群体中均有单倍型出现在Haplogroup A,Haplogroup C和D也有部分区域混合。Haplogroup B的构成单倍型最少,可能是因为样本数不够,需要更多的样本支持。网络图中心单倍型H3的主要群体有DX和HZL,表明这2个群体古老单倍型保留得最多,分布最广。此外,曲水地区的藏山羊群体传播范围广,在进化上处于更近的时期。这样的分布现况可能是多个母系祖先的影响、世代重叠、不同地理位置的群体混合,以及伴随着基因漂变、自然选择等原因导致[35]。 本研究表明,8个藏山羊群体总的遗传多样性水平较高,但群体间的遗传多样性水平不一,差距较大。各群体间大部分发生了明显的遗传分化,但没有形成明显的地理分隔格局;8个藏山羊群体分成了A、B、C、D 4个单倍型组,提示藏山羊有4个母系起源。 [1] 王 永, 王 杰, 许期树, 等. 西藏日土藏山羊ISSR标记遗传多态性研究[J]. 畜牧兽医学报, 2010, 41(9): 1208-1212. WANG Y, WANG J, XU Q S, et al. Analysis of genetic diversity in Ritu Tibetan goats by ISSR[J].ActaVeterinariaetZootechnicaSinica, 2010, 41(9): 1208-1212. (in Chinese) [2] 秦国庆, 常 洪. 藏山羊遗传资源及利用状况[J]. 中国养羊, 1997(4): 3-5. QIN G Q, CHANG H. Genetic resources and utilization of Tibetan goats[J].ChinaHerbivores, 1997(4): 3-5. (in Chinese) [3] 王 杰, 华太才让, 欧阳熙, 等. 藏山羊微卫星DNA多态性研究[J]. 中国草食动物, 2006(S1): 122-126. WANG J, HUATAI C R, OUYANG X, et al. Study on microsatellite DNA polymorphism of Tibetan goats[J].ChinaHerbivores, 2006(S1): 122-126. (in Chinese) [4] 文淑义, 王秋福, 王德元, 等. 辽宁绒山羊适应性观察及与藏山羊杂交试验报告[J]. 四川草原, 1995(4): 42-48. WEN S Y, WANG Q F, WANG D Y, et al. Adaptive observation of Liaoning cashmere goats and a report on hybrid experiment with Tibetan goats[J].JournalofSichuanGrassland, 1995(4): 42-48. (in Chinese) [5] 任称罗尔日, 周光领, 周炬钟, 等. 南江黄羊与藏山羊杂交的效果分析[J]. 四川畜牧兽医, 2000, 27(S1): 59-60, 62. RENCHEN L E R, ZHOU G L, ZHOU J Z, et al. Heterosis analysis of crossing Nanjiang yellow goats with Tibetan goats[J].SichuanAnimal&VeterinarySciences, 2000, 27(S1): 59-60, 62. (in Chinese) [6] 王燕军, 张月欢, 肖文平. 丹巴县引进南江黄羊杂交改良本地藏山羊的实践[J]. 中国畜牧兽医文摘, 2015, 31(4): 57, 64. WANG Y J, ZHANG Y H, XIAO W P. The practice of introducing the Nanjiang yellow goat in Danba County to improve local Tibetan goats[J].ChineseAbstractofAnimalHusbandryandVeterinaryMedicine, 2015, 31(4): 57, 64. (in Chinese) [7] 郭淑珍, 马登录, 牟永娟, 等. 内蒙古白绒山羊及其与藏山羊杂交后代在甘南州的适应性观察[J]. 中国草食动物, 2008, 28(3): 38-40. GUO S Z, MA D L, MOU Y J, et al. The adaptive observation of Mongolia white cashmere goat and its hybrid offspring with Tibetan goat in Gannan[J].ChinaHerbivores, 2008, 28(3): 38-40. (in Chinese) [9] LIU J, WANG C M, FU D L, et al. Phylogeography ofNanoranaparkeri(Anura: Ranidae) and multiple refugia on the Tibetan Plateau revealed by mitochondrial and nuclear DNA[J].SciRep, 2015, 5: 9857. [10] LIBRADO P, ROZAS J. DnaSP v5: a software for comprehensive analysis of DNA polymorphism data[J].Bioinformatics, 2009, 25(11): 1451-1452. [11] TAMURA K, DUDLEY J, NEI M, et al. MEGA4: Molecular Evolutionary Genetics Analysis (MEGA) software version 4.0[J].MolBiolEvol, 2007, 24(8): 1596-1599. [12] SANDERSON M J. Confidence limits on phylogenies: the bootstrap revisited[J].Cladistics, 1989, 5(2): 113-129. [13] BANDELT H J, FORSTER P, RÖHL A. Median-joining networks for inferring intraspecific phylogenies[J].MolBiolEvol, 1999, 16(1): 37-48. [14] EXCOFFIER L, LISCHER H E L. Arlequin suite ver 3.5: a new series of programs to perform population genetics analyses under Linux and windows[J].MolEcolResour, 2010, 10(3): 564-567. [15] LIU J B, DING X Z, ZENG Y F, et al. Genetic diversity and phylogenetic evolution of Tibetan sheep based on mtDNAD-loop sequences[J].PLoSOne, 2016, 11(7): e0159308. [16] 黄勤华, 刘若余, 吴 芸, 等. 贵州4个山羊群体mtDNACytb基因多态性研究[J]. 中国畜牧杂志, 2010, 46(1): 4-7. HUANG Q H, LIU R Y, WU Y, et al. Study on polymorphism of cytochrome b gene in four Guizhou goat breeds[J].ChineseJournalofAnimalScience, 2010, 46(1): 4-7. (in Chinese) [17] 黄勤华, 刘若余, 邓书堪. 贵州白山羊与湖南马头山羊mtDNACytb基因系列比较及系统进化研究[J]. 安徽农业科学, 2009, 37(25): 11897-11899. HUANG Q H, LIU R Y, DENG S K. Comparison and Systematic evolution study of cytochrome b gene (Cytb) between Guizhou white goat and Hunan Matou goat[J].JournalofAnhuiAgriculturalSciences, 2009, 37(25): 11897-11899. (in Chinese) [18] LI L, ZHANG J, ZHU J Q, et al. Genetic diversity of nine populations of the black goat (Caprahircus) in Sichuan, PR China[J].ZoologSci, 2006, 23(3): 229-234. [19] DENG J, FENG J, LI L, et al. Polymorphisms, differentiation, and phylogeny of 10 Tibetan goat populations inferred from mitochondrialD-loop sequences[J].MitochondrDNA, 2017: 1-7. [20] KIMURA M. On the probability of fixation of mutant genes in a population[J].Genetics, 1962, 47(6): 713-719. [21] NEI M, MARUYAMA T, CHAKRABORTY R. The bottleneck effect and genetic variability in populations[J].Evolution, 1975, 29(1): 1-10. [22] AVISE J C, ARNOLD J, BALL R M, et al. Intraspecific phylogeography: the mitochondrial DNA bridge between population genetics and systematics[J].AnnuRevEcolSyst, 1987, 18: 489-522. [23] WANG Y, WANG J, ZI X D, et al. Genetic diversity of Tibetan goats of plateau type using microsatellite markers[J].ArchivTierzucht, 2011, 54(2): 188-197. [24] FAN B, CHEN S L, KIJAS J H, et al. Phylogenetic relationships among Chinese indigenous goat breeds inferred from mitochondrial control region sequence[J].SmallRuminRes, 2007, 73(1-3): 262-266. [25] 蔡 欣, 泽让东科, 苟兴能, 等. 川渝黔地区山羊起源于A和B两母系遗传谱系[J]. 四川大学学报: 自然科学版, 2012, 49(4): 887-894. CAI X, TSERANG D M, GOU X N, et al. Goats from Sichuan, Chongqing and Guizhou were originated from A and B maternal lineages[J].JournalofSichuanUniversity:NaturalScienceEdition, 2012, 49(4): 887-894. (in Chinese) [26] LUIKART G, GIELLY L, EXCOFFIER L, et al. Multiple maternal origins and weak phylogeographic structure in domestic goats[J].ProcNatlAcadSciUSA, 2001, 98(10): 5927-5932. [27] SULTANA S, MANNEN H, TSUJI S. Mitochondrial DNA diversity of Pakistani goats[J].AnimGenet, 2003, 34(6): 417-421. [28] SARDINA M T, BALLESTER M, MARMI J, et al. Phylogenetic analysis of Sicilian goats reveals a new mtDNA lineage[J].AnimGenet, 2006, 37(4): 376-378. [29] NADERI S, REZAEI H R, POMPANON F, et al. The goat domestication process inferred from large-scale mitochondrial DNA analysis of wild and domestic individuals[J].ProcNatlAcadSciUSA, 2008, 105(46): 17659-17664. [30] JOSHI M B, ROUT P K, MANDAL A K, et al. Phylogeography and origin of Indian domestic goats[J].MolBiolEvol, 2004, 21(3): 454-462. [31] 武艳平. 中国部分山羊品种多样性及系统进化分析[D]. 北京: 中国农业科学院, 2008. WU Y P. Diversity and phylogenetic relationships of some Chinese goat breeds[D]. Beijing: Chinese Academy of Agricultural Sciences, 2008. (in Chinese) [32] LIU R Y, YANG G S, LEI C Z. The genetic diversity of mtDNAD-loop and the origin of Chinese goats[J].ActaGenetSin, 2006, 33(5): 420-428. [33] LI M. New evidence discovery for maternal lineage B of goats (Caprahircus) in Chinese tropical zone[J].GeophysResLett, 2013, 28(24): 4525-4528. [34] NOMURA K, YONEZAWA T, MANO S, et al. Domestication process of the goat revealed by an analysis of the nearly complete mitochondrial protein-encoding genes[J].PLoSOne, 2013, 8(8): e67775. [35] ZHAO Y J, ZHANG J H, ZHAO E H, et al. Mitochondrial DNA diversity and origins of domestic goats in southwest China (Excluding Tibet)[J].SmallRuminRes, 2011, 95(1): 40-47.2.2 藏山羊群体遗传分化与基因交流
2.3 藏山羊的群体遗传关系与系统发育
2.4 藏山羊单倍型网络关系
2.5 中性检验与种群动态分析
3 讨 论
3.1 藏山羊遗传资源的保护
3.2 藏山羊群体的遗传结构
3.3 藏山羊群体的多个母系起源
4 结 论



