李 畅,库旭钢,王俊伟,李 静,朱 玲,何启盖*(1.华中农业大学农业微生物学国家重点实验室,武汉 430070;2.华中农业大学动物医学院,武汉 430070)
猪圆环病毒(PCV)是一种单股环状负链的DNA病毒,为圆环病毒科,圆环病毒属成员,包括圆环病毒1型(PCV1)和圆环病毒2型(PCV2)两种基因型[1]。PCV1是一种猪肾上皮细胞(PK-15)污染物,无致病性[2]。PCV2在20世纪90年代首次发现于加拿大的断奶仔猪多系统衰竭综合征(PMWS)病猪中,被认为是圆环病毒相关疾病(PCVAD)的主要病原,主要导致断奶仔猪多系统衰竭综合征(PMWS)、猪皮炎肾病综合征(PDNS)、繁殖障碍等,给养猪业造成严重的经济损失[3]。
2016年以来,多篇报道称通过宏基因组等技术从皮炎肾病综合征和繁殖障碍猪的病料以及口腔液中鉴定出一种新型猪圆环病毒,即猪圆环病毒3型(PCV3)[4-7]。根据已有的报道,PCV3在美国、中国和韩国呈流行趋势,且可能存在跨国传播情况[5,7-8]。
鉴于PCV2和PCV3感染能导致相似的临床症状,因此,十分有必要建立一种快速、高效、敏感、特异的检测方法来区分PCV3和其他病毒感染。荧光定量PCR技术凭借其快速、简便、高效、特异、稳定等特点在目前的病原检测工作中被广为应用[9]。
本研究主要目的在于建立一种快速、高效、特异检测PCV3的实时荧光定量PCR方法,并用该方法对湖北地区病料进行PCV3检测,评估PCV3在该地区的流行情况。
1 材料与方法
1.1 材料
1.1.1 样品,毒株和菌株 临床检测样品为华中农业大学动物疫病诊断中心门诊临床病死猪的DNA样品。猪圆环病毒2b亚型(porcine circovirus type 2b,PCV2b)、猪圆环病毒2d亚型(porcine circovirus type 2d,PCV2d)、猪流行性腹泻病毒(porcine epidemic diarrhea virus,PEDV)、猪繁殖与呼吸综合征病毒(porcine reproductive and respiratory syndrome virus,PRRSV)、猪轮状病毒(porcine rotavirus,PoRV)、副猪嗜血杆菌(Haemophilusparasuis,Hps),猪胸膜肺炎放线杆菌(Actinobacilluspleuropneumoniae,App)、猪支气管败血波氏杆菌(Brodetellabronchiseptica,Bb)、多杀性巴氏杆菌(Pasteurellamultocida,Pm)等均由本实验室保存。
1.1.2 主要试剂和仪器 Viral-DNA Kit(病毒DNA提取试剂盒,D3892)购自美国OMEGA公司;TIANamp Genomic DNA Kit(血液/细胞/组织基因组DNA提取试剂盒,DP304)购自北京天根生化科技有限公司;EasyPure Plasmid MiniPrep Kit(质粒小提试剂盒,EM101)购自北京全式金生物技术有限公司;Biomiga Gel/PCR Extraction Kit(胶/PCR产物回收试剂盒,DC3511)购自美国Biomiga公司;HieffTMqPCR SYBR®Green Master Mix (Low Rox Plus)购自上海翊圣生物科技有限公司;iTaqTMUniversal Probes Supermix(172-5134)、384孔PCR反应板、96孔PCR反应板以及S1000TMThermal Cycler(PCR仪)均购自美国Bio-Rad公司、ViiATM7 实时荧光定量PCR仪为美国ABI公司产品。
1.1.3 标准质粒的构建 根据NCBI上已公布的PCV3基因组序列(登录号:MF079254、 MF079253、KY996345、KY996340、KY996338、KY778777、KY075994、KY075988、KX966193、KX898030、KX778720等)以及相应的Cap基因序列,设计引物(表1),以PCV3阳性样品DNA为模板,扩增得到Cap基因片段(645 bp),将该片段连接到pMDTM18-T Vector载体上,筛选阳性克隆并扩大培养相应菌液,EasyPure Plasmid MiniPrep Kit提取标准质粒,并使用Nano drop测定标准质粒浓度,计算拷贝数,并稀释成1.29×102~1.29×109拷贝·μL-1。
1.1.4 定量引物和探针的设计与合成 下载GenBank中已公布的PCV3序列,比较其与PCV1、PCV2的相似性,并根据其保守区域使用Primer Premier 5软件设计4对引物(表2),使用SYBR荧光定量PCR验证其能否有效地检测该片段。根据SYBR荧光定量PCR结果,选定引物对F2/R2和F3/R3扩增序列的交集区域,设计一条TaqMan探针(表2)。引物和探针均由南京金斯瑞生物科技有限公司合成。
表1本研究中使用的PCV3ORF2基因的引物
Table1TheprimersforPCV3ORF2geneinthisstudy
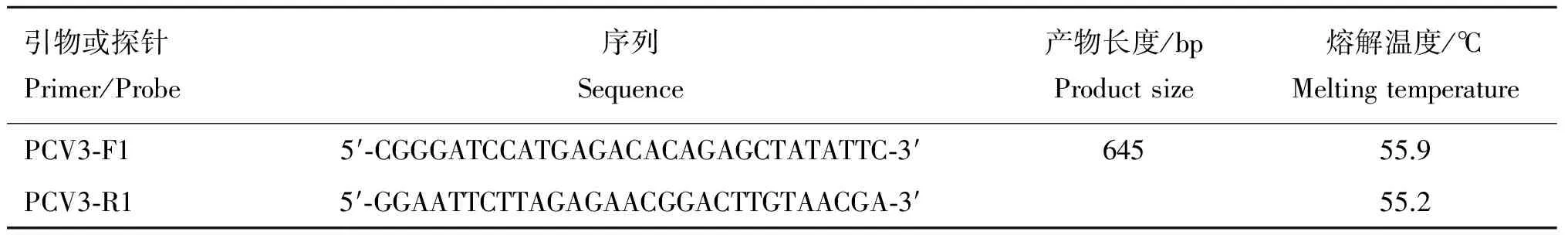
引物或探针Primer/Probe序列Sequence产物长度/bpProductsize熔解温度/℃MeltingtemperaturePCV3⁃F15′⁃CGGGATCCATGAGACACAGAGCTATATTC⁃3′64555.9PCV3⁃R15′⁃GGAATTCTTAGAGAACGGACTTGTAACGA⁃3′55.2
表2本研究中所使用的定量引物和探针
Table2SequenceofqPCRprimersandTaqManprobeinthisstudy

引物或探针Primer/Probe序列Sequence产物长度/bpProductsize熔解温度/℃MeltingtemperaturePCV3⁃F15′⁃CGGACTTGTAACGAATCCAAACT⁃3′7855.9PCV3⁃R15′⁃GGAGCATTTATGTCCCGGAAA⁃3′55.2PCV3⁃F25′⁃TATTCATTAGGAGGCCCACA⁃3′14252.6PCV3⁃R25′⁃GCAGTTTCCCATTCGTTTAG⁃3′52.1PCV3⁃F35′⁃GGAGACGACGACGCCACAG⁃3′14460.3PCV3⁃R35′⁃CGTGCCAGGGCTTGTTATTC⁃3′56.7PCV3⁃F45′⁃GCGGAAAGTTCCACTCGTAA⁃3′11455.3PCV3⁃R45′⁃GAAGAGGCTTTGTCCTGGGT⁃3′56.6PCV3⁃Probe5′⁃FAM⁃ACTCCACCATGAACGTCATTTCC⁃TAMRA⁃3′57.7
1.2 方法
1.2.1 荧光定量PCR反应体系优化 以1.29×102~1.29×109拷贝·μL-1的标准质粒为模板,以FI/R1、F2/R2、F3/R3、F4/R4为引物,按照说明书使用HieffTMqPCR SYBR®Green Master Mix (Low Rox Plus)鉴定四对引物能否有效地检测PCV3。再按照SYBR定量PCR的结果,根据F2/R2和F3/R3引物对所扩增区域的公共序列部分,设计TaqMan探针。再以1.29×105拷贝·μL-1的标准质粒为模板,在10 μL的PCR反应体系中,分别加入终浓度为0.25、0.50、1.00、2.00 μmol·L-1的引物F2/R2和终浓度为0.25、0.50、1.00、2.00 μmol·L-1的Probe,按照说明书使用iTaqTMUniversal Probes Supermix,根据结果选定最佳的引物浓度和探针浓度。
1.2.2 标准曲线的构建 按照说明书使用iTaqTMUniversal Probes Supermix,加入最佳浓度的引物和探针,以1.29×102~1.29×109拷贝·μL-1的标准质粒为模板,进行荧光定量PCR反应。将所得结果中的Ct值为Y轴、1.29 × lg(标准质粒,拷贝·μL-1)为X轴构建标准曲线。
1.2.3 敏感性和特异性试验 以1.29×100~1.29×109拷贝·μL-1的标准质粒为模板,用本研究所构建的荧光定量PCR和本实验室所使用的常规PCR[4]进行检测,确定两种方法的敏感性并作比较;以阳性PCV2b、PCV2d、PEDV、PRRSV、PoRV、HPS、App、Bb、Pm DNA或者cDNA为模板,进行荧光定量PCR以确定该方法的特异性。
1.2.4 重复性试验 分别以1.29×102、1.29×105、1.29×108拷贝·μL-1的标准质粒为模板,进行3次批内和批间荧光定量PCR,根据结果中的Ct值计算批次内变异系数和批次间变异系数,并以此判定该方法的重复性。
1.2.5 临床样品的检测 应用本研究建立的TaqMan荧光定量PCR方法和常规PCR方法检测2016—2017年间湖北省及周边地区所收集的124份DNA样品,将Ct值小于30的样品判定为阳性,同时使用本实验室建立的PCV2荧光定量PCR方法检测样品中的PCV2[10],以此评估PCV3和PCV2在该地区的流行情况。
2 结 果
2.1 荧光定量PCR引物和探针的选定
以1.29×102~1.29×109拷贝·μL-1的标准质粒为模板,分别以FI/R1、F2/R2、F3/R3、F4/R4为引物,按照说明书使用HieffTMqPCR SYBR®Green Master Mix (Low Rox Plus)鉴定四对引物能否有效地检测PCV3,得到4条标准曲线(图略),均呈良好的线性关系,证明四对引物均能有效检测PCV3。因为F2/R2和F3/R3所扩增产物的序列具有交叉区域,故根据此交叉区域设计探针。
2.2 荧光定量PCR反应体系优化
以提高扩增效率和敏感度为标准,以1.29×105拷贝·μL-1的标准质粒为模板,对F2/R2引物浓度和探针浓度进行优化。优化后的反应体系(10 μL):2× iTaqTMUniversal Probes Supermix 5 μL,F2/R2(10 μmol·L-1)各0.5 μL,TaqMan Probe(5 μmol·L-1)0.5 μL,待检模板(100 ng最优)2 μL,补加Nuclease-free water至10 μL。反应条件:50 ℃ 2 min,95 ℃ 10 min;95 ℃ 15 s,60 ℃ 1 min,40个循环。
2.3 标准曲线的构建
利用优化后的定量PCR反应对1.29×102~1.29×109拷贝·μL-1的标准质粒进行定量,以Ct值为Y轴,1.29 × lg(标准质粒,拷贝·μL-1)为X轴构建标准曲线,其标准方程为:Y=-3.258X+36.721,相关系数R2为0.996(图1),扩增效率为100.72%,说明Ct值与模板量之间呈良好的线性关系。本方法判定标准:Ct值低于30,且出现典型的扩增曲线的样品为阳性;Ct值大于32或无有效Ct值的样品为阴性。
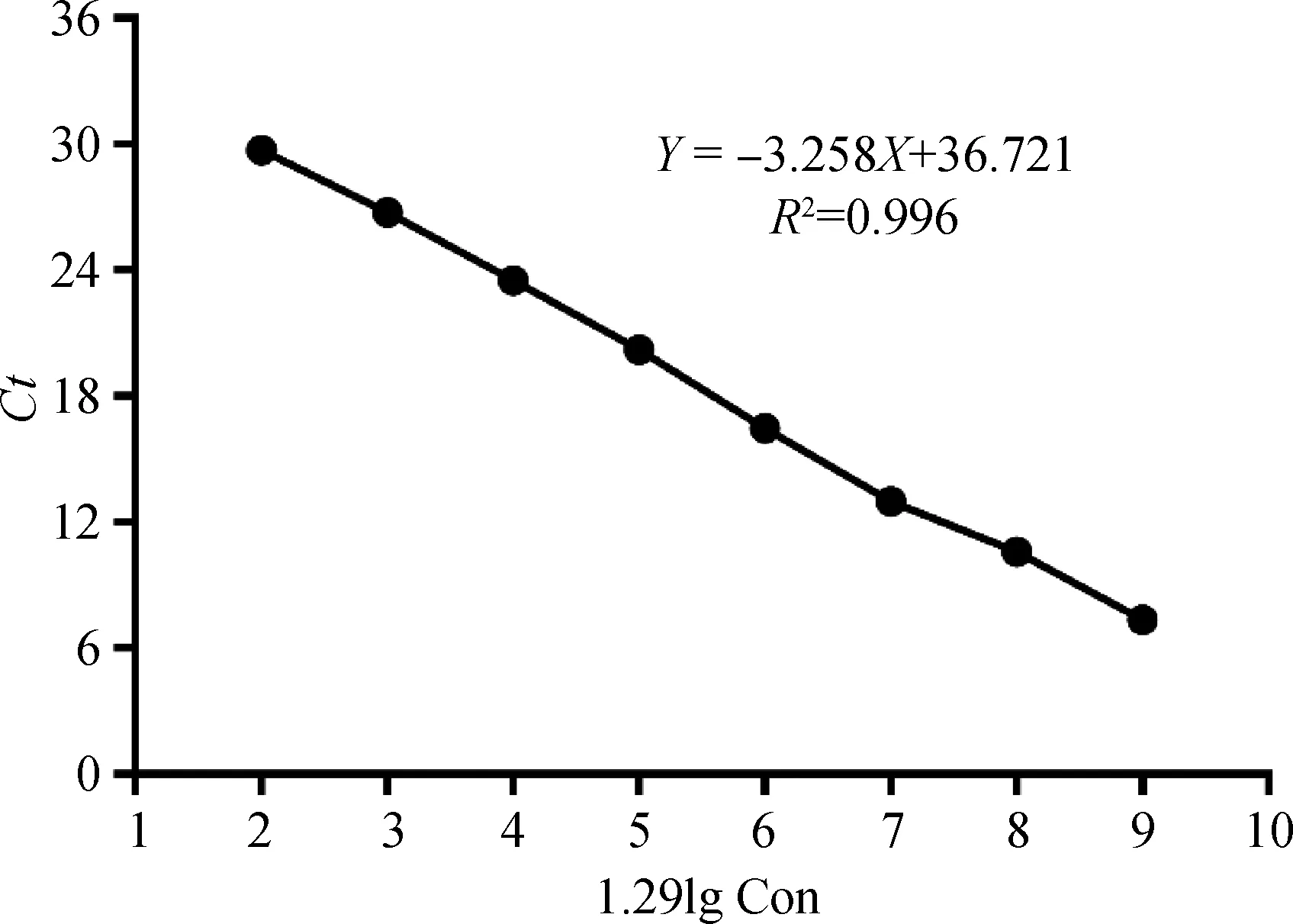
图1 PCV3 TaqMan探针荧光定量PCR标准曲线Fig.1 Standard curve of PCV3 ORF3 genes by TaqMan probe real-time PCR
2.4 敏感性和特异性试验
以1.29×101~1.29×109拷贝·μL-1的标准质粒为模板,分别使用本研究所建立的荧光定量PCR方法和普通PCR方法扩增,检验定量PCR和常规PCR的敏感性并做对比,结果显示定量PCR方法的最低检出量为1.29×102拷贝·μL-1(图2A),而常规PCR在1.29×104拷贝·μL-1时仅能从凝胶电泳图上看到微弱的目的条带(图2B),表明本研究所建立的定量PCR方法敏感性是常规PCR的1 000倍左右。
用建立的PCV3荧光定量PCR方法检测PCV2b、PCV2d、PEDV、PRRSV、PoRV、Hps, App、Bb、Pm DNA或者cDNA样品,均未出现阳性扩增现象,表明该方法特异性良好(图3)。
2.5 重复性试验
选取1.29×102、1.29×105、1.29×108拷贝·μL-1的标准质粒为模板,用本研究所建立的荧光定量PCR分别进行3次批内重复和3个批间检测,各个浓度的Ct值、批内变异系数和批间变异系数见表3,该方法批内重复和批间重复变异系数均小于1.5%,说明本研究所建立的荧光定量PCR方法具有良好的重复性。
2.6 临床样品的检测
结果显示,常规PCR检测出样品中PCV3阳性率为1.62% (2/124),本研究所建立定量方法的阳性率为8.06% (10/124),这与袁万哲研究团队使用TaqMan荧光定量PCR检测的PCV3阳性率12.5%(14/112)基本一致[11],但是显着低于马静云研究团队使用SYBR荧光定量PCR检测的PCV3阳性率86.70% (176/203)[12],这可能与样品来源有关,本研究中所用样品来自科前生物股份有限公司,而其他方法检测的样品主要源于具有典型病症的猪。检测阳性样品和PCV3标准质粒均呈现正常的“S”形扩增曲线,阴性对照无扩增曲线。样品中PCV2的阳性率为72.78% (90/124),PCV2和PCV3共感染率为8.06% (10/124)。
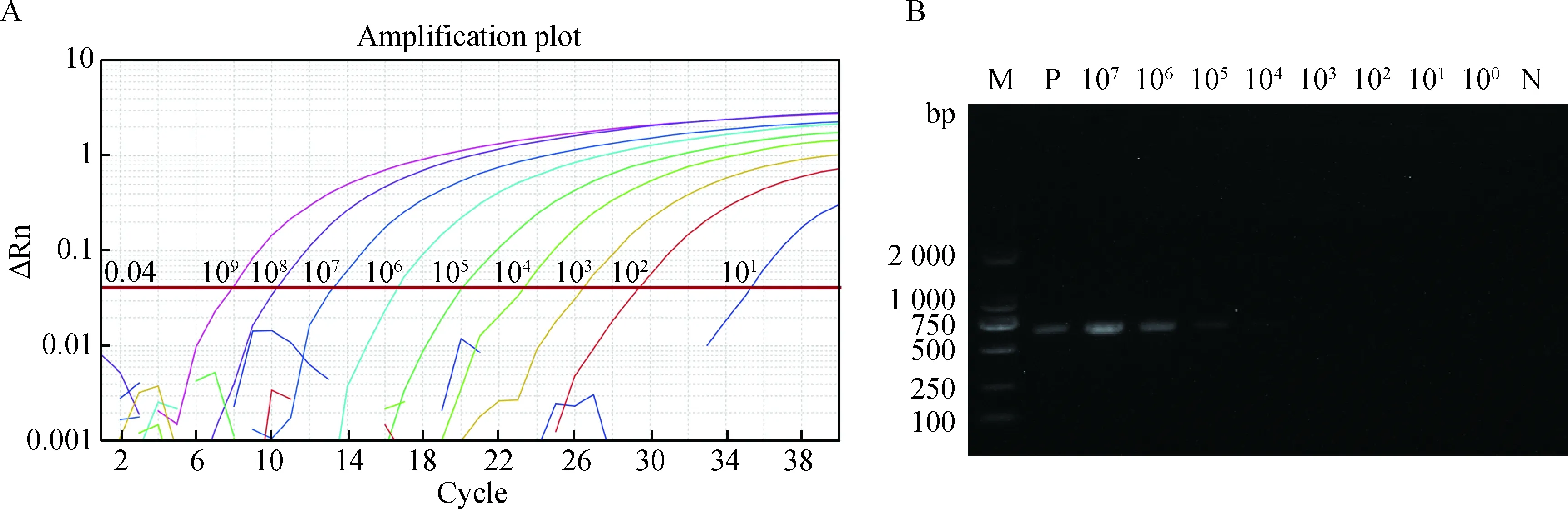
A.荧光定量PCR方法结果(标准质粒浓度1.29×109~1.29×101拷贝·μL-1);B.普通PCR方法结果(标准质粒浓度1.29×107~1.29×100拷贝·μL-1);M. DNA相对分子质量标准;P. 阳性对照;N.阴性对照A. Result of real-time qPCR (standard plasmid concentration: 1.29×109 - 1.29×101 copies·μL-1); B. Result of common PCR (standard plasmid concentration: 1.29×107 - 1.29×100 copies·μL-1); M. DNA marker; P. Positive control; N. Negative control
图2 敏感性试验Fig.2 Sensitivity test
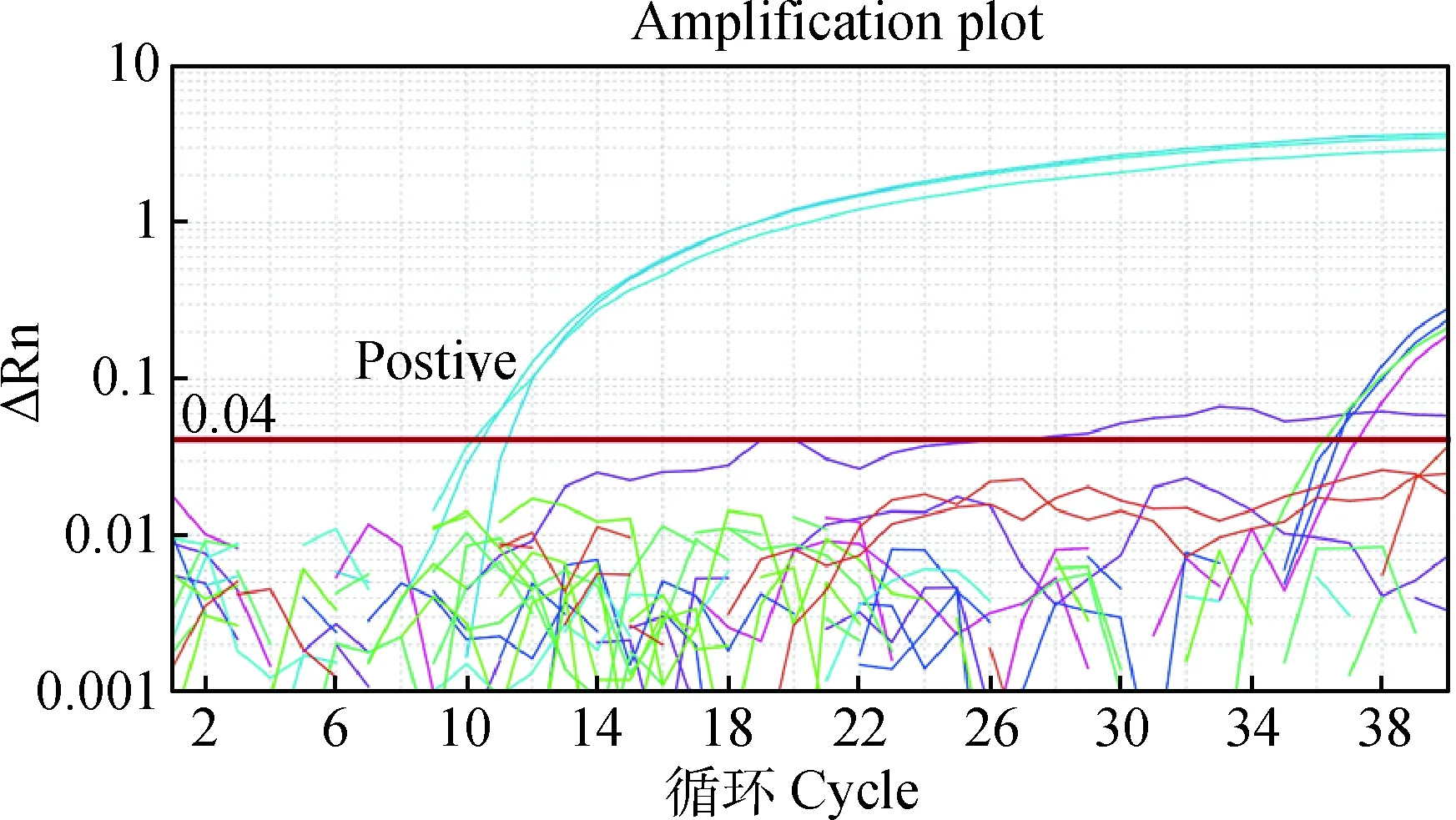
PCV3样品为阳性;PCV2b、PCV2d、PEDV、PRRSV、PoRV、Hps、App、Bb、Pm DNA或者cDNA样品均为阴性PCV3 samples were positive; PCV2b, PCV2d, PEDV, PRRSV, PoRV, Hps, App, Bb, Pm DNA or cDNA samples were negative
图3 特异性检验Fig.3 The specificity test
3 讨 论
2016年以来,中国、美国、韩国、波兰等多个国家均报道称通过宏基因组等技术发现PCV3,其在各国的流行情况呈上升趋势,并且各国之间PCV3的同源性和序列相似性很高,提示PCV3可能存在跨国传播情况[5-8]。PCV3感染能导致和PCV2感染相似的临床症状,并且在PDNS(皮炎肾炎综合征)猪的心、肾、肺、淋巴结组织以及皮肤组织等处均能检测到病毒核酸的存在[4-5]。本实验室前期构建的常规PCR方法[8],灵敏度亟待提高,因此有必要建立一种敏感性更高的检测方法来检测PCV3。
表3荧光定量PCR的批内和批间重复检测结果
Table3Theintra-andinter-assayCVsforCtvaluesresultofreal-timeqPCR
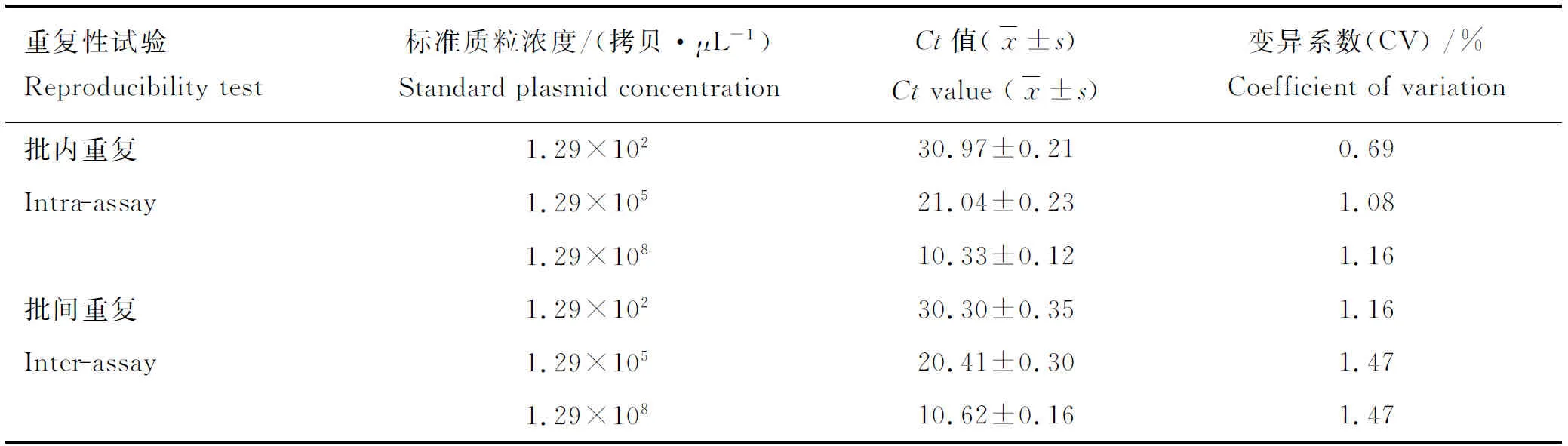
重复性试验Reproducibilitytest标准质粒浓度/(拷贝·μL-1)StandardplasmidconcentrationCt值(x±s)Ctvalue(x±s)变异系数(CV)/%Coefficientofvariation批内重复1.29×10230.97±0.210.69Intra⁃assay1.29×10521.04±0.231.081.29×10810.33±0.121.16批间重复1.29×10230.30±0.351.16Inter⁃assay1.29×10520.41±0.301.471.29×10810.62±0.161.47
荧光定量PCR技术凭借其快速、高效、特异和稳定等特点在目前的病原检测工作中被广为应用[9]。目前最常用的荧光定量方法主要分为两种,SYBR染料为基础的荧光定量PCR方法和TaqMan探针为基础的荧光定量PCR方法。相比于染料法荧光定量PCR方法,TaqMan探针定量PCR技术拥有更高的敏感性[13-14],故本研究中采用两种定量方法相结合的方式,先采用染料法对初设计的多对定量引物进行初步筛选,再根据筛选结果,设计TaqMan探针,建立TaqMan探针定量PCR检测方法雏形并逐步优化完善。本研究中引物和探针的设计基于NCBI中已公布的所有PCV3全基因序列以及相应的PCV3Cap基因序列,并且将所设计引物和探针与Sus scrofa Refseq mRNA数据库,PCV1、不同基因型PCV2、PEDV等多种猪病原体基因序列进行比对,确保本研究定量方法的特异性。本研究所建立的TaqMan荧光定量PCR最低检出限量为1.29×102拷贝·μL-1,明显高于本实验室之前使用的常规PCR方法[8],与袁万哲研究团队建立TaqMan荧光定量PCR(1.02 × 102拷贝·μL-1)[11]和马静云研究团队建立的SYBR荧光定量PCR[12]灵敏性(1.73×102拷贝·μL-1)相近,均能特异性地检测PCV3,且稳定性更高。
利用本研究所建立的TaqMan荧光定量PCR方法检测2016-2017年间湖北省及其周边地区猪场的病猪,核酸样品中PCV3单独存在情况和PCV2/PCV3共存在情况,PCV3的单独感染率为8.06% (10/124),PCV2/3共感染率为8.06% (10/124);在之前的研究中PCV2/PCV3共感染分别是12.5%(14/112)[11]和15.8%(35/222)[8]。以上研究均证明PCV2感染和PCV3的感染之间存在密切的联系,两者之间的共感染机制研究仍有待深入研究。临床中的PCV3感染情况需要持续监测,PCV3的病毒分离以及PCV3与PCV2的共感染机制需要更多的关注。
4 结 论
成功建立PCV3 TaqMan荧光定量PCR检测方法,能够灵敏准确的检测临床样品中PCV3的存在情况,为PCV3的监测和防控提供了有力的手段。
参考文献(References):
[1] MANKERTZ A, ÇALISKAN R, HATTERMANN K, et al. Molecular biology ofPorcinecircovirus:analyses of gene expression and viral replication[J].VetMicrobiol, 2004, 98(2):81-88.
[2] TISCHER I, RASCH R, TOCHTERMANN G. Characterization of papovavirus-and picornavirus-like particles in permanent pig kidney cell lines[J].ZentralblBakteriolOrigA, 1974, 226(2):153-167.
[3] OPRIESSNIG T,MENG X J, HALBUR P G. Porcine circovirus type 2-associated disease: Update on current terminology,clinical manifestations, pathogenesis, diagnosis, and intervention strategies[J].JVetDiagnInvest, 2007, 19(6):591-615.
[4] PHAN T G,GIANNITTI F,ROSSOW S, et al. Detection of a novel circovirus PCV3 in pigs with cardiac and multi-systemic inflammation[J].VirolJ, 2016, 13(1):184.
[5] PALINSKI R, PIEYRO P, SHANG P, et al. A novel porcine circovirus distantly related to known circoviruses is associated with porcine dermatitis and nephropathy syndrome and reproductive failure[J].JVirol, 2016, 91(1):e01879-16.

[7] KWON T, YOO S J, PARK C K, et al. Prevalence of novel porcine circovirus 3 in Korean pig populations[J].VetMicrobiol, 2017, 207:178-180.
[8] KU X, CHEN F, LI P, et al. Identification and genetic characterization of porcine circovirus type 3 in China[J].TransboundEmergDis, 2017, 64(3):703-708.
[9] MACKAY I M,ARDEN K E,NITSCHE A.Real-time PCR in virology[J].NucleicAcidsRes, 2002, 30(6):1292-1305.
[10] 李文涛,李文洁,刘淑清,等.猪圆环病毒2型TaqMan荧光定量PCR检测方法的建立[C]//中国畜牧兽医学会2010学术年会暨第二届中国兽医临床大会论文集.长春:中国畜牧兽医学会,2010:298-301.
LI W T,LI W J,LIU S Q,et al.Establishment of porcine circovirus type 2 TaqMan fluorescence quantitative PCR detection method[C]//Proceedings of Excellent paper of the 2nd China Veterinary Clinical Conference.Changchun:Chinese Animal Husbandry and Veterinary Association,2010:298-301. (in Chinese)
[11] WANG J C, ZHANG Y N, WANG J F, et al. Development of a TaqMan-based real-time PCR assay for the specific detection of porcine circovirus 3[J].JVirolMethods, 2017, 248:177-180.
[12] CHEN G H, TANG X Y, SUN Y, et al. Development of a SYBR green-based real-time quantitative PCR assay to detect PCV3 in pigs[J].JVirolMethods, 2018, 251:129-132.
[13] STRECK A F, HERGEMÖLLER F, RÜSTER D, et al. A TaqMan qPCR for quantitation ofUngulateprotoparvovirus1 validated in several matrices[J].JVirolMethods, 2015, 218:46-50.
[14] SOLTANY-REZAEE-RAD M, SEPEHRIZADEH Z, MOTTAGHI-DASTJERDI N, et al. Comparison of SYBR Green and TaqMan real-time PCR methods for quantitative detection of residual CHO host-cell DNA in biopharmaceuticals[J].Biologicals, 2015, 43(2):130-135.



