李浩东,闵祥玉,周 雅,张禾垟,郑军军,刘琳玲,王 平,王艳梅,杨福合*,王桂武*
(1.中国农业科学院特产研究所,长春 130112; 2.吉林省梅花鹿产业研究院,长春 130600)
我国不仅拥有悠久的梅花鹿饲养历史,还拥有最大的梅花鹿饲养群体,是梅花鹿遗传育种的主战场[1]。特别是在2020年,梅花鹿被列入《国家畜禽遗传资源品种目录》[2],使得养鹿迎来了大发展。其主产品鹿茸具有极高的医学和药用价值,明代李时珍在《本草纲目》上就有关于鹿茸的记载,称其“善于补肾壮阳,生精益血,补髓健骨”。而且鹿肉、鹿鞭等也能很好的契合健康的主题。但长期以来,梅花鹿的选育都以鹿茸为主要经济目的,重茸轻肉,而忽略了对肉用鹿品种的培育。鹿肉性温和,有补脾益气、温肾壮阳的功效,瘦肉多、结缔组织少,蛋白质含量丰富、脂肪少,具有极高的营养价值。目前,鹿肉在国内拥有广阔的市场,但肉用鹿尚无专门化的品种,在市场及饭店出售的鹿肉多为老弱病残的淘汰鹿,品质不佳,安全性难以保证[3]。
在传统育种中,畜禽的选配主要是利用表型以及经验来进行的,后过渡到以最佳线性无偏预测(best linear unbiased prediction,BLUP)为主通过评估畜禽育种值来进行选择的方法[4]。随着高通量测序技术的快速发展和测序成本降低使得畜禽育种已经进入分子育种阶段,分子标记辅助选择(marker assisted selection,MAS)的应用使畜禽遗传改良有了长足的进步。但由于经过功能验证用于畜禽遗传改良的功能基因与分子标记较少,且较多分子标记对遗传变异比例解释太低,限制了分子辅助标记的应用[5-6]。2001年,Meuwissen等[7]提出了基因组选择的概念,全基因组选择(genomic selection,GS)被作为是全基因组范围内的标记辅助选择,是当下畜禽遗传育种的主流策略[8],相较于基于系谱的估计育种值(estimated breeding value, EBV),利用覆盖全基因组的标记位点估计的基因组估计育种值(genomic estimated breeding value, GEBV)更加准确[9]。统计模型是基因组选择的核心,极大地影响了基因组预测的准确性和效率。根据统计模型的不同,可以分为以GBLUP为代表的直接法[10]和以贝叶斯为代表的间接法[11]。牛[12-13]、猪[14-15]、鸡[16-17]等动物育种相继进入基因组选择育种时代。尤其是在奶牛育种中,基因组选择已在美国[18-19]、澳大利亚[20-21]、英国[22-23]、新西兰[24-25]等国家广泛应用,并取得了明显的遗传进展。
目前,梅花鹿的选育工作还停留于传统的育种方法。由于梅花鹿尚未完全驯化,仍具有一定野性,导致生产性能测定难度极大,并且大多数养殖场系谱记录混乱甚至无系谱记录,而基因组选择的优势在于能够降低测定难度以及利用全基因组的标记位点构建亲缘关系替代系谱,提高育种值估计的准确性,缩短时代间隔。随着梅花鹿由小户散养向集约化养殖的转变,基因组选择是提高整体生产水平的最佳选择。因此,本试验基于梅花鹿的生长相关性状来评估各种模型预测的准确性,以探究在梅花鹿群体中实施基因组预测的可行性。
1 材料与方法
1.1 试验动物
本研究动物群体来自吉林省长春市双阳某梅花鹿繁育场2014—2019年所产纯种梅花鹿,共计261只。其中公鹿96只,母鹿165只。测定的生长性状包括体重(body weight)、体高(body height)、体斜长(body length)、管围(shin circumference)、胸围(chest girth)、臀端高(high hips)。利用Excel对表型数据进行预处理,删除表型的缺失值和异常值(平均值±3倍标准差)。通过R语言中的GLM函数校正性别和出生年份。梅花鹿表型描述性统计见表1。

表1 梅花鹿体重与体尺指标的表型统计量
1.2 基因型数据的获取与质量控制
采取颈静脉采血的方式抽取血液用于基因组DNA的提取,将样品送至博瑞迪生物技术有限公司,利用本团队开发的梅花鹿液相40K育种芯片[26]进行基因分型。芯片分型共得到了92 246个SNPs位点,使用PLINK(V1.90)软件[27]对分型结果进行质量控制,质量控制标准:1)个体SNP检出率>90%;2)SNP检出率>90%;3)最小等位基因频率≥5%;4)哈代温伯格平衡P<1×10-6。最终获得261个个体的84 059个SNPs位点用于后续分析。
1.3 基因组选择模型
使用ASReml4.1.0软件[28]通过约束最大似然法进行方差组分的估计与遗传力的计算。基于全基因组SNP信息构建亲缘关系G矩阵,通过加性遗传方差除以总方差计算性状遗传力。计算公式如下:

1.3.1 基因组最佳线性无偏预测(GBLUP) GBLUP通过SNP信息构建分子亲缘关系矩阵(G矩阵)替代基于系谱信息构建的关系矩阵(A矩阵),该方法由 Vanraden[29]提出。该方法的统计模型如下:
y=Xb+Zg+e

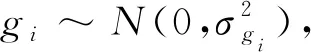
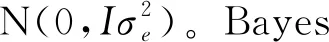
1.3.3 岭回归最佳线性无偏预测法(RRBLUP) RRBLUP是间接法的代表。Whittaker等[32]为了解决标记效应误差的问题,因而提出的一种改良的最小二乘法,类似于GBLUP法。其将标记效应作为随机效应,假定标记效应服从正态分布,利用混合线性模型求解标记效应的总和即为个体育种值。RRBLUP模型的计算使用R语言环境下的RRBLUP包进行[33]。
1.4 基因组预测准确性的评估
对以上6种方法通过5-fold交叉验证的方法来评价GEBV的估计准确性,先将梅花鹿群体随机分成5个独立的子集,然后依次将每个子集作为验证群体,并假定其表型未知,同时利用其余4个子集做参考群体来预测验证群体的GEBV。预测能力为表型与GEBV间的相关性。为避免遗传力对预测准确性的影响,将表型与GEBV间的皮尔逊相关系数除以相应性状遗传力算术平方根作为 GEBV 准确性的评价指标[34]。计算公式为:
其中,y表示校正后的表型值,h2表示性状遗传力。对每个性状的验证进行10次重复,最后用相关系数的平均值作为模型准确性的评估指标。
2 结 果
2.1 梅花鹿生长相关性状遗传参数的估计
利用全基因组SNP信息构建亲缘关系矩阵,建立混合线性模型,通过约束最大似然法对梅花鹿生长相关性状的加性遗传方差、环境方差、遗传力以及标准误进行估计。结果表明,梅花鹿群体重与体尺性状的遗传力范围为0.15(胸围)~0.50(臀端高)。其中,管围与臀端高的遗传力分别为0.43、0.50,属于高遗传力;体重、体高与体斜长的遗传力分别为0.22、0.30、0.27,属于中等遗传力,而胸围的遗传力为0.15,属于低遗传力。各性状方差组分和遗传力及标准误估计见表2。

表2 梅花鹿生长相关性状的方差组分和遗传力估计
2.2 梅花鹿生长相关性状基因组选择不同模型的预测准确性比较
通过5-fold交叉验证,利用GBLUP、Bayes A、Bayes B、Bayes C、Bayes Lasso、RRBLUP六种基因组选择模型对生长相关性状的预测准确性进行评估,将表型与GEBV间的皮尔逊相关系数除以相应性状遗传力算术平方根作为 GEBV 准确性的评价指标。
2.2.1 梅花鹿体重基因组选择预测准确度 如表3所示:体重的预测准确度分别是0.231、0.143、0.121、0.123、0.086、0.077。在体重中,GBLUP预测准确性最高,Bayes A、Bayes C、Bayes B和Bayes Lasso次之,RRBLUP最低。
2.2.2 梅花鹿体高基因组选择预测准确度 如表3所示:体高的预测准确度分别是0.205、0.324、0.339、0.329、0.310、0.336。在体高中,Bayes B预测准确性最高,RRBLUP、Bayes C、Bayes A和Bayes Lasso次之,GBLUP最低。
2.2.3 梅花鹿体斜长基因组选择预测准确度 结果如表3所示:体斜长的预测准确度分别是0.256、0.336、0.333、0.338、0.314、0.314。在体斜长中,Bayes C预测准确性最高,Bayes A、Bayes B、RRBLUP和Bayes Lasso次之, GBLUP最低。
2.2.4 梅花鹿管围基因组选择预测准确度 如表3所示:管围的预测准确度分别是0.197、0.222、0.230、0.222、0.223、0.217。在管围中,Bayes B预测准确性最高,Bayes Lasso、Bayes A、Bayes C和RRBLUP次之, GBLUP最低。
2.2.5 梅花鹿胸围基因组选择预测准确度 如表3所示:胸围的预测准确度分别是0.190、0.601、0.593、0.579、0.529、0.583。在胸围中,Bayes A预测准确性最高,Bayes B、RRBLUP、Bayes C和Bayes Lasso次之, GBLUP最低。
2.2.6 梅花鹿臀端高基因组选择预测准确度 如表3所示:臀端高的预测准确度分别是0.192、0.318、0.319、0.318、0.311、0.325。在臀端高中,RRBLUP预测准确性最高,Bayes B、Bayes A、Bayes C和Bayes Lasso次之,GBLUP最低。

表3 基于6种基因组选择模型对梅花鹿生长相关性状的预测能力与准确性
2.3 梅花鹿生长相关性状遗传力对基因组预测准确性的影响
如表3所示,预测能力的范围为从0.036(RRBLUP法中体重)到0.233(Bayes A法中胸围)。在GBLUP中,各性状的预测能力由高到低依次为臀端高、体斜长、管围、体高、体重、胸围;在Bayes A与Bayes B中,各性状的预测能力由高到低依次皆为胸围、臀端高、体高、体斜长、管围、体重;在Bayes C、Bayes LASSO与RRBLUP中,各性状的预测能力由高到低依次皆为臀端高、胸围、体高、体斜长、管围、体重。有大量研究表明,基因组选择的预测能力与性状的遗传力估计值呈正相关关系[35-37]。为了探索遗传力与预测能力之间的关系,对遗传力与预测能力进行回归分析。如图1所示,6种基因组选择模型的相应回归系数为0.144(GBLUP)、0.074(Bayes A)、0.101(Bayes B)、0.1(Bayes C)、0.153(Bayes Lasso)、0.132(RRBLUP)。
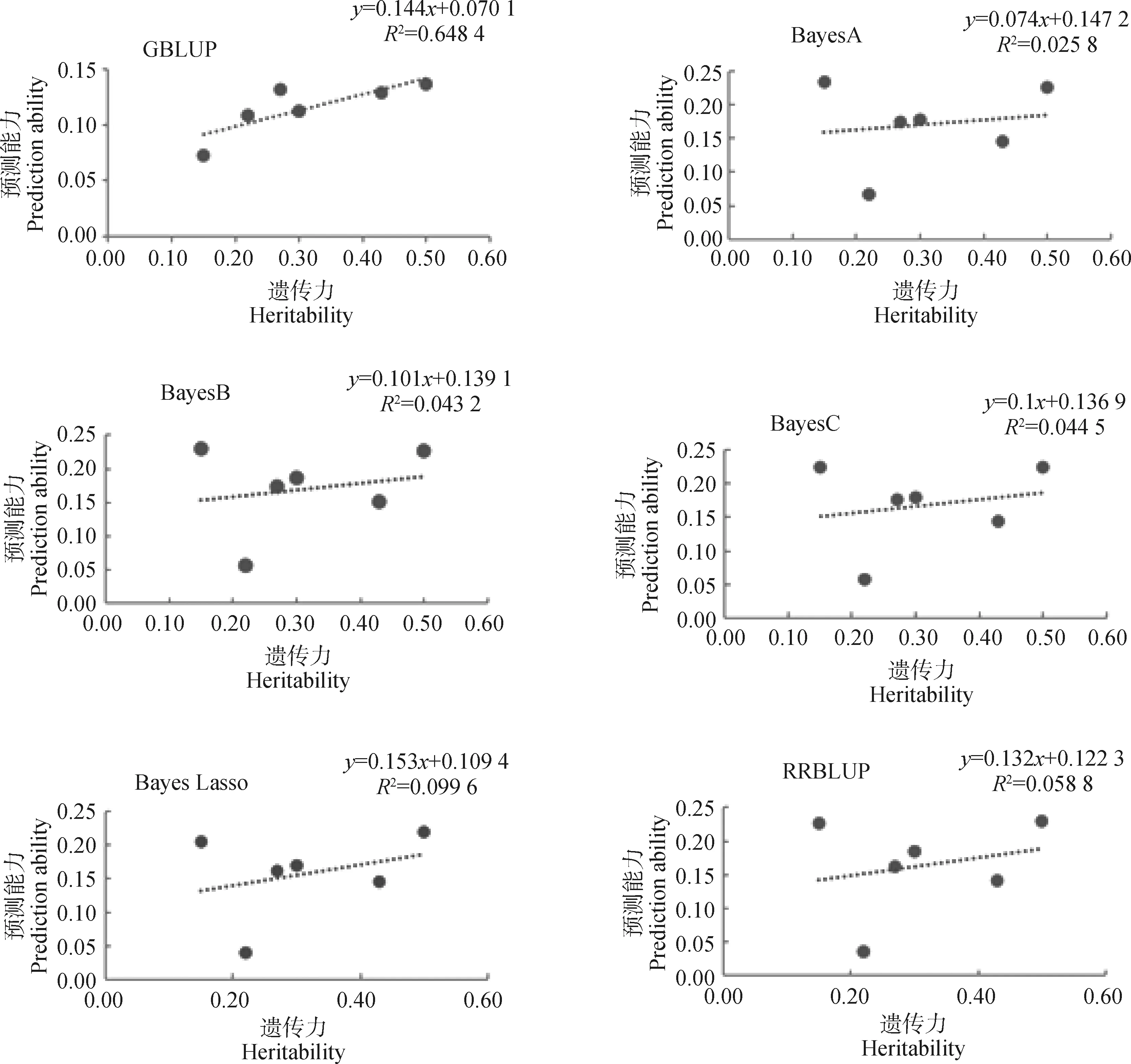
图1 6种基因组选择模型的预测能力与遗传力回归分析
3 讨 论
本研究利用基因组数据对梅花鹿体重体尺性状进行了遗传力估计,并系统的探讨了以GBLUP为代表的直接法模型和以Bayes类方法为代表的间接法对生长相关性状基因组选择的预测能力与预测准确性。利用基因组信息基于GBLUP通过约束最大似然法对方差组分进行估计,进而计算遗传力。遗传力的估计受动物群体种类、群体大小以及标记密度等因素的影响。但在本研究中,动物群体以及大小等因素是固定存在的,因此均已无法探讨,但已将性别与年龄作为固定效应加入遗传力的估计中。在本研究中,体尺性状的遗传力均为首次估计。其中,管围与臀端高属于高遗传力;体重、体高与体斜长属于中等遗传力,而胸围属于低遗传力。基于全基因组SNP信息构建的亲缘关系估计的梅花鹿体重遗传力远低于周世朗等[38]基于系谱构建的亲缘关系估计的遗传力,这可能有两方面原因:1)模型高估了显性遗传方差,因此低估了加性遗传方差,导致遗传力的估计偏低[39];2)全基因组SNP信息构建的亲缘关系矩阵相较于系谱构建的亲缘关系矩阵能够更加准确的反映个体间的遗传关系[40]。在基于系谱构建的亲缘关系中,共同的环境组分会导致高估加性遗传方差,使得遗传力的估计偏高[41]。因此,在对梅花鹿生长相关性状进行遗传评估时,利用基因组信息构建亲缘关系矩阵会更加准确。
本研究基于5-fold交叉验证对GBLUP、Bayes A、Bayes B、Bayes C、Bayes Lasso、RRBLUP六种基因组选择模型预测的准确度进行了比较,分析了不同模型对不同性状的适用性。由于交叉验证分组存在随机性,单次的交叉验证不具有代表性。验证过程中,重复的次数越多,预测的准确性更高。本研究对各个性状的验证进行10次5-fold交叉验证重复,用以校正评估的准确性。在体重中,GBLUP法的预测准确度均优于Bayes类与RRBLUP法。证明在样本量较少的情况下,GBLUP的预测准确度并非一定低于间接法[42];Bayes B在各个性状中的预测准确性都较高,可能是由于模型假设更加符合数量性状的遗传结构。它假设有效应位点比例为1-π,通常为0.05左右;Bayes A在体重、体斜长、胸围等低遗传力性状中预测准确度优于Bayes B。因而在低遗传力性状中,首选Bayes A进行预测是最佳选择;体重、体高、体斜长、管围、胸围、臀端高预测准确度最高的分别为GBLUP、Bayes B、Bayes C、Bayes B、Bayes A、RRBLUP。因此,在基因组选择中进行模型选择时并没有统一的标准,要根据性状的遗传结构选择最适合的模型才能使得预测更加精准。
本研究利用6种基因组选择模型探讨了对体重体尺性状的预测能力,并对遗传力与预测能力进行了回归分析。研究发现,性状遗传力的高低会影响基因组选择预测的准确性[25-27]。在GBLUP中,各个性状遗传力与预测准确性大致呈正相关关系,这与相关研究结果相一致。但在Bayes类方法与RRBLUP中,低遗传力的胸围却拥有较高的预测准确度,原因可能是梅花鹿的特殊性导致,尚未完全驯化,具有较大的野性,在鹿群进食过程中饲料营养不均衡,使得鹿只胸围的变异系数较大,导致在进行遗传力估计时高估了环境方差,导致遗传力的估计偏低。因此,在梅花鹿的实际生产过程中,要更加注意饲养管理,以达到鹿只的体况均匀。既能保障生产水平的提高,同时也能提高基因组选择预测的准确性。
4 结 论
本研究以梅花鹿作为研究群体,对梅花鹿体重体尺性状进行了遗传力估计,并基于5-fold交叉验证对GBLUP、Bayes A、Bayes B、Bayes C、Bayes Lasso、RRBLUP六种基因组选择模型进行了基因组选择分析。研究发现,体重、体高、体斜长、管围、胸围与臀端高的遗传力分别为0.22、0.30、0.27、0.43、0.15、0.50,且性状遗传力的高低对基因组预测准确性有重要影响。体重、体高、体斜长、管围、胸围、臀端高预测准确度最高的分别为GBLUP、Bayes B、Bayes C、Bayes B、Bayes A、RRBLUP。说明在实际生产中,没有能够完全适应所有性状的模型,必须根据预测的准确性以及预测的时效性来特异的选择最佳模型。本研究为梅花鹿生长相关性状的基因组选择与实际应用提供了重要的参考信息。



