吴祎程,冉 涛,周传社*,谭支良
(1.中国科学院亚热带农业生态研究所 亚热带农业生态过程重点实验室 畜禽养殖污染控制与资源化技术国家工程实验室 湖南省动物营养生理与代谢过程重点实验室 农业部中南动物营养与饲料科学观测实验站,长沙 410125;2.中国科学院大学,北京 100049;3.兰州大学草地农业科技学院,兰州 730020;4.兰州大学草种创新与草地农业生态系统全国重点实验室,兰州 730020)
反刍家畜瘤胃是一个复杂且多样的微生态系统,其内栖息了细菌、真菌、原虫、古生菌和病毒等多种微生物,瘤胃内病毒主要由噬菌体(bacteriophage)构成[1]。在微生物生态系统中,病毒尤其是噬菌体的侵染作用会极大地影响微生物群落的结构及功能[2]。但由于病毒基因组缺乏合适标记基因和分析基准,且多数病毒无法被归类或找到对应宿主,因此,难以通过PCR等常规技术来检测未知病毒序列[3],以致病毒组研究相对滞后。近年来,宏基因组学和生物信息学的发展极大地促进了宏病毒组的研究,并已应用于研究不同环境样本中的病毒群落,如海洋[4]、土壤[5]、人类粪便[6]等。研究人员很早便关注瘤胃液中的病毒群落,陆续分离鉴定了多种瘤胃噬菌体,包括以坏死梭杆菌(Fusobacteriumnecrophorum)、白色瘤胃球菌(Ruminococcusalbus)、溶纤维丁酸弧菌(Butyrivibriofibrisolvens)和瘤胃拟杆菌(Prevotellaruminicola)等为宿主菌的10种噬菌体[7]。但是,目前仍缺乏对瘤胃病毒群落的整体认知。
宏病毒组测序的关键在于从待测样品中富集病毒组分(virus-like particles,VLPs),获取高质量的病毒核酸用于宏基因组测序。已有研究发现,DNA病毒占据了病毒的绝大部分,本研究仅关注DNA病毒。由于病毒的基因组非常小,如果直接对样品中所有的微生物组DNA进行宏基因组测序,虽然能够获得部分病毒的基因序列,但是容易丢失那些读长短、丰度低的病毒序列,不能真实反映病毒的群体构成[8]。同时,病毒组分中的噬菌体具有溶源性、溶菌性和慢性感染等生命周期,其中溶源性噬菌体在溶源性周期中将遗传物质整合到其细菌宿主的基因组中[9],通过生物信息学分析可以从细菌宏基因组数据中挖掘到溶源性噬菌体的基因序列,但是不能有效获取仅有溶菌性周期的噬菌体。因此,VLPs的富集对全面解析样品中游离病毒组分尤为重要。由于瘤胃液样本具有特殊性,如存在饲料大颗粒、厌氧环境、中性偏酸环境,因此有必要建立专门的瘤胃病毒颗粒分离方法[10]。理想情况下,该方法应能快速、高效获得病毒组分,易于在实验室中操作。
氯化铯(CsCl)密度梯度离心是病毒浓缩和纯化的常用技术之一,但是该方法需要用到超速离心机,不仅价格昂贵,而且操作耗时较长,更重要的是可能导致大量病毒组分的损失[11]。氯仿处理被认为是一种可代替 CsCl密度梯度离心的快速纯化病毒的方法,其可通过破坏细菌的胞膜结构,以达到有效去除细菌的效果[12]。鉴于此,本研究在提取瘤胃液病毒组分基因组DNA之前,参考Castro-Mejía等[13]对人粪便样品采用的聚乙二醇(PEG 8000)沉淀法,设计4种流程瘤胃液病毒组分富集流程:(P1)离心+稀释、(P2)离心+过滤+核酸酶处理、(P3)离心+稀释+过滤+聚乙二醇沉淀、(P4)离心+稀释+过滤+聚乙二醇沉淀+氯仿处理。PEG 8000广泛应用于病毒(噬菌体)的纯化过程,可使病毒沉淀,而不受其他有机质的干扰。本研究旨在比较过滤、PEG沉淀以及氯仿处理对瘤胃液样品中游离病毒组分DNA质量(产量、纯度和完整性)的影响,并利用Illumina Novaseq 6000平台对获得的瘤胃病毒基因组DNA进行测序,通过对瘤胃病毒群体结构进行分析来评价不同流程对瘤胃病毒组分富集的效果,以期为进一步研究瘤胃病毒功能提供技术支撑。
1 材料与方法
所有动物程序均遵循动物护理和使用指南,并经中国科学院亚热带农业研究所动物护理委员会批准(批准号 ISA-W-201609)。选用3只体重相近((31.13±3.72)kg)、体况良好的成年雄性湘东黑山羊作为试验动物,安装永久性瘤胃瘘管,按本团队成员曾波等[14]的报道进行术后护理和饲喂。试验羊的基础日粮由52%玉米秸秆、21.52%玉米、16.96%麦麸、5.98%豆粕、0.86%菜籽饼、1.08%尿素、0.60%食盐以及1%预混料组成。日粮营养水平为10.72%粗蛋白、58.76%中性洗涤纤维以及27.79%酸性洗涤纤维。
1.1 瘤胃液样品采集步骤
山羊晨饲后4 h,通过瘤胃瘘管采集瘤胃内容物,用无菌搅拌棒搅动使瘤胃内容物样品混匀,经4层无菌纱布过滤至50 mL无菌离心管,离心管装至2/3处后立刻盖严,投入液氮罐中速冻,置于干冰上迅速转移至实验室,于-80 ℃冰箱中保存备用。
1.2 瘤胃液病毒组分制备
试验流程参考Castro-Mejía的方法[13],并加以修改,以有效分析瘤胃液病毒组。为消除动物个体差异,将3头黑山羊的瘤胃液等体积混合,分成3份,操作流程如图1所示。
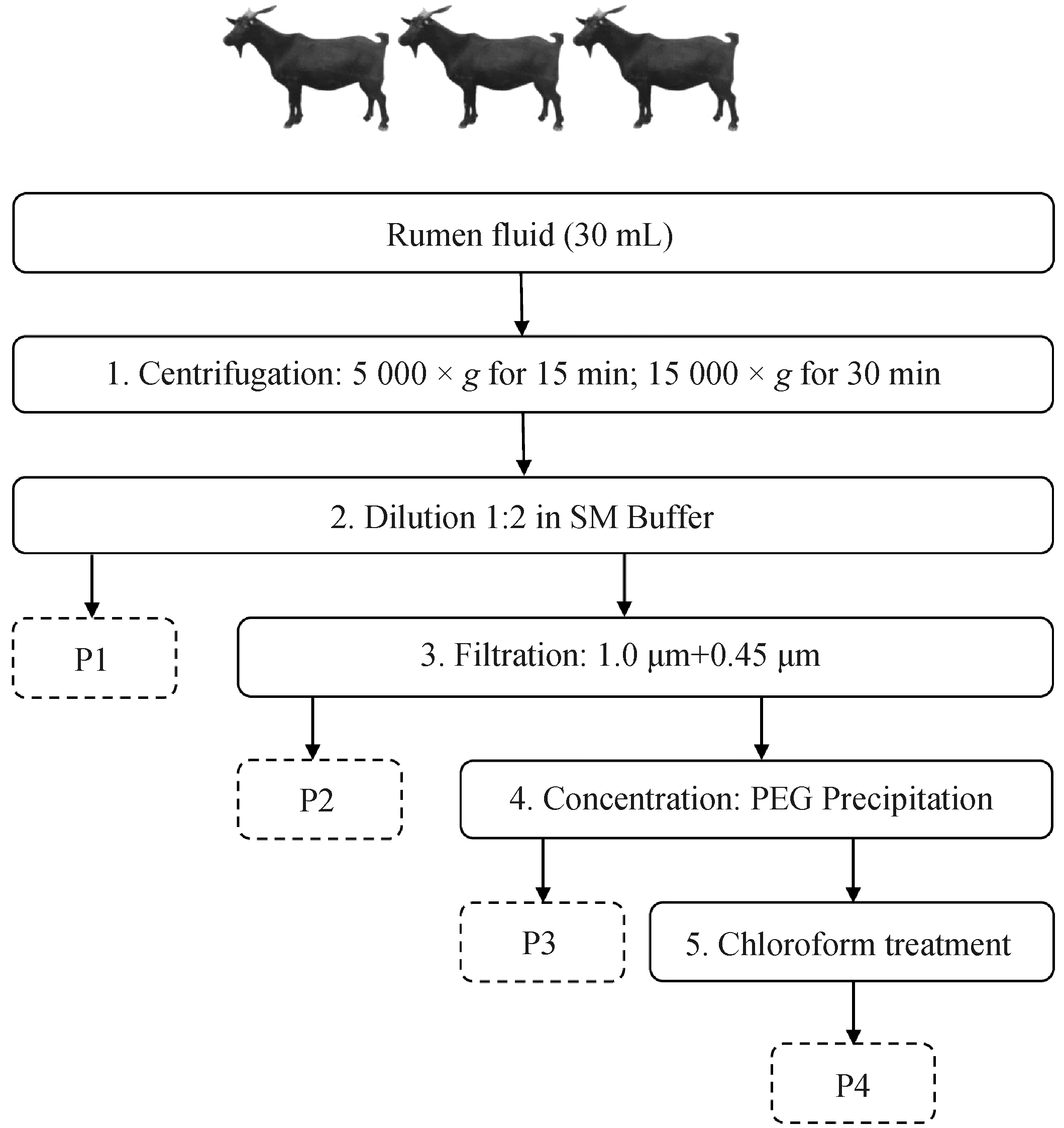
将瘤胃液混合后,分成三份按照四组流程进行处理,命名为P1至P4,下同。步骤1(离心)、2(稀释)为所有流程的共同步骤,且P1只执行步骤1和2,P2执行步骤1、2和3(微孔滤膜过滤),P3执行步骤1、2、3和4(聚乙二醇沉淀),P4执行步骤1、2、3、4和5(氯仿处理)The rumen fluid of three goats were collected and mixed, then divided into three parts and processed according to four routes named P1 to P4, the same as below. Steps 1 (centrifugation) and 2 (dilution) are the common steps of all processes, and P1 only performs steps 1 and 2, P2 performs steps 1, 2 and 3 (microporous membrane filtration), P3 performs steps 1, 2, 3 and 4 (PEG prrecipitation), and P4 performs steps 1, 2, 3, 4 and 5 (chloroform treatment)图1 从山羊瘤胃液中分离富集病毒组分的流程示意图Fig.1 Schematic representation of the procedures for the extraction of viruses from goat rumen fluid
离心:将解冻瘤胃液在4℃下以5 000×g离心15 min,将液体部分转移至新的无菌离心管中,以去除瘤胃液样品中的饲料残渣;接着在4℃下以15 000×g转速离心30 min,以去除其他小颗粒碎片,收集上清液。
稀释:将上述含有游离病毒样颗粒的上清液与预冷的SM缓冲液(200 mmol·L-1NaCl, 10 mmol·L-1MgSO4, 50 mmol·L-1Tris pH 7.5)进行 1∶2稀释。
过滤:通过1.0 μm的玻璃纤维滤纸(Whatman,GF/C,UK)过滤,以去除瘤胃液中的粒径较大的细菌、原虫等,随后经过孔径为0.45 μm的PVDF无菌滤膜(Millipore Billerica,MA,USA)过滤,进一步去除细菌等。
PEG沉淀:将200 g PEG8000与146.1 g NaCl溶于1 L蒸馏水中,经高压蒸汽灭菌后获得PEG-NaCl溶液。将过滤步骤后得到的上清液转移到新离心管中,向每个样品中加入25%(v/v)的PEG-NaCl溶液,于4 ℃下震荡孵育22 h,孵育结束后将样品于4 ℃、13 000×g离心45 min,弃上清,沉淀即为病毒组分,并将其溶解于1 mL预冷的0.01 mol·L-1磷酸盐缓冲液(PBS)中。
氯仿处理:向获得的病毒组分浓缩液中加入等体积的氯仿,涡旋1 min,使氯仿与样品均匀混合,形成乳状物,于4℃、15 000×g离心5 min,小心吸取上清液至新的无菌离心管,即为病毒组分。
1.3 瘤胃液基因组DNA提取
核酸酶处理:在提取DNA之前,用1.25 μL DNase I和1.25 μL RNase A处理样品30 min,以消除游离DNA和RNA污染,随后在37 ℃水浴中孵育10 min,接着加入1 μL 100 mmol·L-1EDTA使核酸酶失活,最后在65 ℃下水浴孵育10 min终止EDTA反应。使用DNA提取试剂盒(Omega D3892-01Viral DNA Kit, USA)提取制备的瘤胃液病毒组分样品的核酸,提取步骤严格按照试剂盒说明书进行。完成病毒基因组DNA抽提后,使用Qubit®DNA广谱分析仪(ThermoFisher Scientific)测定浓度、OD260 nm/280 nm及OD260 nm/230 nm,并使用1.0%(w/v)琼脂糖凝胶电泳检测基因组DNA的完整性。提取的DNA样品储存在-20 ℃,直至文库制备及测序。检测合格的DNA样品采用Covaris M220超声破碎仪将样品DNA随机打断成长度约为450 bp的片段,经末端修复、加入A尾、加测序接头、纯化、PCR全基因组扩增等步骤完成文库制备,质检合格的文库将采用Illumina Novaseq 6000平台进行测序(上海凌恩生物技术有限公司)。
1.4 数据质控
由于Illumina Novaseq 6000的原始测序数据中包含测序接头序列、低质量读段、N率较高序列及长度过短序列,这将严重影响后续组装的质量。为保证后续的生物信息分析的准确性,首先对下机的原始测序数据进行质控,过滤掉低质量片段和宿主片段从而得到高质量的测序数据(clean data),以保证后续分析的顺利。原始数据已递交GenBank,登录号为PRJNA788346。
1.5 生物信息学分析
按照Roux等[15]描述的方法,使用Megahit(https://github.com/voutcn/megahit)对高质量序列进行拼接、组装,挑选≥ 2 000 bp的组装序列,使用如下软件进行病毒序列鉴定:VirFinder(https://github.com/jessieren/VirFinder)、VirSorter2 (https://github.com/simroux/VirSorter)、CAT(https://github.com/dutilh/CAT),并对初步鉴定为病毒的序列进行vOTUs(viral operational taxonomic units)分析。鉴定得到病毒序列后,获得其物种分类学信息,并使用VPF-Class工具对病毒进行物种注释和宿主预测。挑选置信度(Confidence_Score)> 0.36的结果,进行病毒科水平物种注释;挑选置信度(Confidence_Score)> 0.5且成员比对率(Membership_Ratio)超过0.3的结果,进行病毒(噬菌体)潜在宿主预测[16]。
1.6 数据分析
利用Microsoft Excel(Microsoft Inc., Washington, USA)对数据进行初步统计,随后用SPSS 19.0(SPSS Inc., Chicago,USA)对不同流程提取的基因组DNA质量相关指标 进行单因素方差分析(ANOVA)[17]。对宏基因组数据进行正态检验和方差齐性检验,符合方差分析的数据用SPSS进行ANOVA分析,不符合方差分析的数据用SPSS软件进行非参数分析,P<0.01表示差异极显着, 0.01≤P<0.05表示差异显着,0.05 ≤P<0.10表示存在趋势。瘤胃液病毒相对丰度图、箱线图分别利用凌恩生物在线云平台(http://cloud.biomicroclass.com/CloudPlatform)和联川生物在线云平台(https://www.omicstudio.cn/tool)的工具在线绘制。
2 结 果
2.1 不同提取流程对瘤胃病毒核酸提取质量的影响
各流程提取的基因组DNA质检结果见图2。由图2a可知,P2流程中DNA产量显着高于P1、P3和P4处理流程(P<0.001)。P1、P2与P3流程获得的DNA的OD260 nm/280 nm均> 1.90,说明提取的核酸相对纯净(图2b)。经氯仿处理(P4)的基因组核酸DNA,因产量过低(图2a)而不满足高通量测序需求,故没有进行后续上机建库及组学分析。
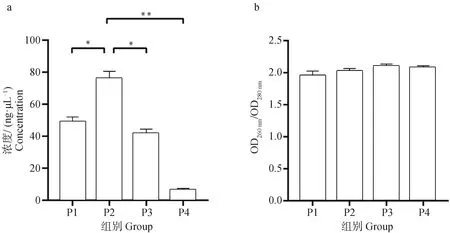
a. 核酸浓度;b. 核酸纯度。*表示P<0.05,**表示P<0.01a. Concentration of nucleic acids; b. Quality of nucleic acids. * P<0.05 and ** P<0.01图2 四种流程处理后所提取的山羊瘤胃液病毒组分的核酸质量(n=3)Fig.2 Quality of nucleic acids of ruminal viral-like particles extracted from the goat rumen fluid in four groups (n=3)
2.2 不同提取流程对瘤胃病毒群落α多样性的影响
使用Illumina Novaseq 6000平台对符合测序要求的9个样本进行了病毒宏基因组学测序,共获得1 058 773 724个reads,每个样品的平均reads数为117 641 525,范围为105 495 627至125 358 829。不同提取流程的瘤胃病毒α多样性指数见图3,不同提取流程对测序时获得的goods_coverage比例没有显着影响(图3a),同时不同流程间Chao 1指数(P=0.834)和Shannon指数(P=0.255)均无显着差异。
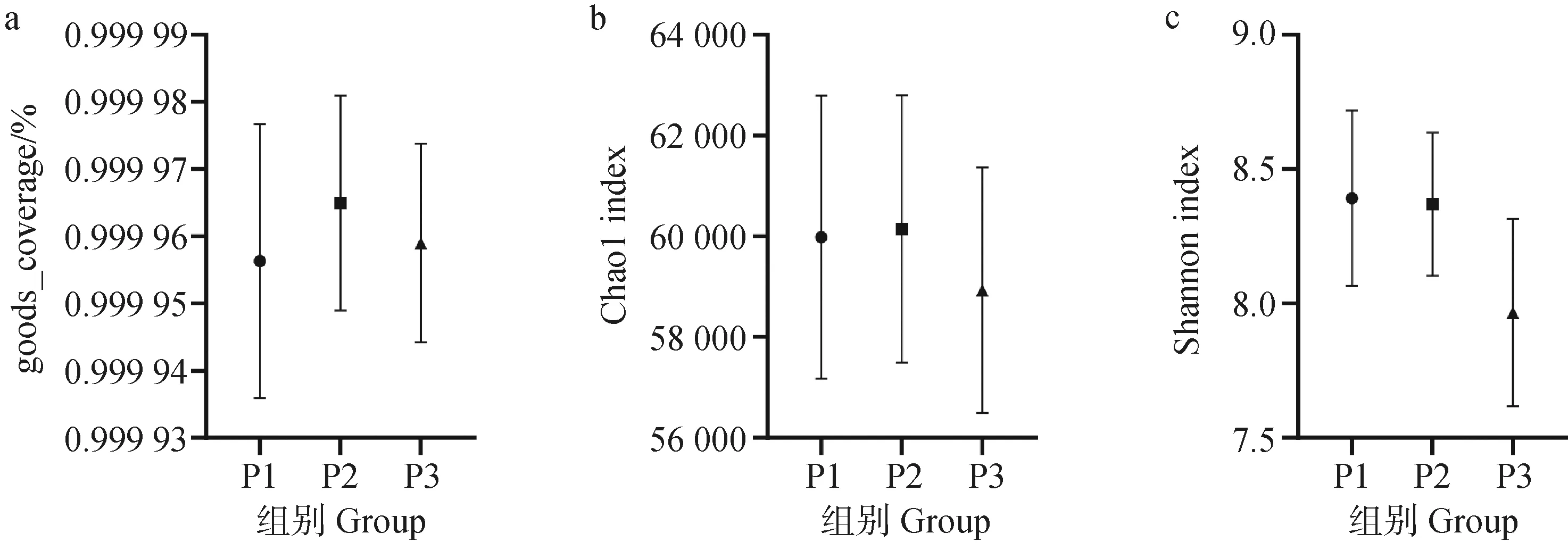
a. goods_ coverage; b. Chao 1指数,用于估计群落中vOTU丰度;c. Shannon指数,用来描述群落中病毒多样性a. goods_ coverage; b. Chao 1 index, represents the richness of vOTU; c. Shannon index, represents the diversity of virus图3 不同处理流程获得的瘤胃液病毒的α多样性指数Fig.3 Alpha diversity indexes of ruminal virome obtained from different processing procedure
2.3 不同提取流程对瘤胃病毒群落β多样性的影响
为评估不同样品处理流程对瘤胃病毒群落组成的影响,进行了基于Bray-Curtis距离的PCoA分析及ANOSIM分析。在PCoA图中,P2流程与P1和P3在PCoA2上分开(26.01),但差异不显着(ANOSIM R=0.695,P>0.05;图4)。结果表明,虽然不同DNA提取流程会导致病毒群落组成的差异,但对群落组成的影响较小。
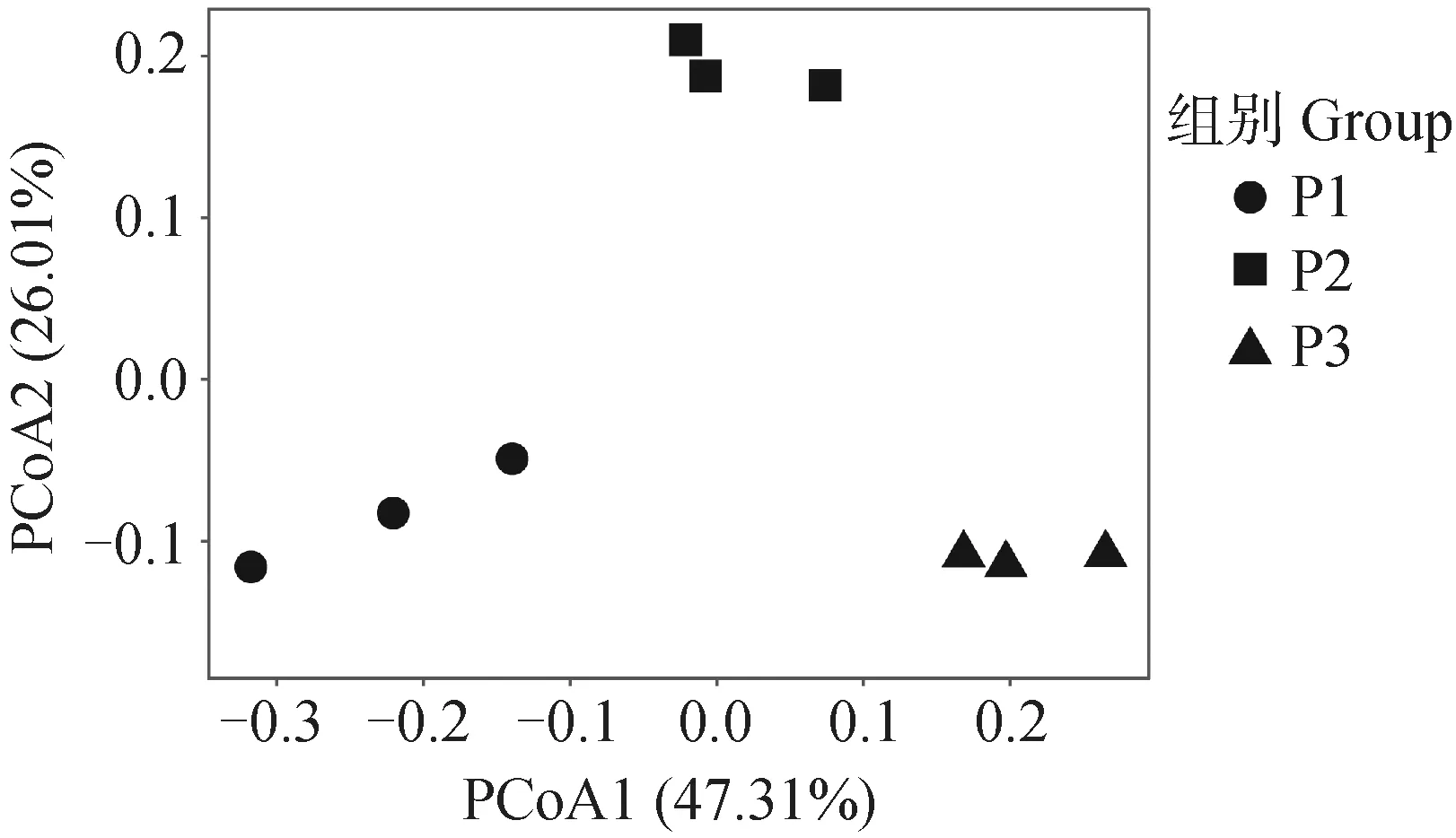
图4 不同处理流程对瘤胃病毒组分的主坐标分析(基于Bray-Curtis距离,n=3)Fig.4 Principal co-ordinates analysis based on Bray-Curtis distances of ruminal virome obtained from different processing procedure (n=3)
2.4 不同提取流程对瘤胃病毒群落分类学特征的影响
宏基因组测序结果表明,不论采用何种瘤胃液样品病毒组分富集流程,均显示山羊瘤胃中的DNA病毒主要为噬菌体(图5)。其中,绝大多数噬菌体序列来自长尾病毒科(Siphoviridae,63.29%)、肌尾病毒科(Myoviridae,11.87%)和短尾病毒科(Podoviridae,5.64%),这3个病毒科同属于有尾病毒目(Caudovirales)。进一步分析了不同富集流程对瘤胃病毒相对丰度的影响,结果表明PEG处理对Siphoviridae(P<0.05)及Ackermannviridae科(P<0.01)的病毒有显着富集效果(图6)。
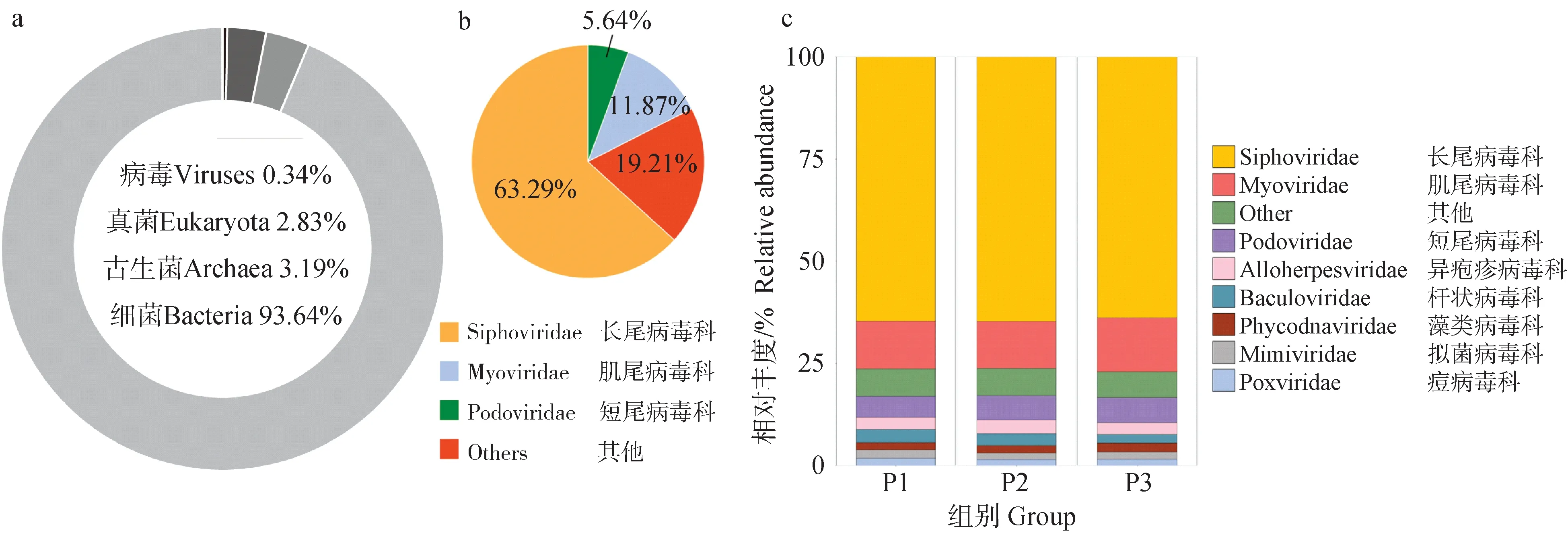
a. 总体瘤胃微生物组成;b. 总体病毒组分组成;c. 不同处理流程瘤胃液中病毒相对丰度(科水平)a. Overall composition of rumen microbial; b. Overall composition virus-particles; c. Relative abundance of viruses in different processing procedures (family level)图5 黑山羊瘤胃液中微生物组成Fig.5 Composition of rumen microbial of black goat
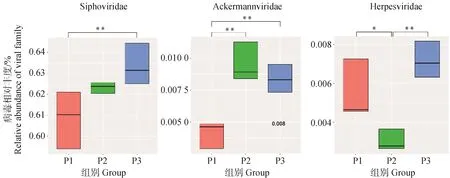
*表示P<0.05,**表示P<0.01* means P<0.05 and ** means P<0.01图6 在不同流程中病毒相对丰度(n=3)Fig.6 Relative abundance of viral family in different processing procedures (n=3)
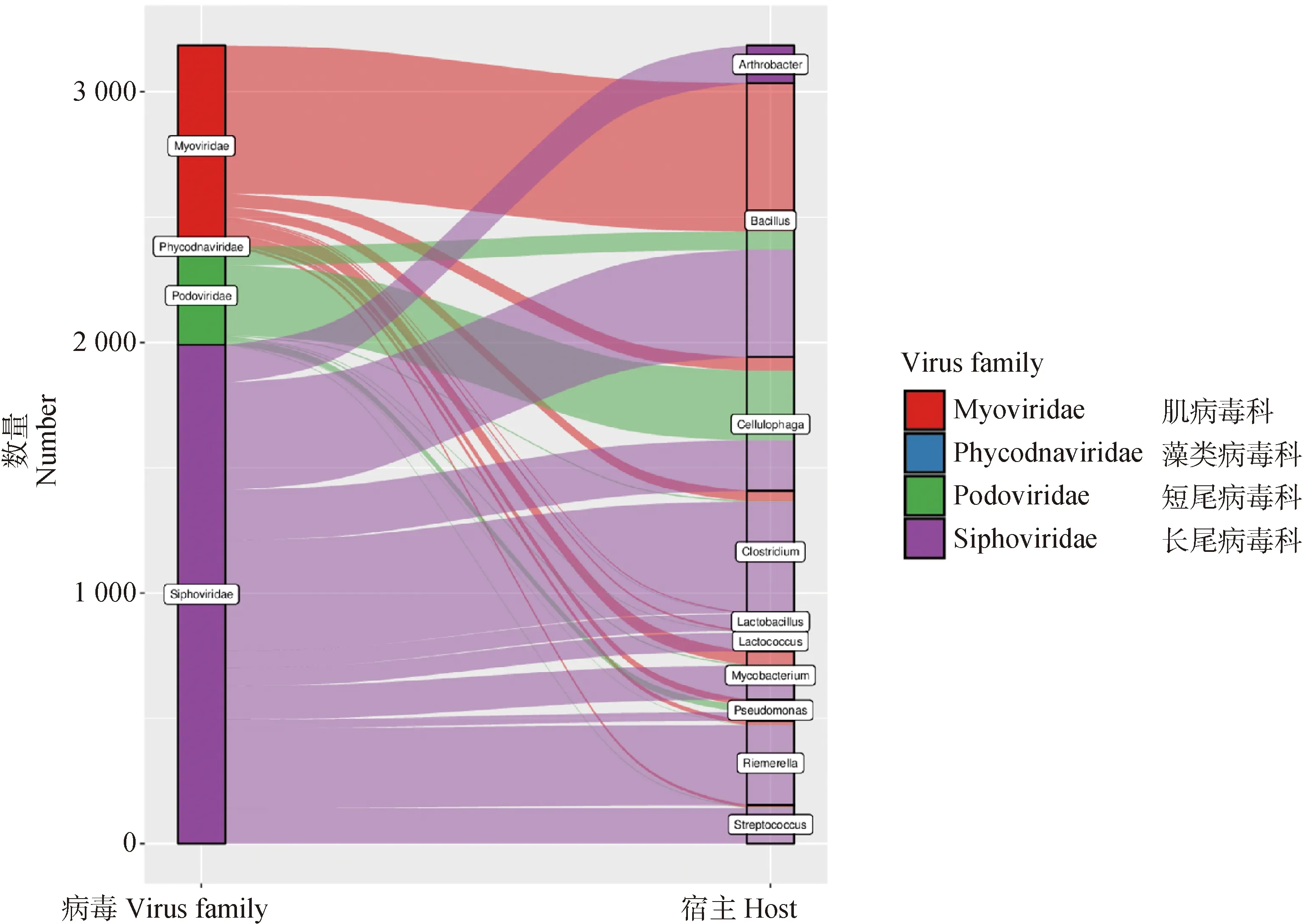
图7 山羊瘤胃病毒群落的潜在宿主种类预测Fig.7 Prediction of potential hosts of ruminal viruses in rumen fluid of goats
2.5 瘤胃病毒群落宿主预测
利用VPF-Class工具对山羊瘤胃中最丰富的4个DNA病毒科噬菌体成员进行了宿主预测,并列出了噬菌体与宿主间的对应关系(图6)。结果显示瘤胃病毒的宿主多为细菌,优势宿主预测排序结果为:节杆菌属(Arthrobacter)、芽孢杆菌属(Bacillus)、噬纤维素属(Cellulophaga)、梭菌属(Clostridium)、乳酸球菌属(Lactococcus)、分岐杆菌属(Mycobacterium)、绿脓杆菌属(Pseudomonas)、里氏杆菌属(Riemerella)、链球菌属(Streptococcus)。其中,Myoviridae科的噬菌体主要侵染芽孢杆菌属(Bacillus)细菌,Podoviridae科的噬菌体主要侵染噬纤维素属(Cellulophaga)的细菌,而Siphoviridae科的噬菌体主要侵染芽孢杆菌属(Bacillus)、梭菌属(Clostridium)、里氏杆菌属(Riemerella)和噬纤维素属(Cellulophaga)等属的细菌。
2.6 不同生境病毒群落比较
本研究将山羊瘤胃病毒群落与其他环境,如人类粪便[4]、海洋[5]或土壤[6]病毒群落进行比较(图8)。结果表明,不同生境中病毒群落构成存在很大差异:在生境病毒群落多样性方面,以土壤生境最高;在病毒群落相对丰度方面,人类粪便和山羊瘤胃病毒群落的相似性更高,均为以Siphoviridae科的病毒为主,但二者又有区别,体现在山羊瘤胃病毒群落具有更高丰度的Myoviridae科病毒;在病毒核酸类型方面,人类粪便和山羊瘤胃中的DNA病毒以双链DNA(double-strain DNA,dsDNA)病毒为主,而土壤生境中还含有微病毒科(Microviridae)、球状病毒科(Globuloviridae)、矮化病毒科(Nanoviridae)等单链DNA(single-strain DNA,ss-DNA)病毒。
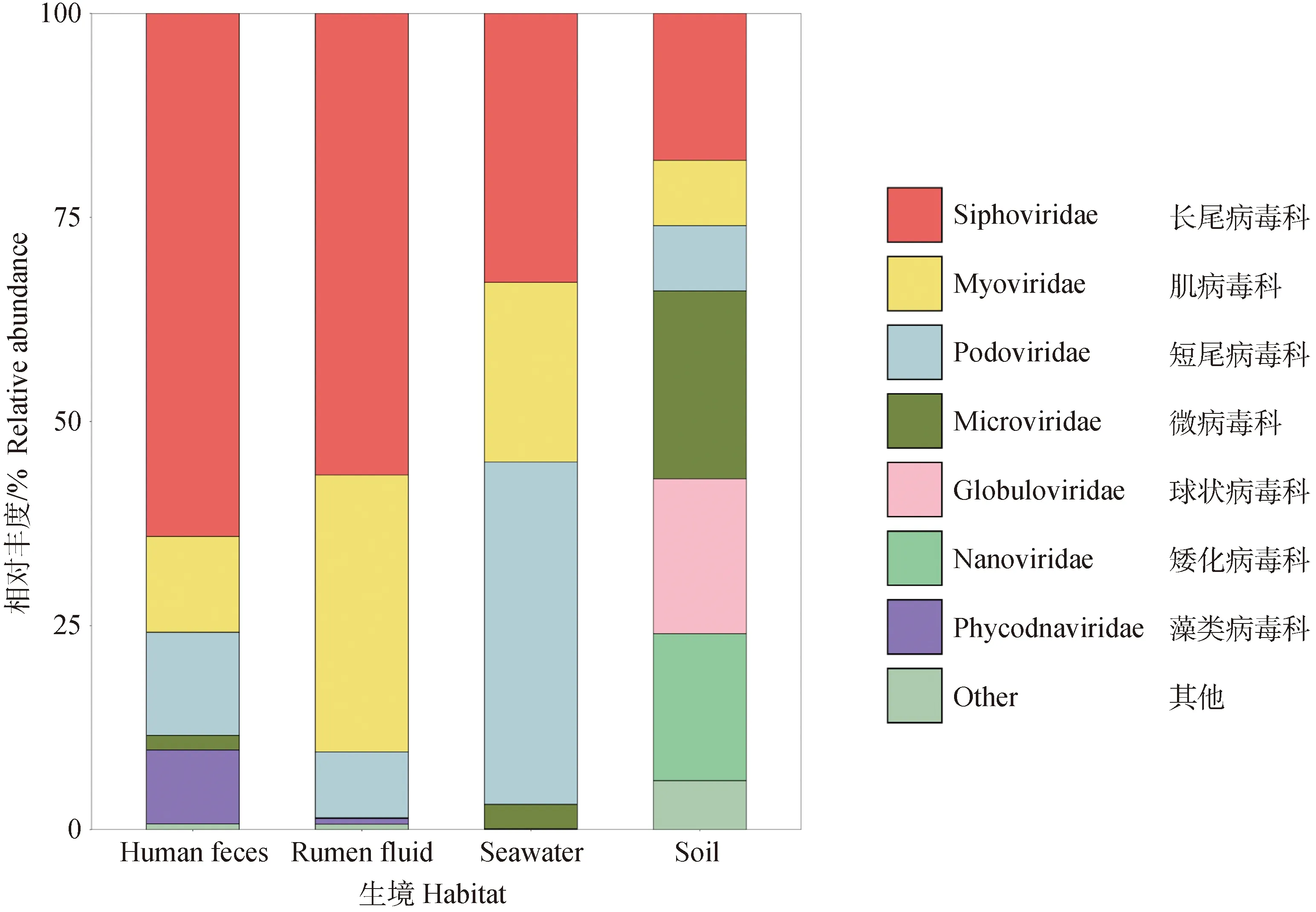
图8 山羊瘤胃液、人类粪便[4]、海水[5]及土壤[6]中的病毒相对丰度Fig.8 Relative abundance of viral communities in goat rumen fluid, human faeces[4], seawater[5] and soil[6]
3 讨 论
瘤胃微生物组成十分复杂,绝大多数瘤胃细菌是严格厌氧或兼性厌氧,且目前仅有很少的瘤胃细菌被分离培养,这导致基于传统培养手段的瘤胃病毒(主要是噬菌体)研究开展起来困难重重。近年来宏基因组学广泛运用于动物胃肠道微生态的研究,可规避体外培养微生物的限制。该技术也逐步应用于病毒组的研究,借助各种生物信息学分析工具,极大地推进了病毒特异性序列数据集(病毒组)的获取和分析[18]。高通量测序过程中,样品DNA的质量尤为关键,提高瘤胃病毒组分基因组DNA的产量及质量,可以有效地提高后续生物信息学分析的准确性。在核酸提取和上机测序之前,病毒组分预处理的主要作用为病毒富集和病毒纯化[11]。具体步骤需要根据所研究的样本类型进行调整。本研究参考Castro-Mejía等[13]提出的方法,设置了4种流程来评估过滤步骤、PEG沉淀以及氯仿处理对DNA提取质量以及宏基因组测序结果的影响。
OD260 nm/280 nm是衡量DNA提取质量的重要指标,会受RNA污染以及DNA降解的影响[19]。P3流程所获得的核酸产量并不是最高的,但P3流程OD260 nm/280 nm在数值上最高,P4流程所获得的核酸产量最低。病毒学研究过程中常使用传统的苯酚-氯仿提取法对病毒基因组进行提取[20],而如今市场上已经出现了多款用于从环境样品中提取病毒基因组DNA的试剂盒[9,21]。本研究选用了价格为Qiagen试剂盒1/5的Omega病毒DNA提取试剂盒,该试剂盒原用于提取血清病毒基因组,本研究表明,该试剂盒也能用于提取瘤胃液样品中的病毒基因组,且满足测序需求。
不同流程间瘤胃病毒多样性并没有显着差异,这可能是由于病毒组在整体微生物组中所占比例过小,正如Hess等[22]报道的,即使在大多数环境中噬菌体的数量级为细菌的10倍,但由于宿主DNA(细菌和真核生物)的污染,病毒reads只占可注释DNA的2%~5%。
病毒群落结构分析表明,本试验选用的山羊瘤胃中的优势病毒群落依次为长尾病毒科(Siphoviridae)、肌尾病毒科(Myoviridae)和短尾病毒科(Podoviridae),这与前人发表的瘤胃病毒群落以长尾病毒科、肌尾病毒科和短尾病毒科占主导地位一致[9]。过滤步骤需考虑滤膜孔径大小,常用微孔滤膜孔径为0.22或0.45 μm。先前研究表明胃肠道中最丰富的长尾病毒科(Siphoviridae)及肌尾病毒科(Myoviridae)都大于0.22 μm[9],故本研究选用了孔径为0.45 μm的微孔滤膜。本研究中丰度最高的为长尾病毒科(Siphoviridae),也证明了0.45 μm滤膜的有效性。由于病毒颗粒小,在核酸提取之前,通常使用PEG配合NaCl来沉淀样品中的VLPs以实现病毒的浓缩。本研究表明,PEG可显着富集有尾病毒目中的长尾病毒科(Siphoviridae)及Ackermannviridae科噬菌体。本研究还发现疱疹病毒科(Herpesviridae)的病毒在该瘤胃液样本中的存在,但丰度十分低。
CsCl密度梯度离心步骤被认为是纯化病毒的最佳方法[12],此法可对特定密度范围内的噬菌体进行纯化,并根据密度将VLPs与其他组分分离。但这一技术耗时、昂贵且需要特定的技术技能和实验室设备。有研究发现,氯仿处理能代替氯化铯密度梯度离心步骤用于纯化病毒[23],此方法已成功用于血清病毒纯化和发酵食品病毒纯化[24-25]。但本研究发现,氯仿处理在瘤胃液病毒纯化中会造成大量颗粒损失,以至于不能满足高通量测序的需求。这是因为瘤胃细菌仍为瘤胃液的主要部分,氯仿会严重干扰包膜类物质,比如细菌,而且有些病毒也存在膜结构,膜结构被破坏后,病毒的蛋白质外壳会变得不稳定[26]。而部分病毒蛋白质外壳也对氯仿十分敏感,氯仿处理则造成这类病毒的大量丢失[27]。DNA产量也因此出现显着差异,P4提取流程的核酸因产量过低,不适用于高通量测序建库。因此,氯仿处理代替CsCl密度梯度离心步骤用于纯化瘤胃病毒还有待后续试验进一步研究。
环境中病毒的多样性和丰度与微生物群落息息相关。有研究表明同一环境中病毒群落具有很高的相似性,而来自不同环境的病毒及其宿主分布的相似性较低[28-30]。反刍动物会从外界摄入大量食物,瘤胃作为一个天然发酵罐,微生物群落共生于一个相对封闭的容器中,这导致瘤胃微生物具有非常独特的群落特征[1],瘤胃液中病毒的丰富度反映了瘤胃微生物宿主的多样性。由于病毒宏基因组学建库限制以及测序平台对核酸样品扩增的需求,本研究主只检测了山羊瘤胃液中的DNA病毒,发现样品中主要为dsDNA病毒,它们都属于有尾病毒目。之前的大量研究也表明瘤胃病毒以dsDNA为主,其中大部分为有尾病毒目的噬菌体[9,21,31-32]。另外,由于技术的局限性,本方法提取的DNA不一定满足长读长测序平台,例如PacBio(www.pacb.com)or Oxford Nanopore(https://nanoporetech.com)。此外,本试验只关注了病毒组分中dsDNA的核酸,此病毒组分富集方法是否适用于ssDNA或RNA病毒尚不清楚[33]。
4 结 论
本研究比较了不同DNA提取流程对核酸产量、质量、病毒多样性以及病毒概况的影响,表明了PEG沉淀步骤对长尾病毒科及Ackermannviridae科噬菌体具有富集作用。研究人员可根据试验需求进行选择,当需要重点关注这类噬菌体的情况下,可采用P3流程,即对瘤胃液进行离心、稀释以及微孔滤膜过滤操作,若对特定病毒没有富集需求,则采用P2流程即可,即无需进行微孔滤膜过滤步骤,此两种流程均能达到病毒宏基因组测序要求。基于以上结果,本试验构建了提取瘤胃液中病毒组分DNA的流程,此方案可大大促进反刍动物瘤胃液中噬菌体群落的研究进程。



