李鹏飞, 龚真莉,杜晓华,蒋 韬*
(1.甘肃农业大学 动物医学院,兰州 730070;2.中国农业科学院 兰州兽医研究所 家畜疫病病原生物学国家重点实验室国家口蹄疫参考实验室,兰州 730046)
口蹄疫(foot-and-mouth disease,FMD)分布于世界各地,是一种具有高度传染性的病毒性疾病[1-2],因其具有重大的经济影响,被认为是影响畜牧业发展最重要的疫病之一。口蹄疫病毒(foot-and-mouth disease virus,FMDV)是引起FMD的病原体,属于小RNA病毒科,口蹄疫病毒属,由O、A、C、SAT1、SAT2、SAT3和Asia1 7种血清型组成[3]。FMD完整的病毒粒子为正二十面体对称结构, 由VP1、VP2、VP3、VP44种结构蛋白各60个拷贝组成, RNA分子被包裹在内[4]。VP1~VP3 3种结构蛋白暴露在病毒颗粒表面且聚集成蛋白质三聚体复合物,较小的结构蛋白VP4隐藏于内部[5]。VP1蛋白是FMDV颗粒表面的主要结构蛋白,在其易变的 G-H 环上不仅含有病毒主要的抗原位点,而且含有一个高度保守的RGD基序(精氨酸-甘氨酸-天门冬氨酸),该基序是病毒粒子与细胞表面整联蛋白结合引起感染的主要结合位点。尽管RGD依赖的整联蛋白受体通过RGD基序识别配体,但FMDV-VP1蛋白G-H环中VP1侧面的氨基酸残基在FMDV感染中同样起重要作用。如RGD依赖性整联蛋白αvβ5和α5β1并未充当FMDV的受体[6]。VP1 S154D突变导致Asia1 型FMDV使用猪整联蛋白受体αvβ6和αvβ8的能力增加,从而增加了其在宿主细胞中的复制水平,增强了对宿主猪的致病性[7]。
整联蛋白是哺乳动物细胞黏附和信号传导的关键蛋白之一[8]。迄今为止,已鉴定18种不同的α亚基和8种β亚基非共价结合构成24种整联蛋白异二聚体跨膜糖蛋白受体,可与多种细胞或细胞外配体结合,通常整联蛋白不具有活性,在活化后才能成为黏附剂[9]。整联蛋白可以通过结合细胞外配体或通过细胞内结构域与细胞骨架相互作用,在细胞膜上双向传递信号[10]。在非活性状态下,整联蛋白的胞外结构域不与配体结合,以弯曲构象形式存在。然而,来自细胞的信号引起构象变化,暴露外部配体结合位点,结合配体并将信号从细胞外传递到细胞内[11]。在体外,至少有4种整联蛋白(αvβ1、αvβ3、αvβ6和αvβ8)可作为FMDV受体来介导FMDV感染宿主细胞[12]。研究发现,整联蛋白αvβ6可能是决定FMDV 组织嗜性的主要受体,且FMDV可能会激发整联蛋白αvβ6在上皮细胞中的表达,然后以网格蛋白依赖的方式内化。表明整联蛋白αvβ6不仅是FMDV的吸附受体,而且在病毒与受体结合后的脱壳、复制等过程中也发挥着重要作用[6]。研究证实,可溶性截短形式的牛整联蛋白αvβ6通过二价阳离子依赖的方式结合FMDV-RGD,可用作FMDV的捕获试剂,并不影响病毒的结合[13]。除整联蛋白外,在细胞培养中,FMDV可以迅速适应利用替代细胞受体: 硫酸乙酰肝素(HS)[14]和尚未确认的替代受体[15]。研究证实,整联蛋白α 和 β 亚基 N-末端形成的球形区域为胞外配体结合域[16]。
表面等离子体共振(surface plasmon resonance,SPR)技术是一种基于光学的实时监测方法,且因SPR技术无需标记、灵敏度高、特异性高、实时和快速等优势被广泛应用于各种生物分子相互作用的研究[17]。目前,尚未见国内外采用SPR技术研究FMDV RGD基序与猪源整联蛋白αvβ6胞外区结构域、αv亚基胞外区结构域和β6亚基胞外区结构域的结合动力学方面的报道。本研究在国内首次利用基于SPR技术的Biacore 3000系统,研究了FMDV RGD基序与猪源整联蛋白αvβ6胞外区结构域、αv亚基胞外区结构域和β6亚基胞外区结构域的相互作用,研究结果将为进一步阐明FMDV与宿主细胞整联蛋白受体的相互作用机制提供依据。
1 材料与方法
1.1 材料和仪器
猪源整联蛋白αvβ6胞外区、αv亚基胞外区和β6亚基胞外区由本实验室原核表达并纯化、鉴定得到。RGD(Arg-Gly-Asp)由南京金斯瑞生物科技有限公司合成。Biacore 3000生物传感器;Biacore 3000控制软件;氨基偶联试剂盒;CM5 Sensor Chip(CM5传感器芯片);不同 pH (4.0、4.5、5.0 和 5.5)的10 mmol·L-1醋酸钠缓冲液;HBS-EP 缓冲液;再生试剂盒均购自GE Healthcare公司。
1.2 方法
1.2.1 芯片表面预处理 开机后,首先将系统温度设置为25 ℃,取出Maintaince 芯片,插入 CM5 芯片并固定,使用primer命令用纯水冲洗管道,再换HBS-EP缓冲液,再次使用primer命令冲洗管道,最后通过HBS-EP缓冲液,直到基线稳定,为下一步的试验做好准备。
1.2.2 偶联pH的选择 分别使用氨基偶联试剂盒中pH4.0、4.5、5.0、5.5的10 mmol·L-1醋酸钠缓冲液稀释目的蛋白样品整联蛋白αvβ6胞外区、αv亚基胞外区和β6亚基胞外区至20 μg·mL-1,使其带上不同量的电荷。再将整联蛋白样品以10 μL·min-1的流速流过芯片表面,注射2 min,通过整联蛋白样品与芯片的结合曲线确定合适的偶联pH。最后,以20 μL·min-1流速注射30 μL 50 mmol·L-1NaOH洗脱液完全去除芯片表面吸附的样品蛋白。
1.2.3 在CM5芯片上偶联整联蛋白 使用预处理筛选得到的最合适的pH偶联缓冲液稀释目的蛋白至20 μg·mL-1,应用蛋白固定自动化程序进行氨基偶联。
1.2.4 结合试验 将RGD基序溶解在HBS-EP运行缓冲液中,并稀释至低浓度(31.25 μg·mL-1)和高浓度(500 μg·mL-1),以10 μL·min-1的流速进样3 min,观察响应值,看是否有结合。每次进样结束后,用Glycin-HCl(pH 3.0)再生30 s。
1.2.5 动力学测定 首先,将RGD样品在HBS-EP缓冲液中稀释至500、250、125、62.5、31.25 μg·mL-1的系列浓度。然后,以最大稀释量开始,以最小稀释量结束,分别在整联蛋白固定表面和参考表面以30 μL·min-1的流速注射稀释的RGD样品3 min。最后,停止注射RGD样品,观察其在5 min内的解离率,在此期间芯片上只通过HBS-EP缓冲液。每个浓度的测定设置2个循环,在每个结合循环后,以30 μL·min-1的注射速度注入Glycin-HCl(pH 3.0),对芯片表面进行再生。
1.2.6 数据分析 将所得的实时SPR传感图导入Biaevaluation 分析软件,选择1∶1 Lamgmuir 结合模型进行局部拟合。
2 结 果
2.1 芯片表面预浓缩-偶联pH的选择
通过Biacore 3000生物传感器信号响应值的变化,根据预浓缩得到的结果选择最合适的偶联pH。结果表明,整联蛋白αvβ6胞外区、αv亚基胞外区和β6亚基胞外区均在pH4.0的偶联缓冲液中与芯片表面产生最佳的静电相互作用。
2.2 在CM5芯片上偶联整联蛋白
通过蛋白固定自动化氨基偶联程序,最终得到样品蛋白的实际偶联量:整联蛋白αvβ6胞外区为13 333.3 RU、αv亚基胞外区为15 000.8 RU、β6亚基胞外区为10 000.6 RU。
2.3 结合试验与动力学测定
结合试验RU值表明RGD溶液与整联蛋白αvβ6胞外区和αv亚基胞外区有结合,且在低浓度下结合量少,在高浓度下结合量多,而RGD与β6亚基胞外区在两种浓度下均基本没有结合。由动力学曲线RU值可知,随着反应质量浓度的增加,结合信号也逐渐升高,说明结合量增大,且都表现为快结合快解离(图1)。采用 1∶1 Lamgmuir 结合模型拟合曲线,得到RGD基序与猪源整联蛋白αvβ6胞外区结构域和αv亚基胞外区的结合动力学常数(表1)。由平衡解离常数可知,RGD基序与整联蛋白αvβ6胞外区结构域的亲和力比与整联蛋白αv亚基胞外区结构域的亲和力高。
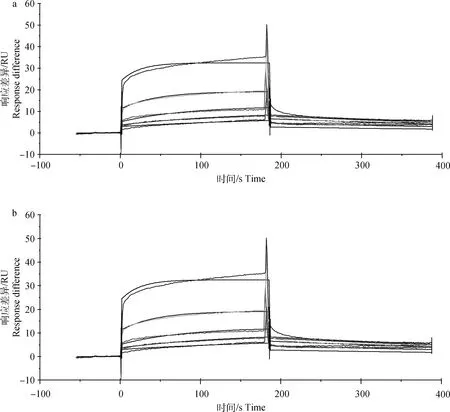
a.整联蛋白αvβ6胞外区;b.整联蛋白αv亚基胞外区a. αvβ6 extracellular domain; b. αv subunit extracellular domain图1 RGD基序与整联蛋白αvβ6胞外区和整联蛋白αv亚基胞外区的结合动力学曲线Fig.1 kinetic curves of RGD motif binding to the αvβ6 extracellular domain and αv subunit extracellular domain

表1 RGD基序与整联蛋白αvβ6胞外区和整联蛋白αv亚基胞外区相互作用的动力学常数Table 1 Kinetic constants of the interaction between the RGD motif and the αvβ6 extracellular domain and αv subunit extracellular domain
3 讨 论
FMDV通过细胞受体介导的内吞作用进入宿主,这一过程是由病毒与细胞表面受体(主要是整联蛋白)的结合启动的[18]。研究证实,在体外病毒通过结构蛋白VP1上的RGD基序与细胞表面整联蛋白结合并诱导内化[19]。另外,研究发现,形成FMDV病毒颗粒的衣壳蛋白中氨基酸残基的变化不仅会影响病毒与细胞受体之间的吸附能力,还可能影响病毒的生物学特性[7,20-22]。
本研究结果表明,RGD基序与整联蛋白β6亚基胞外区结构域之间几乎没有结合;RGD基序与整联蛋白αvβ6胞外区结构域的亲和力最强,平衡解离常数KD为7.33×10-6M;与整联蛋白αv亚基胞外区结构域之间的亲和力小于与整联蛋白αvβ6胞外区结构域的亲和力,平衡解离常数KD为9.79×10-5M。结合常数表明RGD基序与整联蛋白αvβ6胞外区结构域的结合速度快于与αv亚基胞外区结构域的结合速度。同时,动力学曲线表明,RGD基序与整联蛋白αvβ6胞外区结构域和整联蛋白αv亚基胞外区结构域的结合过程均为快结合快解离,有部分原因可能是由于本试验使用的RGD基序缺少上下游氨基酸,同时在体外整联蛋白构象改变,从而导致结合不稳定。
分子模拟FMDV RGD结构域的氨基酸与整联蛋白氨基酸之间可能的相互作用,发现RGD结构域的精氨酸(Arg143)与家牛α-整联蛋白的保守结构域(SIPLQ)的亮氨酸(Leu679)相互作用,除此之外其他氨基酸之间也存在相互作用,如FMDV与β-整联蛋白之间的其他相互作用包括Asn47-Gln1710、Arg152-Asn1643、Gly202-Gln1187和 Ser154-Lys1669;对不同牛种α-整联蛋白外显子区体外扩增的研究表明,嗜性位点(SIPLQ)具有保守性[23]。在本研究中并未得到RGD基序与整联蛋白β6亚基胞外区结构域之间的结合能力,可能是由于本研究中使用的是单一的Arg-Gly-Asp3肽,并没有RGD上下游的氨基酸,因此,并没有体现出与上述研究报道相似的结果。有研究表明,FMDV VP1衣壳蛋白中GH环的RGD序列可与细胞整联蛋白形成极其稳定的复合物,并且该复合物对EDTA具有抗性,特定氨基酸的替换表明,与αvβ6结合的稳定性取决于靠近RGD序列的螺旋结构,RGD +1和RGD +4位点的两个亮氨酸残基是这种稳定相互作用的关键位置[24]。
此外,有研究报道,整联蛋白对RGD基序的特异性识别是通过α亚基β螺旋桨结构域与精氨酸结合,天冬氨酸通过Mg2+或Mn2+在金属离子结合位点(MIDAS)与整联蛋白β亚基β1结构域结合[25-26]。本研究的样品中并没有加入Mg2+或Mn2+等金属离子。研究证实,整联蛋白β亚基βI结构域含有多个二价金属阳离子结合位点(MIDAS、ADMIDAS、LIMBS),配体与整联蛋白的结合受到二价金属阳离子的调节,二价金属阳离子(Ca2+、Mg2+和Mn2+)不仅是整联蛋白与配体结合的前提,而且还可以影响整联蛋白的激活状态[17,27],同时,二价金属阳离子对配体与αv整联蛋白上RGD位点结合的影响在同一配体的整联蛋白和同一整联蛋白的配体之间变化[28]。本研究结果将为进一步阐明FMDV与宿主细胞整联蛋白受体的相互作用机制提供依据。
4 结 论
利用SPR技术从动力学角度实时分析了RGD基序分别与猪源整联蛋白αvβ6胞外区结构域、αv亚基胞外区结构域和β6亚基胞外区结构域的相互作用,通过数据拟合分析得到特异性相互作用的动力学常数,结果表明,SPR技术能够实时、快速地检测相互作用过程。



