张 萍,庄林林,张 笛,董永毅,盛中伟,王成明,徐 步,窦新红,龚建森*
(1.中国农业科学院家禽研究所,扬州 225125;2.江苏农林职业技术学院畜牧兽医学院,句容 212400;3.江苏省动物疫病预防控制中心,南京 210036;4.奥本大学兽医学院,奥本36849)
沙门菌(Salmonella)是一种常见的食源性致病菌,常寄居在人和动物体内,特别是家禽、家畜的肠道中。沙门菌主要通过污染食品进而导致人类食物中毒,因而在公共卫生方面具有重要意义[1]。全世界因沙门菌感染引起的食物中毒病例常年高居首位[2],其中非伤寒沙门菌感染约导致15.5万人死亡,9 380万人患病[3],中国每年的发病人数也超过300万[4]。2015年,世界卫生组织对全球食源性疾病的调查报告显示,沙门菌发病数在22种细菌、病毒和原虫疾病中排名第一[5]。
沙门菌属包含肠道沙门菌(Salmonellaenterica)和邦戈尔沙门菌(Salmonellabongori)2个种,其中肠道沙门菌种又包含6个亚种[6]。此外,不同抗原型(菌体O抗原、鞭毛H抗原和荚膜Vi抗原)的沙门菌根据疾病类型(如鸡白痢沙门菌、肠炎沙门菌、鼠伤寒沙门菌等)或首次发现地点(如海德尔堡沙门菌、印第安纳沙门菌、肯塔基沙门菌等)命名为特定的血清型(其中具有相同O抗原的血清型隶属于同一血清群),目前报道的血清型种类已超过2 600种[7-8],沙门菌分类的复杂性无疑给检测、分型、溯源等工作增加了难度。传统沙门菌检测方法包括增菌培养、选择分离、生化鉴定和血清学分型等,通常耗时5~7 d,不利于及时发现和控制病原的流行[9]。1992年,Rahn等[10]首次根据鼠伤寒沙门菌invA基因设计引物并建立PCR检测方法,开创了沙门菌分子检测的先河。此后,随着分子生物学的快速发展及基因组数据的不断丰富,基于沙门菌特异性核酸靶点的分子检测方法的研究报道日益增多,目前可从沙门菌属、种、血清群、血清型等多个层面分别进行检测[11]。
本文旨在对近三十年来国内外报道的各类沙门菌分子检测方法(包括聚合酶链式反应、荧光定量PCR、多重PCR和环介导等温扩增技术等)进行总结,并从属、种、血清群、血清型等不同层面进行梳理归类,以期为沙门菌分子检测方法的广泛应用提供参考。
1 沙门菌分子检测的研究背景
1.1 传统检测方法存在的问题
沙门菌的传统检测采用选择性培养结合生化分析的方法,该方法要求样品中细菌浓度需达到20~200 CFU·mL-1以上,灵敏度偏低[12];大多数样品还需进行增菌培养才能成功分离,操作繁琐,周期较长;部分特殊样品(如受高温、高盐、冷冻等处理)易出现“活的非可培养状态”,难以通过传统方法检测;此外目前商品化的沙门菌选择性培养基种类繁多,但每种培养基均可能存在难以培养的沙门菌血清型或菌株,因而至少需要同时使用2种以上培养基来提高分离效率,增加了检测的难度和费用。
1.2 沙门菌血清学分型存在的问题
沙门菌血清学分型结果可以为流行病学调查、病原溯源等提供重要依据。沙门菌血清学分型依赖特异性强的分型血清进行凝集试验,依据Kauffmann-White血清分型表可鉴定得到不同菌株的血清型[13-14]。但是该方法操作较为繁琐复杂,效率较低,对分型血清质量要求也较高,如果血清特异性不强极易产生交叉反应,进而造成错误分型[15],而且血清品种不全也会影响到分型的顺利开展;此外,一些特殊状态的细菌需处理后再进行鉴定,如产生自凝的菌株,O抗原(H抗原)表达较弱的菌株等[16-17]。
1.3 沙门菌分子检测方法的特点
基于沙门菌特异性核酸靶点建立的分子检测方法(如常规PCR、荧光定量PCR、多重PCR、环介导等温扩增等)已在病原快速检测方面发挥重要作用。常规PCR技术根据沙门菌属(型)等特异性靶点设计引物进行体外扩增,灵敏度高、特异性强,简单快速[11]。荧光定量PCR技术可在扩增的每个循环中标记和监测PCR 产物,其检测灵敏度和特异性优于常规PCR[18]。多重PCR技术通过在同一反应体系中加入多对特异性引物,可实现对沙门菌的高通量检测与快速分型,进一步节省时间和成本[11,19]。环介导等温扩增技术在恒定温度条件下实现核酸高效扩增,相对于PCR技术,操作简单,对设备要求低,耗时更短[20]。
2 沙门菌分子检测方法的研究进展
2.1 沙门菌属分子检测方法
目前报道了很多沙门菌属的特异性分子检测方法(表1),此类方法的检测靶点主要涉及沙门菌属特有的毒力基因或看家基因,下面详细介绍。
bcfD:沙门菌属的一种保守菌毛基因,存在于已知所有血清型中,包括肠道沙门菌种和邦戈尔沙门菌种[60-61]。2014年,Zhuang等[24]针对bcfD设计引物建立了沙门菌属LAMP检测方法,对44株沙门菌的检测结果均为阳性,而非沙门菌则全为阴性。
fim操纵子基因:fim基因簇编码沙门菌I型菌毛,包含fimA等9个基因[62],目前,已有fimA、fimW和fimY报道作为沙门菌属检测靶点。fimA编码菌毛主要亚单位蛋白,但沙门菌、大肠杆菌和克雷伯氏菌fimA基因具有同源性,1996年,Cohen等[26]对fimA基因序列进行研究,选取其中高特异性片段设计引物并建立PCR方法,能够准确区分376株沙门菌和34株非沙门菌,从而确定了fimA作为沙门菌属特异性靶点的应用功能。fimW和fimY都是调控基因,2014年,Zhang等[27]建立的fimW-PCR能够将受试的68株沙门菌与12株非沙门菌进行区分;2002年,Yeh等[28]建立了fimY-PCR检测方法,结果表明仅45株沙门菌可以扩增出特异性条带,而20株非沙门菌不能扩增。
gyrB:是一种编码细菌DNA回转酶β亚基的看家基因,比16S rDNA具有更强的物种区分能力[63]。2011年,Ye等[30]通过分析gyrB基因序列,得到沙门菌特异性片段,经PCR检测所有被检沙门菌均可扩增得到366 bp的特异性产物。
hilA:编码侵袭基因正调节蛋白,位于沙门菌致病性岛1(SPI1),在沙门菌感染的过程中起着重要作用,在其它革兰阴性菌中不存在[33]。2000年,Guo等[32]基于hilA设计了两对PCR引物,其中HILA2可用于沙门菌特异性检测。
hns:编码DNA结合蛋白,与肠杆菌科其它属细菌的基因序列差异较大,仅与大肠杆菌具有一定的同源性。1993年,Maciorowski等[37]在对沙门菌hns基因进行分析的基础上,成功建立了特异性PCR检测方法,对所有受试沙门菌的检测均为阳性。此外还有其它多个研究也使用该基因做为检测靶点[64-65]。
invA:编码吸附和侵袭上皮细胞表面蛋白,1992年,Rahn等[10]首次以invA基因为靶点建立了沙门菌PCR检测方法,通过对630株沙门菌和142株非沙门菌进行检测发现99.4%的沙门菌能够检测出特异性条带,而非沙门菌则无法扩增出相应的条带或仅能扩增出非特异性条带,证明了invA广泛存在各个血清型的沙门菌中,可作为沙门菌属的候选检测靶点。此后该基因一直被广泛应用于各种沙门菌分子检测方法[38-39]。2018年,Yang等[29]对沙门菌LAMP检测方法的统计表明,invA基因占比高达74%。
ompC:编码外膜蛋白,是沙门菌及其它革兰阴性菌主要的结构蛋白。1996年,Kwang等[43]根据ompC序列设计引物建立PCR检测方法,能够成功区分60株沙门菌和42株非沙门菌。
oriC:是DNA的复制起点,基于该基因建立的PCR检测方法,可以将沙门菌与大肠杆菌等其它微生物进行选择性区分,已广泛用于沙门菌属检测[47,66]。Fluit等[46]利用沙门菌B群特异性单克隆抗体和基于oriC的特异性PCR引物,建立磁性免疫聚合酶链反应(MIPA),在大肠杆菌和鼠伤寒沙门菌浓度分别107和101CFU的情况下,仍可检测出样品中鼠伤寒沙门菌。
phoP:是磷酸化调节子的一部分,调节沙门菌的毒力和巨噬细胞存活相关基因的表达[49]。Li等[50]对115株肠杆菌科的phoP序列进行分析,发现沙门菌属phoP基因序列具有特异性,进而建立了phoP-LAMP检测方法,可准确区分66株沙门菌和73株非沙门菌。
rRNA靶点:包括16S rRNA 和16~23S rRNA靶点等。由于不同种的真细菌与古细菌间的16S rRNA基因的高度保守性,因而16S rRNA常被用于细菌的分类或种属鉴定[67]。Lin和TSen[22,68]通过分析沙门菌16S rRNA基因保守序列,设计多条探针,其中以16S Ⅲ和16SF1为引物对,建立了能特异性检测沙门菌的PCR方法。16S rRNA与23S rRNA的内部转录间隔区(internal transcribed spacer,ITS)也是一种rRNA,且不同细菌ITS长度和组成不同,因而也可以作为种属鉴定的靶序列。2005年,Chiu等[21]通过比较沙门菌和非沙门菌ITS序列,成功设计引物建立PCR方法,并对40株沙门菌和48株非沙门菌进行准确区分。
ttrRSBCA:该基因簇包含5个基因,位于沙门菌毒力岛2(SPI2)附近,ttrA、ttrB和ttrC编码四硫代还原酶结构蛋白,ttrS和ttrR编码双组分调节系统的传感器和响应调节器组件。分子杂交结果表明,该操纵子存在于所有肠道沙门菌种和邦戈尔沙门菌种的菌株。2004年,Malorny等[56]首次以ttrRSBCA为靶点建立实时PCR方法,成功区分了110株沙门菌和87株非沙门菌。
此外,还有一些沙门菌属分子检测方法,涉及的靶点如组氨酸转运操纵子基因hisJ[36]、inv基因簇侵袭相关基因invE[40]、插入元件IS200[42]、质粒毒力相关基因spv[54]和肠毒素基因stn[55]等。
2.2 沙门菌种分子检测方法
沙门菌属包括肠道沙门菌种和邦戈尔沙门菌种[6],目前只有关于肠道沙门菌种分子检测方法的报道。iroB是铁调节蛋白基因,1997年Bäumler等[69]通过Southern杂交发现,iroB存在于肠道沙门菌种的所有亚种中,但在邦戈尔沙门菌种和其它肠杆菌科细菌中均不存在。针对iroB建立的一种肠道沙门菌PCR检测方法,应用于197株细菌的检测,仅肠道沙门菌出现阳性条带。
2.3 沙门菌血清群分子检测方法
沙门菌根据O抗原的差异可分为46个血清群[1],其中以A、B、C、D和E群最为常见,而目前报道的分子检测方法也主要用于鉴定以上常见血清群。沙门菌血清群检测靶点主要针对参与合成O抗原的编码基因,这些基因通常聚集在rfb基因簇上。
rfb基因簇:包括rfb、wzx和wzy等。rfb编码糖基合成酶和转移酶,用于合成和组装O抗原。1993年Luk等[70]根据rfbJ(B)、rfbJ(C2)和rfbS(D)设计3对PCR引物,在总共123个测试的临床分离物中准确鉴定了属于血清群B、C2和D(A)群的所有40株沙门菌。但该方法不能鉴定血清群C1,且由于血清群A和D的靶基因仅有一个核苷酸碱基不同,同源性极高,难以区分。wzx(rfbX)编码一种具有12个跨膜片段的膜蛋白,负责将O抗原单位从细胞质转移至细胞周质中[71-72]。2007年Herrera-Len等[73]利用wzx-mPCR成功区分了沙门菌血清群B、C1、C2、D和E的分离株。wzy编码O抗原聚合酶,负责将细胞周质中的O抗原单位聚合在周质表面。2007年Fitzgerald等[7]将沙门菌的rfb基因簇中的abe、wzx、wzy等基因作为检测靶点,可以区分血清群A(D)、B、C1、C2、E和O13的菌株。
IS200/IS1351:IS200和IS1351属于沙门菌插入基因,研究表明沙门菌D(O9)群菌株与其它血清群相比,其序列中包含一段IS200/IS1351联合序列,因此可将该联合序列作为D(O9)群的检测靶点。2008年Okamura等[74]以IS200/IS1351联合序列为靶点,成功建立了特异性检测D(O9)群的LMAP方法。
2.4 沙门菌血清型分子检测方法
以肠炎沙门菌、鼠伤寒沙门菌等为代表的沙门菌血清型是目前流行最广泛的病原,能够感染人和各类动物,引起各种沙门菌病,造成严重的经济损失,具有重要的公共卫生学意义。这里将主要介绍此类流行性较广泛的血清型[75-100](表2)。
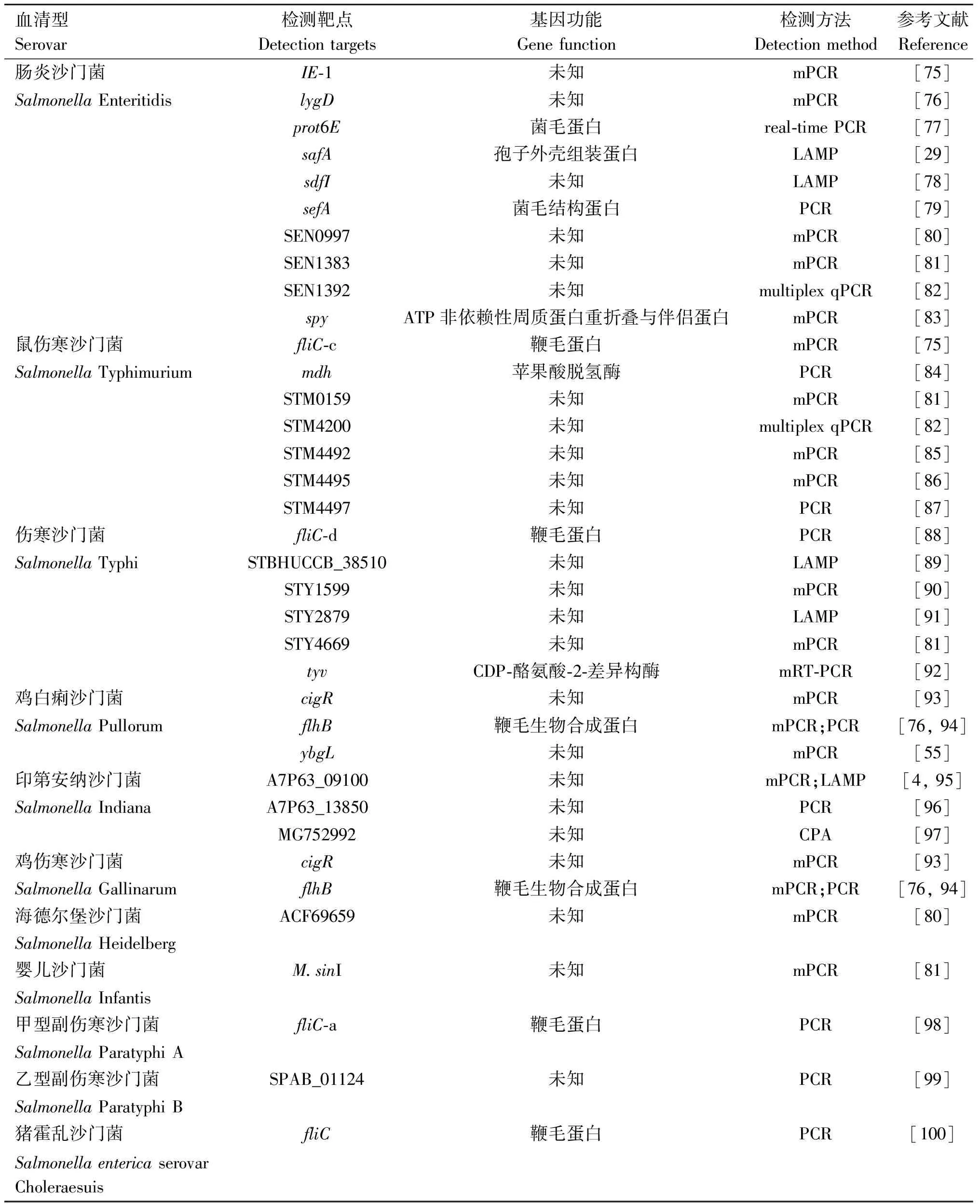
表2 沙门菌血清型分子检测方法
2.4.1 肠炎沙门菌 肠炎沙门菌的相关研究较多,主要靶点如下。
prot6E:属于沙门菌菌毛蛋白编码基因。2006年Clavijo等[101]研究肠炎沙门菌在蛋清中存活相关基因时发现两个肠炎沙门菌特异性基因:prot6E和SEN4287。Cornelia和Reiner[77]以prot6E为靶点建立的实时PCR,能够准确鉴定95%(75/79)的肠炎沙门菌,而其它54个血清型(119株)非肠炎沙门菌均没有检出。
sdfI与lygD:sdfI编码沙门菌差异性基因片段I,常作为靶点用于肠炎沙门菌分子检测方法的建立[78],但该基因与都柏林沙门菌同源性较高,需要进一步截取高特异性片段作为检测靶点。lygD是sdfI基因片段中一段高特异性序列,仅存在于肠炎沙门菌,所有非肠炎沙门菌血清型均不含有该基因,Xiong等[76]利用上述基因成功建立了区分不同D群血清型的多重PCR方法。
sefA:编码SEF14菌毛主要亚单位结构蛋白基因。1996年,Woodward等[79]首次以该基因为靶点建立PCR法用于肠炎沙门菌的检测。但该基因也存在于鸡伤寒等少数D群血清型中,Gong等[102]建立了以该基因为靶点的LAMP法用于检测肠炎沙门菌和鸡伤寒沙门菌,进一步的区分可通过菌落形态特征。
2.4.2 鼠伤寒沙门菌 主要靶点包括苹果酸脱氢酶编码基因mdh和多种未知功能基因。
mdh:苹果酸脱氢酶编码基因,是三羧酸循环的一种酶,普遍存在于各种生物体中。1999年Lin和Tsen[84]将鼠伤寒沙门菌和其它细菌的mdh基因序列进行比较,成功建立了能特异性扩增鼠伤寒沙门菌的PCR方法。对所有鼠伤寒沙门菌均可扩增出261 bp的产物,而其它血清型和非沙门菌菌株均未出现假阳性结果。
除上述基因外,目前还报道多种未知功能基因,包括STM0159、STM4200、STM4492、STM4495、STM4497等[81-82,85-87],其中STM4497使用频率最高。2006年Kim等[87]通过比较基因组学方法获得10个鼠伤寒沙门菌特异性基因,分别设计引物,进行PCR验证,最终确定STM4497特异性最高,此后该基因被多次用于鼠伤寒沙门菌的检测靶点[4,103]。
2.4.3 鸡白痢/鸡伤寒沙门菌 鸡白痢和鸡伤寒沙门菌属于无运动性沙门菌。由于鸡白痢、鸡伤寒沙门菌的cigR和flhB序列比其它血清型沙门菌短,利用该特点,以cigR或flhB为检测靶点,可进行无运动性沙门菌的检测[76,93-94]。此外由于鸡白痢、鸡伤寒沙门菌都不具有运动性,且在抗原型、病原学、流行病学和临床症状等多方面具有相似性,难以通过常规的诊断或血清学方法进行区分[104],因此分子检测方法至关重要。目前已报道了多种分子鉴别方法,涉及fimH、fliC、glgC、ratA、rfbS和speC等检测靶点,其中基于glgC与speC可变区的二重PCR和基于ratA可变区的PCR均具有良好的特异性[104]。
2.4.4 基于fliC基因靶点的血清型检测fliC编码沙门菌鞭毛抗原。沙门菌属具有两相鞭毛抗原,第1相称为特异相,采用小写字母a、b、c……z1、z2、z3、……z88表示,为部分血清型特有;第2相称为非特异相,采用阿拉伯数字1、2……7表示,为许多沙门菌共有[105]。沙门菌鞭毛基因高变区通常具有独特的核苷酸序列,在沙门菌各血清型中呈多样性,目前已成功应用于甲型副伤寒沙门菌、鼠伤寒沙门菌、伤寒沙门菌和猪霍乱沙门菌等的检测[75,88,98,100]。
Zhou等[98]基于甲型副伤寒鞭毛基因fliC-a建立的PCR检测方法检测结果与血清检测结果一致,但使用的样品量仅有常规方法的一半。Paião等[75]根据鼠伤寒沙门菌鞭毛基因fliC-c高变区(794—1 226)设计引物,建立多重PCR方法,对所有鼠伤寒沙门菌均检测出目的片段,而其它血清型及非沙门菌则没有扩增条带,表明鼠伤寒沙门菌fliC-c高变区可作为其检测靶点。伤寒沙门菌鞭毛抗原fliC-d,基因全长1 530 bp,其中d抗原Ⅵ区(969—1 077)具有高度特异性。Song等[88]以fliC-d为伤寒沙门菌检测靶点,建立的PCR检测方法成功区分了伤寒病例样本和健康者样本。Chiu等[100]比较了猪霍乱沙门菌与其它血清型及非沙门菌属肠杆菌科细菌的fliC基因序列,成功建立PCR检测方法。该方法结合viaB靶点能将猪霍乱沙门菌与其它细菌进行区分。
2.4.5 其它血清型 除上述常见血清型外,其它部分血清型也成功筛选到特异性靶点并建立了分子检测方法,如印第安纳沙门菌(A7P63_09100、A7P63_13850和MG752992)[95-97]、海德尔堡沙门菌(ACF69659)[80]、伤寒沙门菌(tyv、STBHUCCB_38510、STY1599、STY2879和STY4669)[81,89-92]、乙型副伤寒沙门菌(SPAB_01124)和婴儿沙门菌(M.sinI)等[81,99]。但这些基因的功能往往没有得到鉴定,其应用也不够广泛,还需进一步研究与验证。
3 讨 论
根据沙门菌的分类特点,可将分子检测方法分为四类,即沙门菌属、肠道沙门菌种、沙门菌血清群和沙门菌血清型。其中沙门菌属和沙门菌重要血清型的分子检测方法报道最多,其应用也最广泛。
沙门菌属分子检测方法的靶点主要涉及抗原决定簇、外膜蛋白、DNA结合蛋白和毒力相关基因等。这些基因中有些在个别沙门菌血清型中缺失,会引起假阴性的结果,需要特别注意,如invA[106];也有些基因普遍存在于肠杆菌科细菌中,易发生交叉反应,出现假阳性结果,如fimA和ompC[106-107]。为找到最合适的检测靶点,多个报道曾对这些基因的特异性进行了比较与评价。如Endley等[108]比较了fimA和hns引物的特异性,结果显示hns引物可对梭菌非特异性扩增,而fimA引物特异性更强。Malorny等[106]评估了ompC、oriC和invA等,结果表明invA在沙门菌PCR检测方面更加有效。Shabarinath等[64]比较了hns、invA和invE,其中hns的检测效率优于invA和invE。不同报道选择的基因不同,使用的血清型和菌株不同,得到的最优靶点也不同,但没有报道所选择的全部基因均是最优靶点,因此选择沙门菌属特异性检测靶点建立分子检测方法时,可以多选几种进行分析验证。
沙门菌血清型分子检测方法针对的靶点主要是基于比较基因组学方法分析所获得,其中很多为功能未注释的基因。本文整理了11个血清型共计36个检测靶点,其中超过一半的基因功能未注释。由于目前分析时所用的基因组数据量较大,往往具有较好的特异性。其中有些是经典的靶点,如鼠伤寒沙门菌的STM4497[87]。fliC是沙门菌另一比较重要的血清型检测靶点,其编码的鞭毛抗原是划分沙门菌血清型的重要依据,根据高变区序列设计引物,可进行不同血清型的鉴定[75,88,98,100],但fliC单独使用时不能区分某些具有相同O抗原的血清型(如猪霍乱沙门菌与丙型副伤寒沙门菌),此时需要结合其它靶点才能有效区分[100]。多个靶点结合使用的情况还出现在属和型的检测,如Khaltabadi等[109]进行鼠伤寒沙门菌鉴定时结合使用了属特异性靶点invA和型特异性靶点mdh。因此,为进一步提高沙门菌分型的特异性与敏感性,建议可同时选择两种以上的检测靶点来建立分子检测方法。
目前关于肠道沙门菌种和血清群的分子检测方法报道数量有限。由于邦戈尔沙门菌常见于冷血无脊椎动物,很少引起温血动物沙门菌病例[110],因此相关研究较少,目前仅报道了针对iroB靶点的肠道沙门菌种特异性分子检测方法[69]。而沙门菌血清群的分子检测方法主要针对几种参与合成O抗原的编码基因(如rfb、wzx和wzy等)[7,70-71]。
自2003年人类基因组计划完成之后,基因测序技术发展迅猛,尤其当2005年第一台高通量测序仪454推出后,全基因组测序技术飞跃发展,细菌全基因组测序也在不断完成[111]。采用比较基因组学方法发掘新检测靶点的研究应运而生,并发现了越来越多的沙门菌新检测靶点[96,112]。相信随着分子生物学研究的持续深入,更多、更可靠的沙门菌分子检测方法将会应用于临床检测中。



