谢 菲,黄东兰,岑东芝,张积仁
(南方医科大学附属珠江医院肿瘤中心,广州 510282)
乳腺癌是女性最常见的恶性肿瘤,其发病率占女性肿瘤的20%以上,随着年龄的增加而增加,而且早期即有远处转移,其死亡原因几乎都是远处转移所致。乳腺癌的转移是一个较为复杂、由多基因参与及多步骤完成的过程。与肿瘤转移密切相关的基因被认为是抗转移干预治疗的潜在靶点,深入阐述乳腺癌转移的机制将为预测和阻止乳腺癌转移提供坚实的基础。目前临床上病理和免疫组化指标不能准确判断乳腺癌患者转移的风险[1],严重影响肿瘤患者治疗的疗效和预后。为了了解目前乳腺癌转移相关基因的研究概况及发展趋势,本文对国际上2001~2010年10年间乳腺癌转移相关基因研究的文献进行了计量学分析。
1 资料与方法
1.1 一般资料 数据库的选择以荷兰Elsevier Science出版公司建立的Embase数据库、美国国立医学图书馆建立的Medline/Pubmed数据库及美国生物科学信息服务社建立的世界上最大的有关生命科学的文摘和索引数据库——BIOSIS Preview数据库为数据源,统计这3个数据库2001年1月至2010年12月收录的有关乳腺癌转移相关基因的文献,选择这3个收录范围既有交叉又有区别的权威数据库来确保数据的完整性及准确性。
1.2 方法
1.2.1 检索方法 为提高文献的查准率,对Embase数据库进行Emtree扩展检索,辅以自由词的题目及摘要字段的检索;对Pubmed/Medline数据库进行MeSH检索,辅以自由词的题目及摘要字段的检索;对BIOSIS Preview数据库进行主题检索。拟定的检索词为乳腺癌、转移及基因。乳腺癌的检索词包括:(1)MeSH 词breast neoplasm;(2)EMtree词breast cancer;(3)自由词检索使用 breast cancer、breast tumor*、breast neoplasm*、breast carcinoma*、mammary neoplasm*。转移的英文 检索词包括:(1)MeSH 词 neoplasm metastasis;(2)EMtree词检索使用 metastasis、cancer recurrence、cancer relapse及metastasis inhibition;(3)自由词检索使用 metastasis、metastases。基因的英文检索词为gene。
1.2.2 文献纳入与排除 文献形式主要为论着、会议论文及会议摘要,纳入全部与乳腺癌转移相关基因及基因产物相关且以人类为研究对象的临床研究,能提供足够信息的摘要或可以获取全文资料的文献。排除了综述、个案报道、回信、书籍、社论、无法获取摘要的文献、与转移相关的非基因研究、针对基因的结构和机制等研究、细胞株体外研究、老鼠及其他动物体内试验及重复文献与其他与研究目的不符的文献。纳入与排除的过程,由2名研究人员独立进行,最终结果互相比较,不一致的地方互相讨论,或向临床医生、实验研究人员(研究员)请教。
1.2.3 文献计量分析 采用NoteExpress X3软件对所检索文献进行管理,使用Excel 2007软件对最终纳入的文献的出版年、作者、国家、研究机构、发表期刊及基因进行计量学分析。
2 结 果
2.1 检索结果 Embase数据库检出文献2246篇,其中论着1573篇,综述353篇,会议论文105篇,会议摘要77篇,信件35篇,笔记33篇,社论31篇。Pubmed/Medline数据库检出文献1009篇,其中综述126篇。BIOSIS Preview数据库检出文献1441篇,其中论着974篇,会议论文440篇,书籍目录22篇,信件9篇。三库共检出文献4696篇。根据前述的文献纳入排除标准,最终纳入研究的文献共有363篇。
2.2 文献发表年限分析 对最终纳入的363篇文献进行发表年限分析,其年度发表数量及变化见图1。从各年度的文献量分布上可以看出,近10年乳腺癌转移相关基因的临床研究在2001~2007年间呈缓慢增长趋势,研究处于萌芽阶段,2008年总体开始呈现明显上升趋势,2009~2010年间呈平稳发展状态,说明科研人员对该领域的研究越来越多,目前研究正处于稳定发展的阶段。由此推测,今后知识研究文献仍将会保持一定的增长速度,进一步达到研究的成熟阶段。
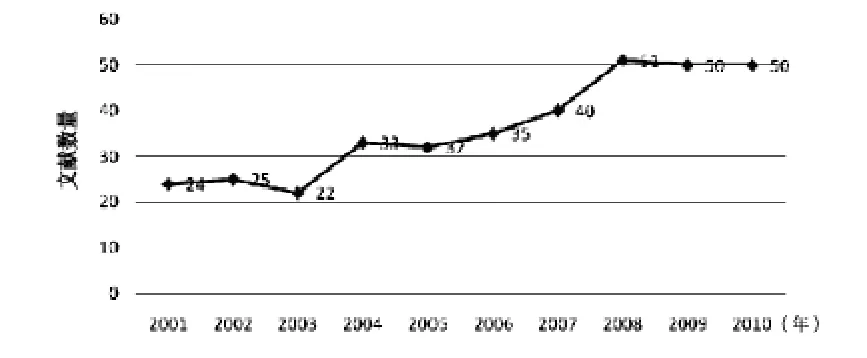
图1 乳腺癌转移相关基因文献年份分布
2.3 文献发文量国别分析 通过对363篇文献的发表国家的统计分析得知,共有45个国家发表了关于乳腺癌转移相关基因方面的研究。发文量前20位的国家见图2。可以发现,中国发文量为84篇,位列第1位,为发文量最多的国家,占据了文献总数的23.1%,遥遥领先于其他国家。美国继中国之后,发文量为65篇,占据了文献总数的17.9%,德国、日本、意大利、英国、法国、波兰、韩国及土耳其分列3~10位。以上表明,中国目前是研究乳腺癌转移相关基因最活跃的国家,体现了我国在该领域研究的先进地位和较高的科研能力,其他传统型生物学领域研究大国也是这一领域的主要力量。发文量前10名的国家中,除中国外,均为高收入国家。除中国和美国外,在发文量前20名的国家中,半数为欧洲国家,说明欧洲在该领域研究中也占据了重要地位。从学术生产力方面分析[2],由于我国人口基数大,人均发文量与美、德、日等国相距甚远,因此我国的研究者应加大对该领域研究投入,吸取国外优秀科研经验,加强与国外权威科研机构合作,从而提高我国的学术生产力,增强我国的学术影响力。
2.4 文献期刊分析 对最终纳入的363篇论文进行统计分析,共发表在140种生物医学期刊上,平均每刊发文2.59篇。发文量达8篇的期刊有9种,根据2009年公布的SCI期刊目录,上述期刊均为SCI收录期刊,其发文量及影响因子见表1,其中Cancer Research的影响因子最高。根据布拉德福分散定律[4],从发文量的角度,Breast Cancer Research and Treatment是该研究领域核心的英文期刊。通过查阅以上9种期刊可以较快地了解该领域的研究动态。
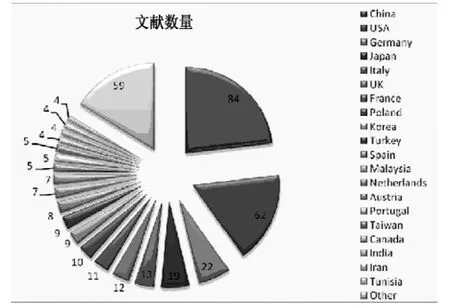
图2 乳腺癌转移相关基因文献国家分布
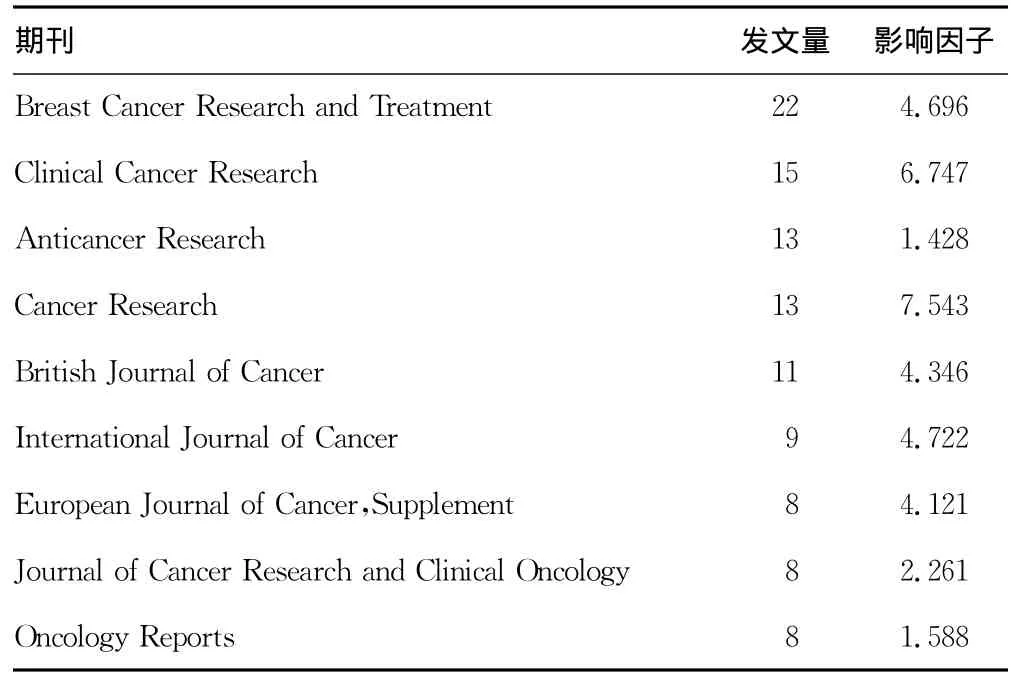
表1 发文量达8篇及其以上的期刊
2.5 文献发表的研究机构 按第一作者所在单位进行统计,共统计出280个研究机构在进行乳腺癌转移相关基因方面的研究,发文量达4篇的研究机构有6个,分别为Tianjin Medical University(12 篇),The Netherlands Cancer Institute(6篇),The University of Texas M.D.Anderson Cancer Center(4篇),General Hospital of Chinese People′s Liberation Army(4篇),University of Lodz(4篇),Fu Dan University(4篇),University of Hong Kong(4篇),其中我国研究机构有4个,占据了主要地位,说明我国在乳腺癌转移相关基因方面实力较强。但总体来讲,在乳腺癌转移相关基因研究领域,无论国际上还是国内,均未形成一些核心研究机构。
2.6 核心作者分析 经调研统计得知,Netherlands的Mook、Malaysia的Naidu在10年间以第一作者发表了乳腺癌转移相关基因方面的论文3篇,为该领域发文量最大的作者。国外有学者主要研究MammaPrint70基因模型在预测乳腺癌预后及转移风险方面的作用[4-6],也有很多学者研究方向为基因多态性与乳腺癌患病风险的关系[7-9]。根据洛特卡和普赖斯对科学家的生产率和活动规律的研究,发表论文为N篇以上的作者为该学科的杰出科学家,即核心作者。计算公式为:N=0.749×(ηmax)1/2。式中ηmax为发文量最多的作者发表的论文数量[10]。根据上述公式,发表论文达2篇的作者为该研究领域的核心作者。经统计核心作者共有20位,对核心作者所在国家进行统计发现,美国、英国各有3位,日本、荷兰、爱尔兰、法国各有2位,马来西亚、加拿大、德国、韩国、意大利、瑞典各有1位,所有核心作者均在发达国家,特别是欧美地区,说明欧美的学者在该研究领域造诣较深。
2.7 乳腺癌转移相关基因的文献量分析 通过对最终纳入的363篇文献的详细阅读,共统计出567个乳腺癌转移相关基因。其中研究文献量达6篇的基因有18个,它们的基因名称及所涉及的文献量如下:ERBB2(52)、TP53(26)、ESR1(23)、NME1(16)、KRT19(14)、PGR(12)、VEGFA(12)、EGFR(10)、PTGS2(10)、BRCA1(8)、CD44(8)、CDH1(8)、MMP9(7)、BCL2(6)、CCND1(6)、GSTP1(6)、MKI67(6)、PTEN(6)。其中对ERBB2基因研究的文献量最多,共52篇,远远多于其他基因,TP53基因作为最早发现的抑癌基因,紧随其后,有26篇文献进行研究。而在2006~2010年间文献量达4篇的基因有19个,ERBB2(33)、ESR1(14)、VEGFA(10)、TP53(10)、PTGS2(6)、PGR(6)、MMP9(6)、KRT19(6)、EGFR(6)、NME1(5)、BRCA1(5)、BCL2(5)、VEGFC(4)、TERT(4)、SCGB2A2(4)、GSTP1(4)、CXCL12(4)、CD44(4)、CCND1(4)。可以发现,近10年间与近5年间研究较多的基因稍有变化,ERBB2、TP53、ESR1、NME1、KRT19、PGR、VEGFA、EGFR、BRCA1、CD44、BCL2等基因仍然是研究的热点,CDH1、MKI67、PTEN基因在近5年的研究逐渐减少,对CXCL12、TERT、VEGFC基因的研究在近5年逐渐增多。
3 讨 论
通过对最终纳入的363篇乳腺癌转移相关基因研究文献的计量学分析,可以发现,乳腺癌转移相关基因方面的研究目前正处于稳定发展阶段。中国、美国、日本、英国、德国等国均已经开展了这方面的研究,我国在发文量方面遥遥领先于其他国家,但在人均发文量方面远远落后于其他国家。在发文量达4篇的6个研究机构中,有4个在中国,充分说明我国在乳腺癌转移相关基因方面研究方面已经形成了自己的优势,并有形成核心研究机构的趋势。各研究机构发文量均较少,目前尚未形成该研究领域的核心研究机构,迫切需要国内外研究机构间通力合作。各位学者发表文献数也较少,尚无该领域的领军人物。由此可见,目前的研究较为分散,多为单中心研究,样本量较小,迫切需要进行多中心、大样本的研究,因此研究人员之间甚至是国家之间合作是乳腺癌转移相关基因方面研究的趋势。
基因检测技术的发展,促使基因功能研究从基因产物表达检测逐渐发展为基因突变、启动子甲基化、单核苷酸多态性检测。而随着基因芯片技术的发展,乳腺癌转移相关基因的研究逐渐由单基因研究向多基因研究方向发展。多基因模型开始多用于评估乳腺癌预后,近年来开始有人把多基因模型用于评估乳腺癌转移风险,如国外已进入临床应用的MammaPrint70基因模型[7],以及Oncotype Dx21基因模型[12],Tutt等[12]和Yau等[13]构建的14基因模型,Culhane和 Quackenbush[14]及Landemaine等[15]建立的预测乳腺癌患者肺转移风险的6基因模型。由于乳腺癌转移是一个复杂的、由多基因参与及多步骤完成的过程,应用单一基因预测乳腺癌的转移风险,其敏感度及精确度均欠佳,而应用多基因模型可显着提高其预测的敏感度及精确度。建立多基因预测模型的根本在于寻找预测价值高、性能稳定的候选基因。本文通过对文献的计量学分析,初步筛选了一些研究较成熟、预测性好的基因,为建立高效的多基因预测模型奠定了基础。多基因预测模型为乳腺癌复发转移风险预测、治疗疗效预测及个体化治疗方案的制订提供了可能,逐渐成为乳腺癌转移研究的热点,显示出巨大的潜力和广泛的应用前景。
[1]Fisher B,Jeong JH,Bryanl J,et al.Treatment of lymphnode-negative,oestrogen_receptor-positive breast cancer:long-term findings form National Surgical Adjuvant Breast and Bowel Project randomised clinical trials[J].Lancet,2004,364(9437):858-868.
[2]Swaminathan M,Phillips-Bute BG,Grichnik KP.Bibliometric analysis of global clinical research by anesthesia departments[J].Anesth Analy,2007(105):1741-1746.
[3]罗式胜.文献计量学概论[M].广州:中山大学出版社,1994:34-46.
[4]Mook S,Schmidt MK,Weigelt B,et al.The 70-gene prognosis profile predicts early metastases in postmenopausal breast cancer patients[J].Cancer Res,2009,69Suppl 2:S124.
[5]Mook S,Schmidt MK,Weigelt B,et al.The 70-gene prognosis signature predicts early metastasis in breast cancer patients between 55and 70years of age[J].Ann Oncol,2010,21(4):717-722.
[6]Mook S,Knauer M,Bueno-de-Mesquita JM,et al.Metastatic potential of T1breast cancer can be predicted by the 70-gene MammaPrint signature[J].Ann Surg Oncol,2010,17(5):1406-1413.
[7]Naidu R,Yip CH,Taib NA.Polymorphisms of HER2 Ile655Val and cyclin D1(CCND1)G870Aare not associated with breast cancer risk but polymorphic allele of HER2is associated with nodal metastases[J].Neoplasma(Bratislava),2008,55(2):87-95.
[8]Naidu R,Har YC,Taib NA.Polymorphism of FGFR4 Gly388Arg does not confer an increased risk to breast cancer development[J].Oncol Res,2009,18(2/3):65-71.
[9]Naidu R,Har YC,Taib NA.Associations between hypoxia-inducible factor-1(alpha)(HIF-1(alpha))gene polymorphisms and risk of developing breast cancer[J].Neoplasma,2009,56(5):441-447.
[10]邱均平.信息计量学[M].武汉:武汉大学出版社,2007:106-192.
[11]Cobleigh MA,Tabesh B,Bitterman P,et al.Tumor gene expression and prognosis in breast cancer patients with 10 or more positive lymph nodes[J].Clin Cancer Res,2005,11(24Pt 1):8623-8631.
[12]Tutt A,Wang A,Rowland C,et al.Risk estimation of distant metastasis in node-negative,estrogen receptor-positive breast cancer patients using an RT-PCR based prognostic expression signature[J].BMC Cancer,2008(8):339.
[13]Yau C,Esserman L,Moore DH,et al.A multigene predictor of metastatic outcome in early stage hormone receptor-negative and triple-negative breast cancer[J].Breast Cancer Res,2010,12(5):R85.
[14]Culhane AC,Quackenbush J.Confounding effects in“A six-gene signature predicting breast cancer lung metastasis”[J].Cancer Res,2009,69(18):7480-7485.
[15]Landemaine T,Jackson A,Bellahcene A,et al.A six-gene signature predicting breast cancer lung metastasis[J].Cancer Res,2008,68(15):6092-6099.



