叶 盛,喻 臻,陈 爽,凌 华△,熊 宇,李 勤
(重庆市疾病预防控制中心:1.微生物检测所;2.传染病预防控制所,重庆 400042)
论着·临床研究 doi:10.3969/j.issn.1671-8348.2016.30.016
重庆市2009~2014年甲型H1N1流感病毒分离株HA基因变异分析*
叶 盛1,喻 臻1,陈 爽1,凌 华1△,熊 宇2,李 勤2
(重庆市疾病预防控制中心:1.微生物检测所;2.传染病预防控制所,重庆 400042)
目的 对2009~2014年分离得到的甲型H1N1流感病毒进行血凝素(HA)基因测序分析,与世界卫生组织(WHO)推荐疫苗株A/California/07/2009(H1N1)比对,研究其基因变异情况。方法 按分离时间及地点不同,选取47株2011~2014年分离得到的甲型H1N1流感病毒进行测序,同时选取既往已有的25株2009年甲型H1N1流感病毒的序列结果,经分子生物学软件进行核苷酸及氨基酸序列分析,并绘制系统进化树。结果 2009、2011~2014年的新型甲型H1N1流感病毒与疫苗株均位于一个大的分枝下,各病毒间的关系均较近。72株分离株的HA基因总的核苷酸差异在0~2.7%之间,氨基酸差异在0~3.1%之间;与疫苗株A/California/07/2009(H1N1)的核苷酸差异在0.4%~2.4%之间,氨基酸差异在0.9%~3.1%之间。氨基酸变异情况随着年份增加累积得更多,但在氨基酸序列上仍显示为低致病性流感病毒特征;2009年有9株病毒株丢失481位的糖基化位点,2013年有6株病毒株增加位于162位的糖基化位点。结论 WHO推荐的甲型H1N1疫苗总体上对重庆地区的人群仍有较好的保护作用,但随着时间推移,部分甲型H1N1流感病毒株可能已发生抗原漂移,需继续对其监测。
甲型H1N1;流感病毒;序列分析;HA基因
流行性感冒(简称流感)是由流感病毒引起的具有高度传染性的急性呼吸道传染病,传播迅速,短时间内可造成局部地区的暴发或全球范围的流行,也是人类迄今为止尚无法有效控制的传染病之一。流感病毒属正黏病毒科(orthomyxoviridae),按核衣壳蛋白(nucleocapsid protein,NP)和基质蛋白(matrix protein,MP)不同,分为甲(A)、乙(B )、丙(C)3 型。流感病毒的流行与流感病毒的抗原变异密切相关,甲型流感病毒抗原变异主要有抗原漂移和抗原转变,一般认为抗原漂移常带来甲型流感病毒不同程度的流行。甲型流感病毒的亚型由血凝素(HA)和神经氨酸酶(NA)两种主要表面蛋白决定。HA基因是流感病毒的主要抗原基因,其编码的HA蛋白具有免疫原性,能使人体产生保护性抗体[1];同时其承受的免疫压力也较大,进化和变异速度相对较快。
2009年暴发于墨西哥、美国的新型甲型流感,其病原为重组的新型流感病毒[2],该病毒传播迅速,几个月内即在世界范围内流行。该病毒于2009年5月进入我国大陆[3],2009年6月重庆即出现首例甲型H1N1流感病例,自此,该病毒由输入性个案病例迅速本土化并造成重庆市本地区内的暴发流行[4-5]。随着人群自然免疫和疫苗的接种,甲型H1N1流感病毒活动性似乎逐渐减弱,但其对人群的危害仍未消除。持续关注甲型H1N1流感病毒的HA基因和HA蛋白的变异情况,可为流感疫苗的筛选及病毒演化趋势判断提供重要的参考依据。
1 材料与方法
1.1 病毒来源 2011~2014年,对重庆市疾病预防控制中心流感网络实验室各哨点医院的样品及各区县流感暴发样品进行核酸检测和病毒分离,得到流感病毒株。将得到的毒株中的甲型H1N1病毒株按分离时间及地点不同,选取代表毒株。

表1 甲型H1N1流感病毒HA基因扩增引物序列
1.2 方法
1.2.1 目的基因扩增 将选取的病毒分离株用全自动样品处理工作站(QIA Symphony SP,QIAGEN,德国)及该工作站配套RNA提取试剂(QIAsymphony Virus/Bact Mini Kit,QIAGEN,德国)提取病毒RNA,经逆转录试剂盒(SuperScript Ⅲ First-Strand Synthesis System,Invitrogen,美国)逆转录后制备出病毒cDNA,再取病毒cDNA为模板,用普通聚合酶链式反应(PCR)预混装试剂盒(GoTaq®Hot Start Green Master Mix,Promega,美国)和表1中的引物(Invitrogen合成,美国)完成病毒HA基因核酸的PCR扩增,经电泳(E-Gel®琼脂糖凝胶电泳系统,Invitrogen,美国)后,观察到目的条带确认目的片断扩增成功。
1.2.2 基因测序与序列分析 含有目的片断的PCR反应产物直接送Takara和Invitrogen公司进行基因测序。测序所得序列经校对后采用BIOEDIT 7.0软件进行序列比对和剪切;采用MEGA 4.0.2基于Neighbour-joining(NJ)法生成系统发生树,并进行序列分析和氨基酸位点差异分析;使用DNASTAR 5.0软件包的MegAlign软件进行核苷酸和氨基酸序列差异分析。
2 结 果
2.1 流感监测结果与病毒分离结果 除去2009年6月至2010年3月甲型H1N1在重庆地区的首次暴发流行期,从2010年11月至2015年11月共计61个自然月中,甲型H1N1共计出现两次流行,分别是2010年12月至2011年3月,和2012年12月至2014年4月(图1);在这61个自然月中,共采集标本26 108份,共分离到流感病毒2 098株,总体病毒分离率为8.04%,共分离得到甲型H1N1毒株288株,占所分离到病毒总数的13.7%。在上述288株病毒株中选取47株甲型H1N1病毒株经基因测序后序列整理与拼接,得到其HA基因组序列信息。
2.2 基因进化分析 经与世界卫生组织(WHO)推荐疫苗株A/California/07/2009(H1N1) ,以下简称CA/07/09(HA基因Genbank收录号为GI:758899355)比对,47株2011~2014年分离病毒株均未出现核苷酸插入与缺失。将上述47株病毒的HA基因序列与已经有的2009~2010年流行期的25株H1N1病毒的HA基因序列和A/Beijing/262/1995(H1N1)和A/swine/Jiangsu/s16/2011(H1N1)的HA序列对齐剪切后生成基因进化树(图2)。在图2所示的进化树上,除A/Beijing/262/1995(H1N1)和A/swine/Jiangsu/s16/2011(H1N1)两株毒株外,2009年、2011~2014年的新型甲型H1N1流感病毒与疫苗株均处于一个大的分枝下,各病毒间的关系均较近,相邻年份的病毒聚集并交叉分布在相邻的分枝上:2009与2011年分离病毒株包括疫苗株均交叉分布于一个大的分枝上,而2013~2014年的病毒株分布于另一大分枝上。

图1 重庆市2010年11月至2015年11月流感检测情况
2.3 核苷酸及氨基酸差异情况 72株分离株的HA基因总的核苷酸差异在0~2.7%之间,氨基酸差异在0~3.1%之间;与疫苗株CA/07/09的核苷酸差异在0.4%~2.4%之间,氨基酸差异在0.9%~3.1%之间,见表2。
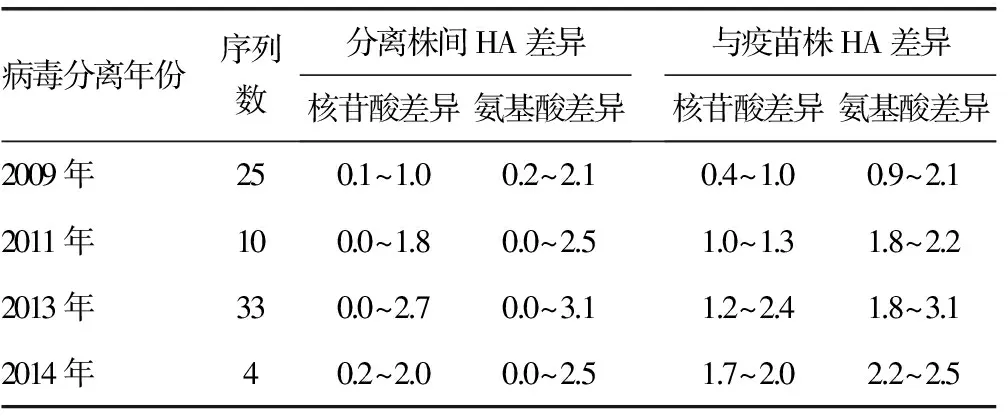
表2 2009~2014年甲型H1N1流感病毒株核苷酸及氨基酸差异情况(%)
2.4 氨基酸变异情况 2009~2014年72株甲型H1N1分离株与疫苗株CA/07/09的HA氨基酸序列存在3个共同的差异位点,分别是P83S (减去信号肽序列号,以下序列位点相同)、S203T及I321V。除此之外,各年度分离到的病毒株与CA/07/2009在不同的氨基酸位点各有差异。2011年的10株病毒株在2009年的变异基础上全部增加R223Q、E374K变异;2013年的33株病毒株全部出现D97N、S185P/T、E374K、S451N变异;2014年的4株病毒株则全部出现D97N,K163Q,S185T,R223Q,A256T,K374E,S451N,E499K变异,见表3。

表3 2009~2014年毒株相对疫苗株CA/07/09氨基酸位点变化情况

图2 基于HA基因的系统进化树
2.5 致病性分析 本次研究的所有甲型H1N1流感病毒均表现为低致病性特征。病毒裂解位点氨基酸序列为VPSIQSR↓GLFGAIA,HA1与HA2间仅由一个碱性氨基酸R(第327位)连接,其水解位点附近未见多个连续碱性氨基酸,属于低致病性流感病毒特征[6]。
2.6 糖基化位点分析 本次研究中的所有甲型H1N1病毒,其糖基化位点大部分与疫苗株CA/07/09相同,共有8个稳定的糖基化位点,其中,HA1区有6个糖基化位点,分别位于10、11、23、87、276和287位点,HA2区有2个糖基化位点,分别位于481和540位点。2009年有9株病毒株(9/25)丢失481位点的糖基化位点,2013年有6株毒株(6/33)增加位于162位的糖基化位点,见图3。
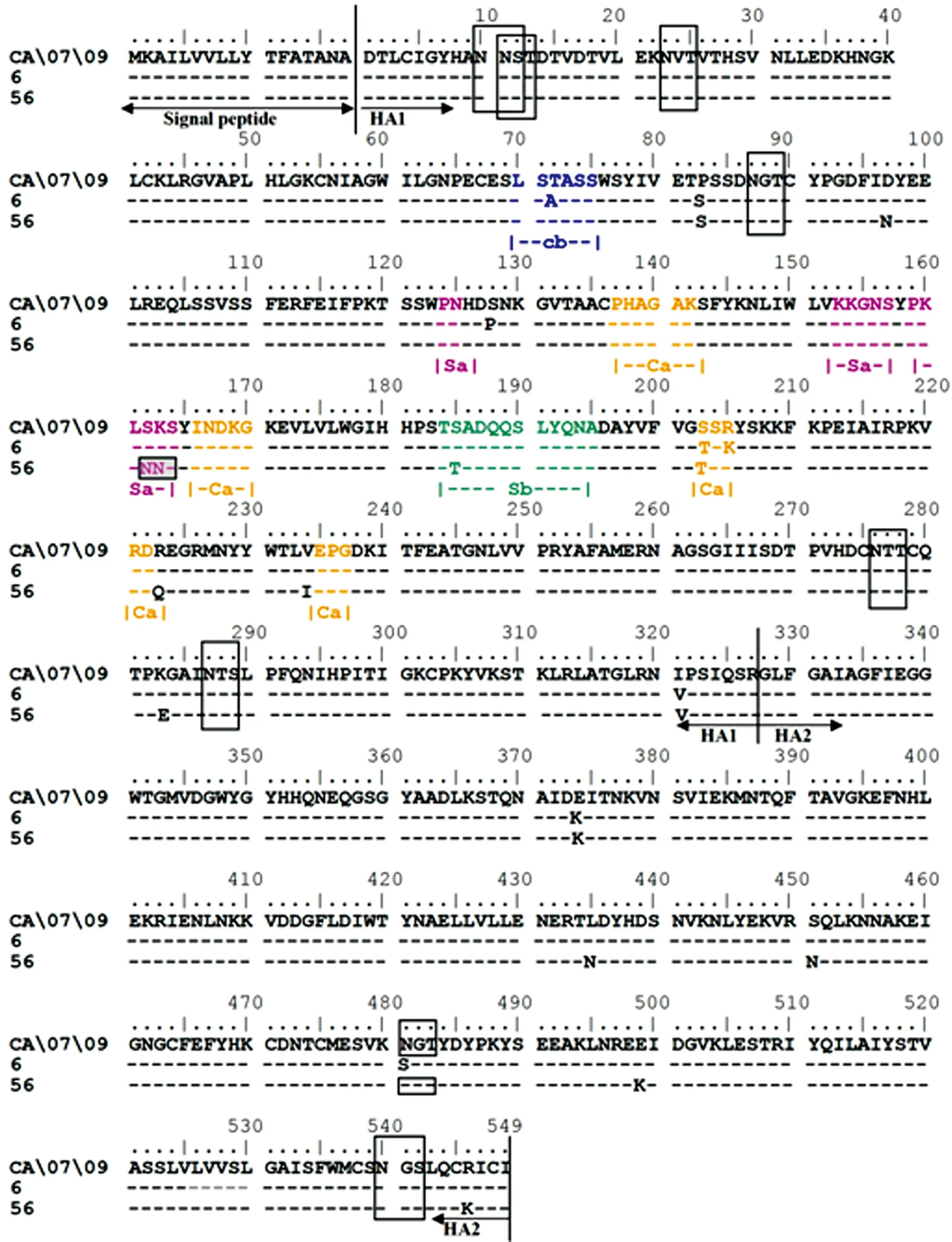
方框“□”区域:潜在糖基化位点;图中数字6 :毒株“A/重庆九龙坡/SWL382/2009”,数字56:毒株“A/重庆渝中/SWL11297/2013(H1)”。
图3 WHO推荐疫苗株与部分病毒分离株的HA基因编码氨基酸序列对比
3 讨 论
自2009~2010年初的甲型H1N1暴发流行过后,重庆地区从2010年11月至2015年11月又出现了两轮甲型H1N1流感病毒流行高峰,这与我国其他地区的情况基本相同[7]。本次研究选择了2011~2014年分离的共47株甲型H1N1流感病毒株,以及25株2009年的甲型H1N1病毒核苷酸资料,对共计72株甲型H1N1病毒的HA基因进行了分析。从核苷酸及氨基酸差异性上看,所有毒株与疫苗株A/California/07/2009(H1N1)仍保持较高的同源性。在基因进化树上,所有甲型H1N1包括疫苗株均在同一大分枝上,各病毒株与疫苗株的距离不大,提示疫苗对重庆地区的甲型H1N1应仍有效;但2013年与2014年的病毒比2009年和2011年的病毒离疫苗株更远,提示随着年份增加,H1N1病毒与疫苗株的基因序列差异在增加,这也与福建、江苏、陕西等地的甲型H1N1病毒与疫苗株的情况类似[8-9]。氨基酸变异情况对比显示,本次研究中甲型H1N1流感病毒共有的氨基酸序列变异,随着年份增加而累积得更多,其中,2013年与2014年的毒株变异情况也与江苏省的甲型H1N1流感病毒变异情况相似。这说明因氨基酸序列改变而发生的抗原性变化逐年积累的可能性较大。
与甲型H1N1流感病毒HA抗原性密切相关的4个抗原决定簇位于HA1区域,分别是Ca(137~142、166~170、203~205、221~222、235~237)、Cb(70~75)、Sa(124~125、153~157、159~164)、Sb(184~195)[10-12]。一般认为,HA1区蛋白分子上有4个以上氨基酸位点发生变异,且变异涉及2个或2个以上抗原决定簇上的位点,或一个涉及抗原决定簇另一个涉及受体结合位点,将可能会出现新的抗原漂移株[13]。本次研究中,2009、2011和2014年的毒株均未出现上述情况。但2013年的毒株有14株毒株(14/33)出现4处及以上的氨基酸位点变异,且涉及2个及以上抗原决定簇。其中,A/重庆渝中/SWL59/2013(H1)出现A73E、K163Q、S185T、S203T 4个位点的变异,且4个位点分别位于Cb、Sa、Sb和Ca区。因此,该甲型H1N1流感病毒株可能出现抗原漂移现象,但2014年的毒株暂未观察到上述位点的持续变异,上述抗原漂移是否能够持续存在及积累,还需要对将来可能出现的甲型H1N1流感病毒继续监测。
糖基化在稳定蛋白的三维结构和其被水解,以及阻碍抗体的识别中起到重要作用。糖基化位点的改变或增减会对病毒的抗原性和生物特性带来一定的影响;特别是当糖基化位点的改变发生在抗原决定簇时,可能会造成病毒抗原性改变,产生抗原漂移[14-15]。本次研究中,有9株2009年的甲型H1N1病毒株丢失481位的糖基化位点,但由于481位点不在抗原决定簇上,481位的糖基化位点丢失对于毒株的抗原性改变影响可能不大;但另有6株2013年的毒株出现162位点糖基化,且162位点刚好位于Sa区,因此,该6株病毒有可能出现抗原性改变。同时,该6株病毒株中,A/重庆渝中/SWL11297/2013(H1)、A/重庆渝中/SWL11434/2013(H1)、A/重庆渝中/SWL58/2013(H1)和A/重庆渝中/SWL11827/2013(H1)病毒株同时出现了3个抗原决定簇的氨基酸变异,这几株病毒的抗原性是否有改变,需对其进行进一步的抗原性分析。
综上所述,本次研究提示,2009~2014年重庆市甲型H1N1流感病毒与疫苗株A/California/07/2009(H1N1)相比,虽然变异程度逐年积累,但总体上甲型H1N1流行株与疫苗株仍保持较高的同源性,疫苗对本地区人群仍有较好的保护作用。但随着时间推移,相对于疫苗株,2013和2014年的甲型H1N1病毒株已经出现一定的变异,部分病毒株的HA蛋白可能已发生抗原漂移。因此,持续监测甲型H1N1流感病毒,可以及时准确地发现抗原漂移株,掌握甲型H1N1流感病毒的流行与进化规律,预测其发生、发展情况,为可能的暴发流行做好充分的准备,为科学有序地防控甲型H1N1流感提供依据。
[1]Drescher J,Aron R.Influence of the amino acid differences between the hemagglutinin HA1 domains of influenza virus H1N1 strains on their reaction with antibody[J].J Med Virol,1999,57(4):397-404.
[2]Smith GJ,Vijaykrishna D,Bahl JA,et al.Origins and evolutionary genomics of the 2009 swine-origin H1N1 influenza A epidemic[J].Nature,2009,459(7250):U107-1122.
[3]王伟,潘明,常国辉,等.中国内地首例确诊甲型H1N1流感病例的实验室检测[J].病毒学报,2009,25(S1):4-7.
[4]李勤,肖达勇,龙江,等.重庆市甲型H1N1流感流行特征及趋势分析[J].现代预防医学,2010,37(21):4003-4006.
[5]肖邦忠,李勤,凌华,等.重庆市2009年6~10月甲型H1N1流感疫情特征分析[J].重庆医学,2010,39(2):222-223,240.
[6]Peiris JS,de Jong MD,Guan Y.Avian influenza virus (H5N1):a threat to human health[J].Clin Microbiol Rev,2007,20(2):243-267.
[7]资海荣,郭艳,邓斐,等.江苏省2013年甲型H1N1(09pdm)流感病毒血凝素和神经氨酸酶分子特征分析[J].南京医科大学学报(自然科学版),2015,35(6):812-822.
[8]马萍,许晶,张蕾,等.陕西省2012~2014年甲型H1N1流感的基因特性研究[J].中国预防医学杂志,2015,16(1):9-13.
[9]谢剑锋,沈晓娜,王美爱,等.福建省甲型H1N1流感病毒基因特征研究[J].病毒学报,2014,30(1):37-43.
[10]Xu R,Ekiert DC,Krause JC,et al.Structural basis of preexisting immunity to the 2009 H1N1 pandemic influenza virus[J].Science,2010,328(5976):357-360.
[11]Igarashi M,Ito K,Yoshida R,et al.Predicting the antigenic structure of the pandemic (H1N1) 2009 influenza virus hemagglutinin[J].PLoS One,2010,5(1):e8553.
[12]Huang JW,Lin WF,Yang JM.Antigenic sites of H1N1 influenza virus hemagglutinin revealed by natural isolates and inhibition assays[J].Vaccine,2012,30(44):6327-6337.
[13]郭元吉,程小雯.流行性感冒病毒及其实验技术[M].北京:中国三峡出版社,1997:41-47.
[14]Schulze IT.Effects of glycosylation on the properties and functions of influenza virus hemagglutinin[J].J Infect Dis,1997,176(Suppl 1):S24-S28.
[15]Abe Y,Takashita E,Sugawara K,et al.Effect of the addition of oligosaccharides on the biological activities and antigenicity of influenza A/H3N2 virus hemagglutinin[J].J Virol,2004,78(18):9605-9611.
Variation of HA gene of influenza A H1N1 influenza virus isolated from 2009 to 2014 in Chongqing city*
Ye Sheng1,Yu Zhen1,Chen Shuang1,Ling Hua1△,Xiong Yu2,Li Qin2
(1.DepartmentofMicrobeDetection;2.DepartmentofInfectionDiseaseControlandPrevention,ChongqingCenterforDiseaseControlandPrevention,Chongqing400042,China)
Objective In order to analyze the variation of HA genes of influenza viruses (H1N1) by being compared with the vaccine strain A/California/07/2009(H1N1) recommended by WHO,influenza viruses (H1N1) isolated from 2009 to 2014 were selected to do this study.Methods According to the different isolating time and place,47 strains of H1N1 collected from 2011 to 2014 were selected.Then the 47 strains′ nucleotide sequence of HA genes which were sequenced in the study and other 25 sequences of HA genes which were sequenced in 2009 were collected.Nucleotide and amino acid sequences were analyzed by using molecular biology software,and the phylogenetic trees were drawn.Results A total of 72 strains isolated from 2009 to 2014 were closely related to the vaccine strain A/California/07/2009(H1N1),the nucleotide variance and amino acid variance between the 72 strains were 0-2.7% and 0-3.1% respectively.Compared with the vaccine strain A/California/07/2009(H1N1),the nucleotide variance and amino acid sequence variance were 0.4%-2.4% and 0.9%-3.1% respectively.The amino acids sequence indicated that,although the variance was increased by years,the H1N1 viruses were still showed characteristics of low pathogenic influenza viruses.It was also found that there were 9 strains lost their potential glycosylation site at HA protein site 481 in 2009,while in 2013 there were 6 strains got new potential glycosylation sites at HA protein site 162.Conclusion The vaccines (H1N1) recommended by WHO was still protective to people in Chongqing.But as time goes by,antigen drift may occur in some new antigenic drift strains and the routine monitoring of influenza viruses should be continued.
H1N1;influenza virus;sequence analysis;HA gene
重庆市卫计委医学科研基金资助项目(2011-2-316)。 作者简介:叶盛(1981-),主管技师,硕士,主要从事流感、禽流感的检测与研究。△
R373.1+3
A
1671-8348(2016)30-4226-04
2016-04-01
2016-06-29)



