范定坤,张吉贤,付域泽,马 涛,毕研亮,张乃锋
(中国农业科学院饲料研究所 农业农村部饲料生物技术重点实验室,北京 100081)
微生物广泛存在于动物消化道内,与宿主形成共生关系,并据此执行基本的生理功能。反刍动物瘤胃微生物相较于其他动物更为复杂多样,具有纤维分解、脂肪降解、植物蛋白水解、微生物蛋白合成等功能[2]。此外,瘤胃菌群产生的代谢物如挥发性脂肪酸等能够通过调控宿主基因表达促进瘤胃上皮发育,与宿主生理功能密切相关[3]。然而到目前为止,瘤胃环境中不同细菌占据的生态位仍不明确,细菌多样性及数量仍然是基于测序算法估测得到的。在现有1 300个瘤胃基因组的基础上[4],Stewart等[1]通过宏基因组测序增加了4 941个新的宏基因组数据,而且仍有未知的微生物存在于反刍动物瘤胃中。目前,瘤胃中可培养微生物仅有500余株,不足瘤胃微生物总数的10%[4],严重阻碍了基于菌株水平对瘤胃微生物菌群结构和功能的研究。培养组学是一种采用多种培养条件,结合高通量测序技术鉴定菌种的培养方法。高通量、并行化的培养组学技术在瘤胃微生物中的应用,为在菌株水平上研究重点菌株功能及其与宿主互作关系提供了新的视角。本文从瘤胃微生物的组成结构出发,系统阐述了培养组学在反刍动物瘤胃微生物培养的应用现状、挑战及展望,以期为进一步探索瘤胃微生物多样性、补充瘤胃菌株资源提供理论基础和参考依据。
1 反刍动物瘤胃微生物的组成与结构
瘤胃微生物是动物界迄今为止所描述的最多样化的消化道生态系统之一,其中包括1010~1011CFU·g-1的细菌、108~109CFU·g-1的古菌、105~106个·g-1的原虫、103~104CFU·g-1的真菌以及未知含量的病毒[1]。在出生后的24 h,反刍幼畜的瘤胃壁开始富集需氧菌和兼性厌氧菌群,并占据主导地位[5]。之后的2周内,瘤胃细菌的种类和丰度发生极大变化,需氧菌和兼性厌氧菌被逐步取代,菌群相对丰度以拟杆菌门升高和变形菌门降低为主要特征,厚壁菌门内部则从链球菌属向普雷沃氏菌属转变[6-8]。成年反刍动物瘤胃菌群的构成较为稳定,主要包括厚壁菌门、拟杆菌门以及变形菌门,与其幼龄阶段的菌群组成有明显差异[2]。厚壁菌门和拟杆菌门是瘤胃微生物的优势菌群,并且在纤维降解中发挥了重要作用[2]。例如,厚壁菌门的黄色瘤胃球菌(Ruminococcusflavefaciens)、白色瘤胃球菌(Ruminococcusalbus)等是重要的纤维素降解菌[9]。除了纤维降解菌,瘤胃微生物优势菌还包括能够将植物性蛋白以及非蛋白氮转化为微生物蛋白供宿主利用的蛋白质降解菌,如Ruminobacteramylophilus、Eubacteriumrumination、Streptococcusbovins、Bacteroideruminicola等[10]。出自Lactobacillus、Streptococcus、Enterococcus和Pediococcus菌属的乳酸相关菌[10],以及通过氢转移与其他瘤胃微生物合作维持瘤胃pH稳定的产甲烷菌[3]等在调节宿主消化系统和瘤胃内环境方面起重要作用。某些低丰度菌群在功能上也需要引起重视。纤维杆菌门的产琥珀酸丝状杆菌(Fibrobactersuccinogenes)仅占细菌多样性的0.5%,然而在大多数核心微生物组研究中均有报道[11-12]。Fibrobactersuccinogenes以纤维素为底物发酵产生的可溶性糖和琥珀酸盐能够促进其他微生物组的代谢,因而被认为是一个关键的纤维降解物种[13]。古菌域通常在瘤胃中的占比较小,仅占瘤胃微生物总丰度的1.7%,其中包括Methanosarcina、Methanomicrobium、Methanobrevibacter、Methanobacterium等产甲烷菌[10],但产甲烷菌仍然普遍存在于反刍动物瘤胃中,因此属于反刍动物核心菌群的一部分[12]。
前人采用传统的分离培养方法分离获得了部分瘤胃可培养菌株[14],如RuminococcusflavefaciensWQ-1[15]。Hungate1000库中保藏有410株瘤胃来源的微生物,覆盖了82个瘤胃菌属[3]。此外,包括美国ATCC菌种保藏中心、哥德堡大学CCUG菌株保藏中心、德国DSMZ保藏中心、日本JCM菌株保藏中心、比利时BCCM菌种保藏中心、英国NCTC菌种保藏中心在内的国际菌种保藏中心共保藏了117种瘤胃微生物纯培养物[16]。Liu等[17]通过原位培养法分离出包括Pseudomonasstutzeri、Proteuspenneri、Klebsiellapneumoniae等在内的12种28株尿素分解菌,为进一步探索微生物参与瘤胃氮代谢提供有效帮助。de Oliveira等[18]从瘤胃中分离出可产生抗菌肽的链球菌,并通过比较基因组学表征了它们的抗菌活性以及与瘤胃环境适应相关的遗传特征。尽管如此,瘤胃微生物的分离培养尚处于探索阶段,能够纯化培养的菌种占比较小,因此需要借助现代培养组学等技术手段加大瘤胃功能菌群分离培养和鉴定的工作力度。
2 培养组学的技术发展概况
培养组学是利用多种培养条件进行细菌培养,并通过MALDI-TOF和16S rRNA鉴定物种的新方法。培养组学具有并行化、可视化、活菌保藏等优势,同时也有筛选不确定性高、培养条件复杂等缺点,如图1所示。作为一种研究复杂微生物生态系统的全新方法,培养组学在传统微生物培养的基础上开创了新的试验方法和研究技术,使其具有高度并行和高通量的特点,因而被赋予了“组学”地位。此前,受限于培养条件和培养技术的滞后,大多数微生物的分离培养并未成功,人们普遍认为超过80%的未分类的细菌无法被分离培养,直至Lagier等[19-20]引入培养组学技术,使得细菌物种的分离培养和基因组测序成为可能。Lagier等[21]采用212种不同的培养条件成功从人类肠道获得1 057种细菌,包括334种已知但未被分离培养的细菌和197个潜在新物种。培养组学微生物分离和培养的技术主要采取了以下两种方式:1)通过基于液滴的微流体将水相中的游离细胞分装在油乳液中;2)在物理微孔或微阀的小型阵列中对单个微生物进行基于有限空间的分区。
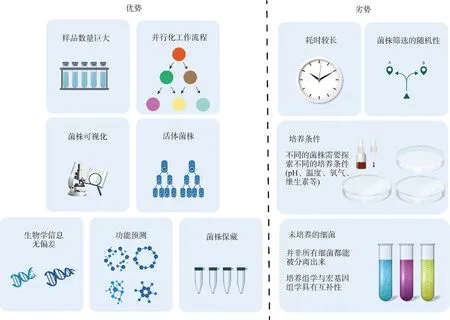
图1 培养组学的优、缺点Fig.1 The strengths and weaknesses of culturomics
2.1 基于液滴微流体的培养组学技术
基于液滴的微流体依靠的是两相微通道流体捕捉分散在球形颗粒中的单个细胞或分子,这些颗粒由生物相容性油包裹,由于其在高通量、并行化、标准化和动态过程控制方面的优势,该技术被视为单细胞微生物分离培养工作流程的关键组成部分[22]。然而,细胞的分离过程符合泊松分布,每个微液滴包裹的细胞数量无法准确预知,只有部分微液滴仅包含单细胞,而更多的微液滴是空的或包含两个及以上数量的细胞[23-24]。目前,提高分离单细胞的概率通常是通过样品的无限稀释来实现的。
瘤胃、肠道等厌氧生态环境中存活的微生物在利用微流体分离、培养、分析单个微生物细胞时会面临额外的挑战,即样品处理、生长检测和微流控分选需要在无氧条件下进行。由于与流式细胞仪或分选装置耦合的微流控管道难以集成到标准厌氧工作站中,目前已开发了一些厌氧条件下微生物生长监测的替代方法。例如,微流控条纹板(microfluidic streak plate,MSP)将微流控与条纹板技术相结合,微流控产生的液滴由均匀的油阻隔开,并允许手动或自动将其移至培养皿中培养[25]。MSP在好氧和厌氧条件下均能从食木白蚁肠道微生物组中分离出单细胞细菌[26]。最近,MSP单细胞培养被整合到高通量功能筛选工作流程中,如咪唑啉酮降解细菌的定向选择[27]。
MicDrop将厌氧封装培养的单个细菌微流体液滴与Illumina MiniSeq测序相结合,实现间接非连续生长监测,进而实现厌氧环境下的微生物培养和鉴定[28]。其原理是将产生的16S rDNA序列变体(sequence variant,SV)在含有相同细菌分类群的微液滴之间共享,通过16S rDNA测序和qPCR数据在SV水平拟合生长曲线,可以在厌氧工作流程之外跟踪单个物种的生长。MicDrop的高通量培养可以进行功能性筛选,评估在选择性培养条件下厌氧富集的微生物的生长动力、分类学特性和微液滴中的单菌丰度。Watterson等[29]开发了一种基于液滴的微流控系统,并且集成了显微检测,将分离、培养和分选三种基本功能结合在一起,通过设定不同的阈值标准,可以将单个细菌扩繁而成的菌落进行选择性地收集和传代,从而进行分类和功能表征。
相较于厌氧培养,有氧的工作条件可以实现空间要求更高的复杂微流控设置,以进行深入的功能筛选。在这方面的创新型进展之一是将生物相容性双水包油乳化液滴微流控技术(microfluidic double water-in-oil-in-water emulsion droplet technology,MDE)和荧光激活细胞分选(fluorescence-activated cell sorting,FACS)相结合,以研究荧光标记的目标菌株和潜在功能菌株之间单细胞水平的微生物相互作用[30]。Terekhov等[31]通过MDE-FACS将绿色荧光蛋白标记的金黄色葡萄球菌与微生物菌群混悬液共包封,鉴定了具有潜在抗金黄色葡萄球菌生物活性的代谢物。
2.2 基于分区的微列阵培养组学技术
基于分区的培养组学技术的主要优势是能够提供更大的微生物培养空间,可以将单细胞扩展到更大体积的高密度微菌落。与微流控相比,从离散的微室中分离选定的微菌落通常更直接。而原位培养概念的引入使得基于分区的微生物培养得以进一步发展。原位培养是通过模拟自然环境,提供微生物生存的化学成分的微生物培养方法。该方法可以最大限度的还原微生物原位生态环境,简化人工微生物培养条件的探索。原位培养法使用了选择通过性膜以允许微生物与其原栖息环境进行有限的接触,纳入其必需生长因子的同时实现微生物之间的隔绝,最终富集形成理想的纯化菌落。此外,目标微生物产生的生长抑制代谢物可以经选择性通过屏障结构进行自由扩散,避免了局部聚集导致的培养环境恶化[32]。Liu等[17]使用透析袋作为选择通过性膜将细菌进行瘤胃液包埋实现原位培养,成功分离出28株含有脲酶基因的细菌。基于原位培养法的原理,Kaeberlein等[33]将扩散室培养引入微生物研究用于培养“不可培养”的微生物,并成功获得2株菌株。然而,扩散室培养仍无法实现有效的高通量微生物培养,极大限制了其适用性。Nichols等[34]研究并设计了由数百个微室构成的隔离芯片(iChip),可将单个微生物细胞装入微室实现高通量的特异性富集培养。滑移芯片(slipchip)是两块由单细胞微室和流体导管组成的微流控芯片嵌合而成的,可用于消化道厌氧菌的培养[35]。滑移芯片可以物理切割微生物群落,一部分用于16S rRNA测序,另一部分用于目标微生物的保存和扩繁[35]。
除了需要分离培养微生物之外,人们逐渐开始对捕捉单个微生物在传代过程中的进化产生兴趣。单细胞分离延时成像技术(single-cell isolation following timelapse imaging,SIFT)通过多代延时显微镜追踪空间受限活细胞,该技术能够揭示目标微生物的生理状况和其对非生物刺激反馈的基本信息[36]。在两层SIFT微流控芯片中,第一个流动层用于细胞分离,而第二层集成了一系列微流控阀门、流动通道和用于收集芯片外单细胞的光镊[36]。目前,SIFT仅应用于单个细菌的无损多代成像和追踪以及分离和扩繁培养,但该技术也可用于高通量分离和筛选动物微生物[36]。
3 培养组学在瘤胃微生物培养中的应用现状
当前培养组学在瘤胃微生物的研究中应用较少,但已有的研究已经在一定程度上揭示了其在缩小瘤胃菌群物种未知范围、探知瘤胃微生物功能等方面的巨大潜力。瘤胃微生物基因组学网络(Rumen Microbial Genomics Network)在全球研究联盟家畜研究小组(Livestock Research Group of the Global Research Alliance)的支持下成立,并将生成瘤胃微生物基因组目录Hungate1000作为主要目标以期推动反刍动物的高效生产并减少温室气体排放[4]。Hungate1000目录中的微生物覆盖9门48科82属,Seshadri等[4]选择410株可培养的瘤胃微生物进行基因组测序,并与91株公开的可培养菌株的基因组联合分析,构建了Hungate1000瘤胃微生物基因组目录(包括480株细菌和21株古菌)。在瘤胃中占据主导地位的瘤胃厚壁菌门(Firmicutes)和拟杆菌门(Bacteroidetes)贡献了Hungate1000基因组目录的大部分,其中Lachnospiraceae家族是最大的单一类群(32.3%);可培养的古菌主要来自Methanobrevibacter属和Methanomassiliicoccales目[4]。Hungate1000中大多数可培养的微生物来自于牛(70.9%)或羊(17.6%),其余可培养微生物则来自于包括骆驼在内的其他反刍动物。de Oliveira等[18]从牛瘤胃中分离到463株细菌,其中5株细菌含有高度保守的生物合成基因簇,与瘤胃链球菌中抗菌肽的产生有关。Liu等[17]通过原位培养法模拟瘤胃环境,从瘤胃微生物菌群中分离出404株细菌,测序后发现28株具有脲酶基因的新物种在能量代谢与氮代谢方面发挥重要作用。然而,仍有已被基因组测序探知的瘤胃微生物尚未被分离培养[4]。Zehavi等[16]研究发现,培养基类型、样品稀释度、系统发育等多种因素能够影响瘤胃微生物的可培养性,其中样品稀释能够显着降低瘤胃微生物可培养性对相对丰度的依赖。另外,微生物的丰度和系统发育是决定瘤胃微生物可培养性的主要因素[16]。瘤胃微生物与多糖降解、蛋白质的水解与合成、纤维素的降解、瘤胃解毒等功能密切相关,因此基于培养组学的瘤胃微生物纯化培养能够分离某些关键功能的标志性菌株,为深入了解瘤胃微生态系统提供新视角[1]。
4 培养组学在瘤胃微生物培养中的挑战
4.1 营养基质和生长条件的鉴定
反刍动物瘤胃中的古菌和部分细菌难以分离培养的事实早已形成共识。在纯化培养瘤胃微生物时,所需的营养基质可用于分离或富集特定菌株。然而,部分瘤胃微生物的培养对生长环境和生长因子(维生素、氨基酸、腐殖酸、无机元素等)要求极为严苛,因此在实验室中探索菌株生长条件具有一定挑战性。对于难以适应富营养环境的瘤胃微生物,可使用寡营养培养基进行分离培养[32]。此外,一些无机物如金属化合物、含硫化合物、含氮化合物参与了微生物生理代谢的微生态循环,但由于其浓度低于实验室检测极限,也成为了瘤胃微生物培养的重要阻碍。对于部分微生物而言,它们所需的宽泛的底物类别可从基因组序列中推断出来,但仍然无法精准确定每个底物类别中的所需物质。这种可变性在一定程度上反映了基于底物基质和生长条件分离培养微生物的痛点和难点。
4.2 休眠期微生物的复苏
瘤胃微生物种群在受到环境胁迫时,部分成员能够以存活为目的通过表型变异进入休眠[37-38]。虽然目前有大量关于微生物休眠的文献报道,但人们对其休眠与复苏的过渡机制仍知之甚少[37]。有研究表明,微生物休眠期的结束可能受到某些信号分子的影响,过程具有随机性[38]。此外,微生物可能已经进化出不同的机制来调节机体的休眠与复苏,这也使得瘤胃微生物分离培养变得更加复杂[39]。
4.3 菌群的共生关系
部分微生物群落成员之间通过直接或间接交换必要的生物分子形成种间依赖的共生关系。然而,人工分离培养微生物时往往会忽视这种共生的微生态关系,导致分离培养的目标单菌无法正常生长和存活。产氢细菌(H2-producing bacteria)和氢利用产甲烷菌(H2-consuming methanogens)是典型的共生微生物,Guzman等[40]使用电生物化学系统模拟产甲烷菌在共生培养中的氢气消耗,成功富集Syntrophomonaszehnderi,但仍不能使其从产甲烷菌中完全分离出来进行单菌纯化培养。此外,Diapherotrites、Parvarchaeota、Aenigmarchaeota、Nanohaloarchaeota、Nanoarchaeota(DPANN)古菌和Candidate phyla radiation(CPR)细菌的基因组较小,通常以未分离的小细胞共生体的形式被检测到,它们主要以共生形式或寄生形式依赖于其他宿主微生物存活[39]。因此,以组合的方式进行微生物分选,可能是突破微生物分离培养瓶颈的契机。
4.4 菌群的竞争关系
在瘤胃复杂的微生物群落中,高丰度微生物对生长环境的广泛适应性提高了其分离及纯化培养的成功率[41]。菌群的竞争性抑制使分离低丰度瘤胃微生物的过程更加复杂。目前常用的分离培养低丰度微生物的方法是利用基因组测序技术预测目标微生物生长的最佳环境,然后对其进行纯化培养。然而,即使低丰度微生物被成功分离,在随后的富集试验中,它们仍有可能被生长速率更快的其他微生物竞争性地取代,导致培养失败。近年来,从样本中分离单个微生物并接种到培养基进行培养已成为分离培养低丰度微生物的有效方法[41-43]。
5 培养组学在瘤胃微生物培养中的展望
培养组学能够增加已知微生物的种类和数量,填补微生物学未知领域的空白。Zehavi等[16]通过研究培养基类型、样品稀释度、系统发育等因素与培养瘤胃微生物之间的关系,发现约23%瘤胃微生物是潜在可培养的且主要来源于低丰度微生物种群。迄今为止,反刍动物瘤胃微生物的多样性研究尚不充分,培养组学的出现可为未知瘤胃微生物的分离鉴定提供新方法。
培养组学不仅能够拓展微生物种质资源,而且可以校正特定环境下微生物群的组成结构及菌群丰度。宿主生理健康及疾病与消化道菌群组成的变化密切相关,可通过移植培养组学分离的菌株来调控宿主菌群结构进而发挥重塑菌群稳态的重要作用,以期维护肠道屏障,改善肠道健康。鉴于微生物培养组学在恢复菌群平衡和校正微生物组成的潜在作用,分离培养与疾病相关的关键微生物可能会对特定环境下的微生物对话机制提供见解。
抗生素可以广泛用于由致病菌感染引起的疾病治疗,其主要来源是细菌,因此利用培养组学深入挖掘细菌基因库与参考菌株库对新型抗生素的发现具有重要作用[44]。研究表明,反刍动物瘤胃中存在部分能够分泌抗菌肽的功能性菌株,如StreptococcilutetiensisUFV09,StreptococcilutetiensisUFV11,StreptococcilutetiensisUFV58等,分离培养瘤胃细菌可能是开发新型抗菌肽的有效途径[18]。因此,培养组学提供的广泛的未知微生物类群和每个物种的多个菌株,可以作为新型抗生素的潜在来源为治疗细菌感染性疾病开拓新思路。
6 小 结
培养组学技术在瘤胃微生物分离培养中的应用为扩充瘤胃菌群数据库、探索瘤胃微生物的组成和功能,以及后续通过调节瘤胃微生物来改善瘤胃健康带来了新机遇。然而,瘤胃微生物培养组学尚处于起步阶段,仍面临诸多挑战,包括培养条件严苛、生存/竞争关系复杂、休眠复苏机理不清等,需要进一步研究以突破相关技术瓶颈。未来建议灵活运用并持续优化培养组学技术方案,注重多组学技术的有机结合,丰富瘤胃微生物图谱、筛选标志菌株及产物,并实现转化应用。随着瘤胃微生物与宿主互作研究成为热点,瘤胃微生物培养组学具有广阔的应用前景。



