苏毅馨,李林潞,毛 昀,褚雪镭,陈 峥,朱世杰
(中国中医科学院望京医院肿瘤科,北京 100102)
食管癌是常见恶性肿瘤之一,每年造成40多万人死亡[1],2018年中国食管癌新发病例占全球新发病例54.1%,死亡病例占全球死亡病例56%[2]。食管鳞癌(ESCC)是主要的组织学亚型,占90%以上[3]。目前常规治疗方式包括手术、放疗、化疗、靶向治疗、免疫治疗等,但ESCC患者的5年生存率低于30%[4]。研究表明,相关基因及信号通路的改变可导致肿瘤细胞的早期转移及高侵袭性,如RTK/RAS/PI3K通路中TP53、 CCND1基因突变等[5],因此亟须进一步探讨ESCC分子机制,以期寻找ESCC早期诊断及靶向治疗潜在的生物标志物。
近年来,基因芯片技术及生物信息学已广泛应用于基因组学的研究,LU等[6]分析ESCC中DNA甲基化驱动基因,发现ABCD1、CCDC8等基因异常与患者生存预后相关。本研究通过整合公共基因芯片数据库(GEO)中GSE17251、GSE45670基因芯片数据集,利用GEO2R和Venn图在线工具获得两数据集中共同差异表达基因(DEGs),其次通过DAVID在线网站及R语言进行基因本体(GO)和基因组百科全书数据库(KEGG)分析并将其可视化,然后,通过SPRING在线工具及Cytoscape软件中MCODE(Molecular Complex Detection Technology)插件筛选出核心DEGs。最后将核心DEGs导入GEPIA(Gene Expression Profiling Interactive Analysis)在线数据库进行表达差异及预后分析获得与ESCC预后相关基因,并利用UALCAN在线数据库验证其在ESCC组织与正常组织表达差异性,探索ESCC预后的相关生物标志物。
1 材料与方法
1.1 ESCC数据收集及差异基因筛选
NCBI(National Center for Biotechnology Information)平台GEO数据库(http://www.ncbi.nlm.nih.gov/geo/)是公开的微阵列/基因图谱公共数据库,利用该数据库进行基因芯片筛选。目标芯片的准入标准:(1)临床ESCC患者标本,排除细胞株和动物实验;(2)入选芯片需含有ESCC组织标本和正常组织标本;(3)入选芯片标准为相同平台。
确定目标芯片后,利用在线工具GEO2R分析各个芯片数据,设置筛选标准为:|logFC|>2,P<0.05,然后利用Venn软件(http://bioinformatics.psb.ugent.be/webtools/Venn)进行在线检测,搜集DEGs。其中logFC<0为下调基因,而logFC>0为上调基因。
1.2 基因功能注释与通路富集分析
DAVID(Database for Annotation,Visualization and Integrated Discovery Database)生物信息资源数据库整合了生物数据和分析工具,能够对基因和蛋白质进行功能注释。通过DAVID进行在线分析,以人源基因为背景进行GO和KEGG对差异基因进行GO分析及KEGG信号通路富集分析,并利用R语言将其可视化。
1.3 蛋白互作网络分析
将DEGs导入在线STRING网站(https://string-db.org/cgi/input.pl)构建蛋白互作网络(PPI),然后运用Cytoscape3.6.0软件中MCODE插件检测核心基因,筛选标准:degree cutoff=2;node score cutoff=0.2;k-core=2;max.depth=100。
1.4 核心基因验证与预后的关系
通过GEO数据库的挖掘,明确差异表达的核心基因,利用GEPIA(http://gepia.cancer-pku.cn/)分析核心基因表达差异性及预后相关性,筛选条件P<0.05;其次利用UALCAN在线工具(http://ualcan.path.uab.edu/)进行验证。
2 结 果
2.1 差异表达基因
根据纳入标准,筛选出两个符合要求的微阵列数据集,分别为GSE17251、GSE45670 (https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi),均来自GPL570平台。GSE17251包含5例ESCC组织标本和5例正常食管组织标本,GSE45670包含28例ESCC组织标本和10例正常食管组织标本,共得到33例ESCC组织及15例正常组织。利用GEO2R分析从GSE17251、GSE45670芯片中分别得到差异基因161、1 087个,其中上调基因分别为81、403个,下调基因为80、684个。利用Venn软件发现2个数据集共表达的差异基因有86个,其中54个为高表达基因和32个低表达基因(图1、表1)。

A:所有差异表达基因;B:上调差异表达基因;C:下调差异表达基因。

表1 86个差异表达基因上调基因及下调基因
2.2 差异表达基因GO功能注释分析及KEGG通路富集分析
依据基因编码的蛋白质在细胞中的作用,GO分析将DEGs功能注释的结果分为3类:生物过程(BP)、细胞组分(CC)和分子功能(MF)。将差异基因进行GO分析,筛选为P<0.05的结果(表2、图2)。表明在生物过程中,主要富集在细胞外基质结构组成、蛋白激酶活性的激活、细胞增殖的调控、内皮细胞分化、细胞有丝分裂、有丝分裂中期/后期转变的调节、胰岛素样生长因子受体信号通路的正向调控、磷酸化的正调控;在细胞组分中,包括纺锤体中央区、核质、Ndc80复合物、驱动蛋白复合物、轴突丘;在分子功能中,包括ATP结合、DNA结合、转录激活性、RNA聚合酶核心启动子近端区序列特异性结合、转录调节区DNA结合等。

表2 ESCC差异表达基因GO富集分析
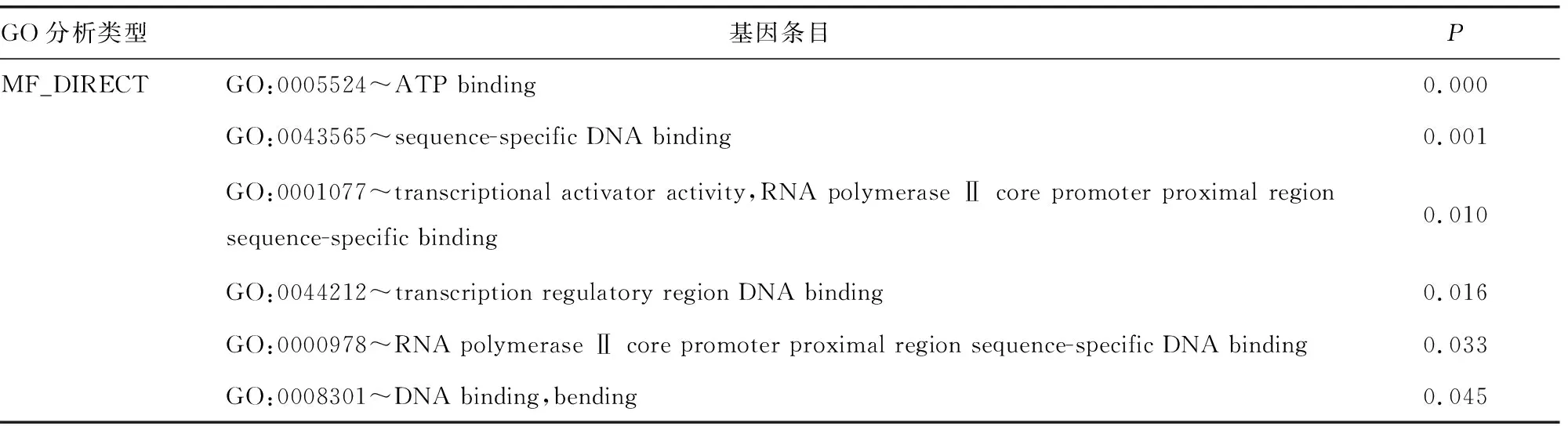
续表2 ESCC差异表达基因GO富集分析

图2 GO功能富集分析结果
通过对肿瘤组织和正常ESCC组织的差异进行进行KEGG通路富集分析并利用R语言将其可视化(图3),结果表明:主要集中在25条信号转导通路上,包括ECM受体结合、蛋白质消化、TGF-β信号通路、卵母细胞减数分裂、血管平滑肌收缩、癌症转录失调、PI3K-AKT信号通路、小细胞肺癌等。

图3 KEGG通路富集分析
2.3 蛋白互作网络可视化
将86个差异基因导入到STRING网站构建蛋白互作网络(图4),剔除孤立节点后,运用Cytoscape 3.6.0软件中MCODE插件按照筛选核心基因,筛选到27个关键核心基因(图5)。
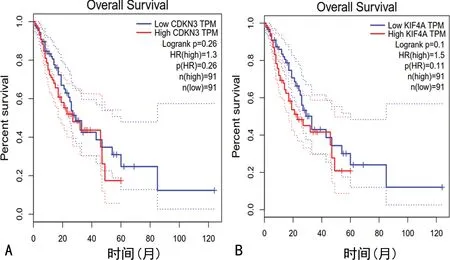
A:CDKN3对ESCC患者OS影响;B:KIF4A对ESCC患者OS影响。
2.4 核心基因验证与预后的关系
通过GEO数据库的挖掘,明确差异表达的核心基因27个,利用GEPIA筛选出预后相关基因结果表明:CKDN3、KIF4A基因在ESCC组织中高表达(图6),并且高表达组的总生存期明显短于低表达组,差异有统计学意义(P<0.05),见图7,经UALCAN验证结果一致(图8)。
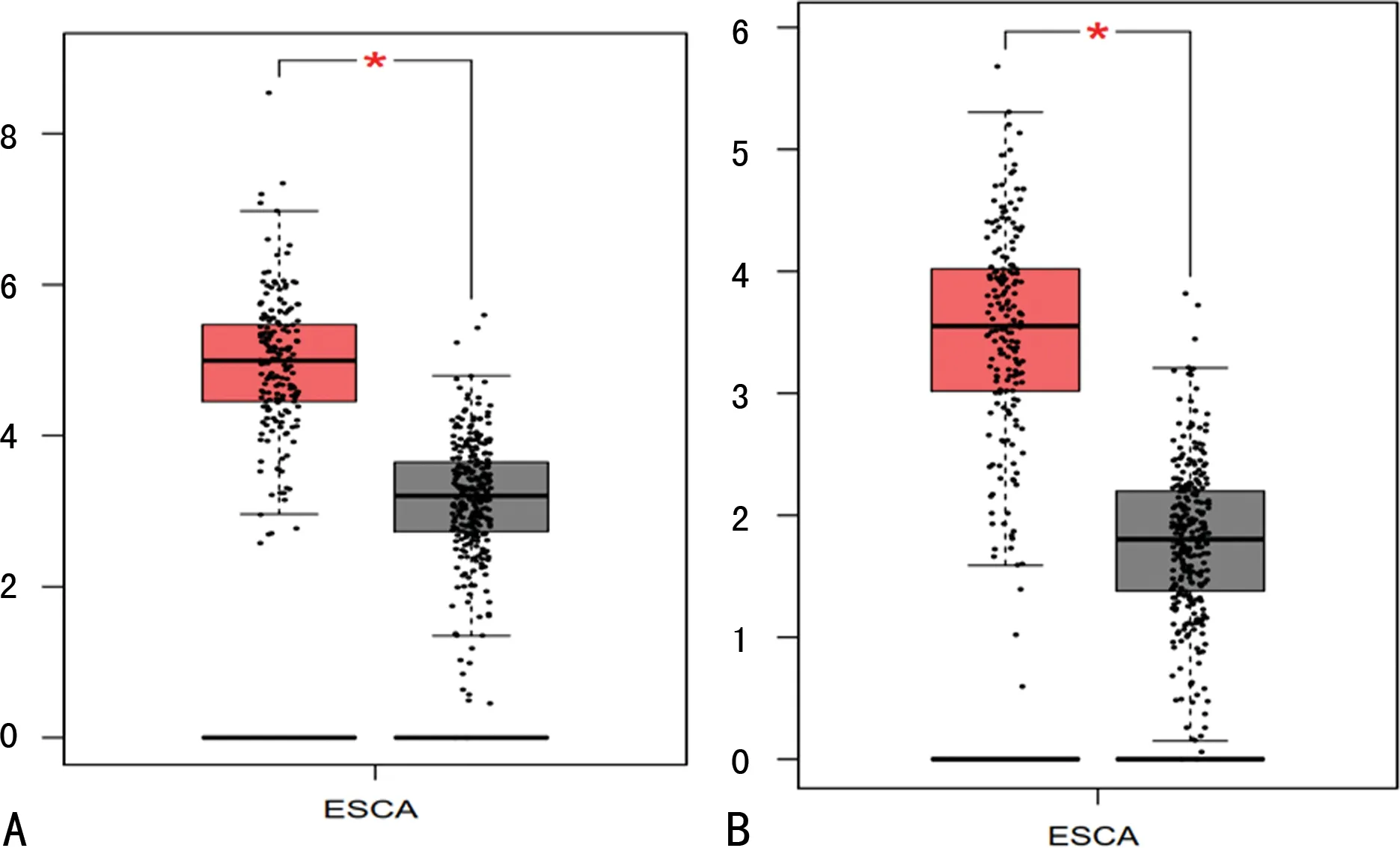
A:CDKN3在ESCC患者高表达;B:KIF4A在ESCC患者高表达。
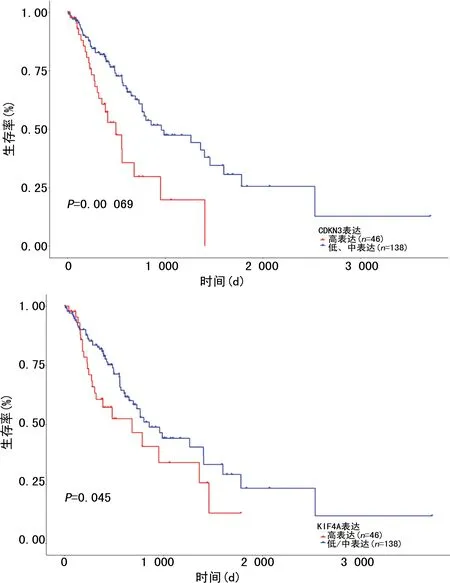
A:UALCAN验证CKDN3在ESCC患者高表达;B:UALCAN验证KIF4A在ESCC患者高表达。
3 讨 论
食管癌是消化系统最常见的恶性肿瘤之一,具有病死率高及预后差等特点,顺铂和5-氟尿嘧啶(5-FU) 的标准化疗方案中位生存时间为201.5 d,1年生存率为27.8%[7]。近年来,伴随测序技术的进步,基因图谱和基因芯片在科研领域得到了广泛的应用,促进了对包括ESCC在内的肿瘤异质性理解,并为识别新的癌症基因和预后生物标志物提供一个强有力的方法[8]。如研究表明CCND1、CTTN、EGFR、TP63和CDKN2A[9]与ESCC密切相关,ANO1可能与ESCC的预后生物标志物[10]。但目前ESCC发病的分子机制尚未明确,迫切需要找到可用的潜在生物标志物,生物信息学可帮助探索基因层面发生的变化、识别潜在的生物标志物。
本研究从GEO数据库中筛选出GSE17251和GSE45670两个芯片数据集,共纳入33例ESCC组织及15例正常食管组织。通过GEO2R和Venn软件发现86个共有 DEG,包括54个上调DEGs和32个下调DEGs。在GO分析及KEGG分析中,主要富集在细胞增殖的调控、细胞周期、细胞分化、DNA复制、PI3K-Akt信号通路、转化生长因子-β(TGF-β)信号通路、卵母细胞减数分裂等方面。食管鳞状细胞癌的演变是一个多步骤的过程,细胞损伤的累积可导致细胞增殖异常及基因不稳定性,细胞周期失控、细胞分化异常是恶性肿瘤的标志,在肿瘤的致癌或进展过程中发挥重要作用,如TP53、CDKN2A基因突变与早期食管肿瘤细胞分化相关[11],NF750和NOTCH1的突变可影响食管鳞状细胞的成熟导致癌变[12],HERG1基因可通过影响PI3K/AKT信号通路促进ESCC细胞增殖、迁移和侵袭[13]。
通过SPRING及Cytoscape3.6.0软件插件构建DEGs的蛋白互作网络图,发现27个高表达的核心基因,经GEPIA分析并通过UALCAN验证CKDN3、KIF4A在ESCC组织的高表达提示预后状态不良。CDKN3基因属于 CDC14s家族,位于染色体位置14q22,包含21个氨基酸,相对分子质量23×103,是一种双特异性磷酸酶蛋白,可对磷酸化丝氨酸/苏氨酸发挥去磷酸化作用调控细胞周期进程,既往在多种肿瘤中报道,不同类型肿瘤组织中发挥不同作用,不仅参与细胞周期调控,对细胞凋亡及侵袭迁移能力也有影响[14]。其作为促癌基因在肺癌、宫颈癌、肝癌中CDKN3高表达提示预后不良[15],在诊断方面,CDKN3高表达识别宫颈癌组织的灵敏度达93%,特异度达96%[16],但在ESCC中的生物学功能尚不清楚。YU等[17]研究报道CDKN3在ESCC细胞系中表达上调,通过激活ESCC细胞的AKT信号通路促进细胞增殖和侵袭。CDKN3敲除可降低ESCC细胞的增殖、侵袭和迁移能力,抑制细胞G1/S期转化,与LIU等[16]实验结果一致,其可通过AKT-p53-p21通路促进ESCC细胞增殖侵袭。KIF4A属于驱动蛋白家族4(KIF4),参与纺锤体组织、染色体排列,在细胞有丝分裂、DNA损伤修复、肿瘤发生、发展发挥重要的作用,在乳腺癌、肺癌、肝癌[18-20]等多种肿瘤组织中高表达,并可作为非小细胞肺癌、乳腺癌的预后因素[18-19],FOXM1通过调节KIF4A的表达促进肝细胞癌的进展[20],但缺乏ESCC中的生物学功能研究。
综上所述,本研究通过生物信息学分析在不同的微阵列数据集的基础上识别出ESCC组织和正常食管组织之间的两个DEGs(CKDN3、KIF4A)在ESCC的发生、转移中作用,在基因层面为寻找新的分子靶点提供了一定的支持,也为实现ESCC的精准治疗提供了一个新思路,但还需进一步进行实验以验正相关结果。



