梁俐兰,刘莉莉,覃超梅,梁 斌,石明连,刘永明,苏何玲
(桂林医学院生物化学与分子生物学教研室,广西桂林 541100)
乙型肝炎病毒(hepatitis B virus,HBV)感染引起的急、慢性肝炎,肝硬化和肝细胞癌(hepatocellular carcinoma,HCC)是全球性的主要卫生问题。全球慢性乙型肝炎患者约2.57亿人,每年约887 000人死于HBV感染导致的肝硬化和HCC。近年来,HBV引起的宿主微小RNA(micro RNA,miRNA)表达变化及其在肝脏疾病发生、发展中的作用已成为HBV感染相关疾病发病机制研究的热点[1-2]。miRNA是一类长度为19~25个核苷酸的非编码单链小分子RNA,可通过与靶基因mRNA的3′非翻译区部分序列同源性结合而诱导基因沉默,在基因表达调控中发挥着重要作用。有研究表明,miRNA可影响HBV在宿主细胞内的复制和基因表达,而HBV则可通过调节宿主miRNA表达为其提供生存与复制的有利环境[3-6]。这种miRNA介导的HBV与宿主的相互关系是HBV感染相关疾病发病机制的重要基础。
miR-10b是新近被发现与多种疾病的发生、发展密切相关的一种疾病相关性miRNA,在一些病毒性疾病的发病机制中具有关键作用[7-9]。本研究分析美国生物技术信息中心(national Center for Biotechnology Information,NCBI)基因表达综合数据库中的GSE19980和GSE121248数据发现,miR-10b在稳定表达HBV的HepG2.2.15细胞中表达明显上调。进而提取HepG2.2.15细胞及其对照HepG2细胞的总RNA进行荧光定量聚合酶链反应(polymerase chain reaction,PCR)检测,证实了HBV对miR-10b表达的上调作用。在此基础上,对HBV上调miR-10b表达在肝脏疾病中的作用进行生物信息学分析,以期为研究miR-10b在HBV感染及其致病机制中的作用提供理论与实验依据。
1 材料与方法
1.1 实验材料
人肝癌细胞HepG2细胞株及稳定表达HBV的HepG2.2.15细胞均购自上海中国科学院细胞库。胰蛋白酶、高糖杜氏改良Eagle培养基(Dulbecco′s modified Eagle medium,DMEM)、青霉素链霉素及胎牛血清均购自美国Gibco公司;RNA提取试剂TRI Reagent购自美国Sigma公司;All-in-OneTMmiRNA qRT-PCR Detection试剂盒购自美国GeneCopoeia公司,反转录引物为试剂盒中的Oligo-dT adaptor primer, PCR反应所用特异性引物由上海生工生物工程公司合成。普通PCR仪及荧光定量PCR仪购自美国BIO-RAD公司。
1.2 方法
1.2.1miRNA芯片GEO2R分析
从NCBI基因表达综合数据库(https://www.ncbi.nlm.nih.gov/)下载miRNA表达芯片GSE19980。GSE19980 miRNA表达芯片数据集有12个样本,6个细胞株HepG2(样本编号:GSM499316~GSM499321)和6个表达HBV细胞株HepG2.2.15样本(样本编号:GSM499322~GSM499327)。采用GEO2R工具(https://www.ncbi.nlm.nih.gov/geo/geo2r/)对GSE19980数据进行分析,定义HepG2细胞为1组,HepG2.2.15细胞为2组,按P值从小到大排序,选取前250个具有显着表达差异的miRNA执行分析。将分析结果置于Echart(http://www.ehbio.com/ImageGP/)中制作火山图,筛选差异miRNA。
从NCBI下载GSE121248 mRNA表达芯片数据集,GSE121248数据集是以慢性乙型肝炎诱导的HCC及其邻近正常组织中分离出的组织,并提取总RNA进行Affymetrix基因微阵列分析芯片,共有107个样本,其中70个HCC样本(样本编号:GSM3428716~GSM3428785),37个邻近正常组织样本(样本编号:GSM3428786~GSM3428822)。采用GEO2R对GSE121248数据进行分析,定义70个HCC样本为1组,37个邻近正常组织样本为2组,按P值从小到大排序,选取P<0.001的差异表达基因进行后续分析。
1.2.2细胞培养
HepG2细胞和HepG2.2.15细胞采用含10%胎牛血清、1%青霉素链霉素溶液的高糖DMEM、37 ℃、5%二氧化碳培养箱中培养。
1.2.3miR-10b表达的荧光定量PCR检测
利用TRIzol提取细胞总RNA,Nanodrop 2000测量总RNA浓度及纯度。miRNA逆转录按All-in-OneTMmiRNA qRT-PCR Detection试剂盒说明书操作,合成的第1链cDNA稀释10倍后于Bio-Rad CFX96实时荧光定量PCR仪进行扩增。PCR条件为95 ℃ 5 s、63 ℃ 30 s、72 ℃ 30 s进 行35个循环。以U6作为内参。hsa-miR-10b上游引物序列:5′-ACACTCCAGCTGGGTACCCTGTAGAACC-3′,内参U6上游引物序列:5′-CTCGCTTCGGCAGCACA-3′,qPCR检测的通用下游引物为试剂盒中的Universal Adaptor PCR Primer。以2-ΔΔCt方法评估miR-10b的相对表达水平。
1.2.4miR-10b的靶基因预测
选择TargetScan (http://www.targetscan.org/vert_72/)、miRTarBase (http://mirtarbase.mbc.nctu.edu.tw/php/index.php)、miRDB (http://www.mirdb.org/)、DIANA TOOLS (http://diana.imis.athena-innovation.gr/DianaTools/index.php) 4种在线靶基因预测工具筛选出miR-10b的靶基因,使用在线工具Venny 2.1(https://bioinfogp.cnb.csic.es/tools/venny/index.html)绘制4个数据库预测靶基因的韦恩图,选取并集及交集靶基因用于后续分析。
1.2.5miR-10b靶基因的生物信息学分析
采用在线工具DAVID(https://david-d.ncifcrf.gov/)对miR-10b的交集靶基因进行基因本体(gene ontology,GO)分析,选择物种为Homo sapiens,背景也选择 Homo sapiens,包括生物过程、分子功能、细胞组分3种类型。以P<0.05为显着性阈值,富集分数为-lg P,采用在线工具STRING (https://string-db.org/)对miR-10b的交集靶基因进行KEGG通路富集分析。
1.2.6miR-10b靶基因PPI网络构建
将上述4个miRNA靶基因预测网站分析的miR-10b靶基因并集与GSE121248数据集GEO2R分析结果进行Venny分析以获得共同基因,并将结果上传在线工具STRING (https://string-db.org/),选择物种为 human species,蛋白质相互作用评分大于0.4执行PPI 网络分析,将PPI 网络分析结果导入Cystoscape,选取节点度大于10的蛋白与miR-10b的靶基因4个数据库预测交集结果进行Venny 2.1分析,获得核心蛋白用于下一步分析。
1.3 统计学处理

2 结 果
2.1 HBV感染上调miR-10b的表达
hsa-miR-10b在稳定表达HBV的HepG2.2.15细胞中表达明显上调,差异有统计学意义(P<0.05),见图1A。HepG2细胞miR-10b的相对表达水平[(0.980±0.079)]明显低于稳定表达HBV的HepG2.2.15细胞[(2.682±0.937)],差异有统计学意义(P<0.05),见图1B。
2.2 miR-10b基因位点与序列保守性
在miRBase数据库中检索人miR-10b的位置及其他物种miR-10b成熟序列,人miR-10b定位于2q31.1(chr2:176150303-176150412),其成熟序列为5′-UACCCUGUAGAACCGAAUUUGUG-3′。将10种其他物种miR-10b成熟序列与人类相比较发现,不同物种miR-10b具有高度保守性,见表1。提示 miR-10b具有潜在的重要生物学功能。
2.3 miR-10b的靶基因预测
使用Targetscan、miRTarBase、miRDB、DIANA TOOLS 4种在线工具预测miR-10b的靶基因,分别为340、325、352、464个,见图2。用Venny2.1软件绘制韦恩图筛选获得23个交集靶基因,见图3。作为后续分析的基因数据集见表2。
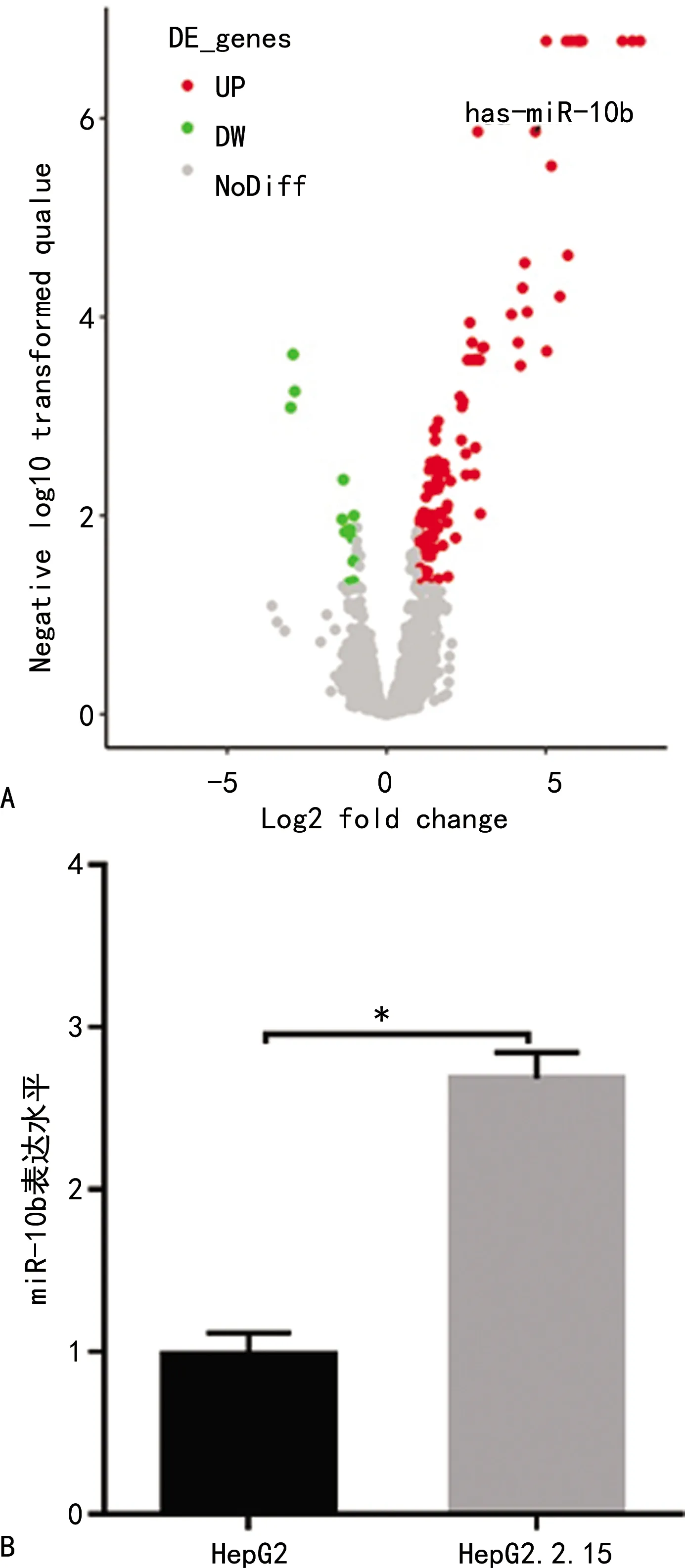
A:差异表达miRNA火山图;B:HepG2与HepG2.2.15细胞中miR-10b的表达水平比较;*:P<0.05,与HepG2.2.15细胞比较。
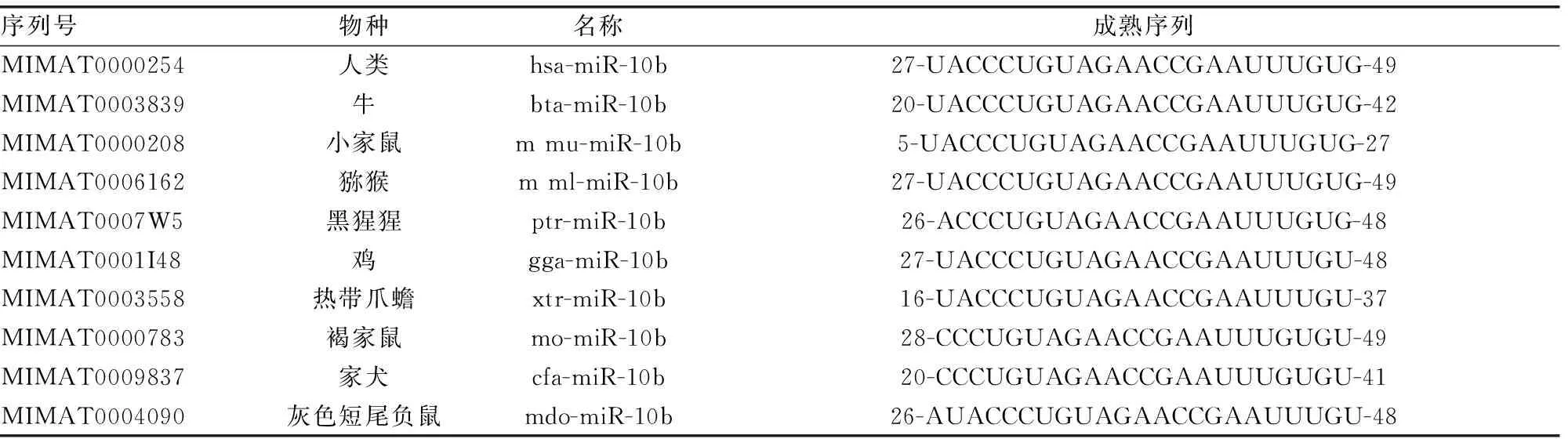
表1 不同物种的miR-10b的成熟序列
2.4 miR-10b靶基因数据集的GO及KEGG信号通路分析
miR-10b的23个靶基因参与了转录因子复合体、囊泡、核质部分等细胞组分;主要富集在转录调控、RNA代谢、基因表达等生物学过程,以及转录调控、转录因子活性和DNA结合等分子功能,见图3。miR-10b的靶基因明显富集在癌症中蛋白多糖、癌症中miRNA、昼夜节律及cAMP信号通路等相关通路中,差异均有统计学意义(P<0.05)。
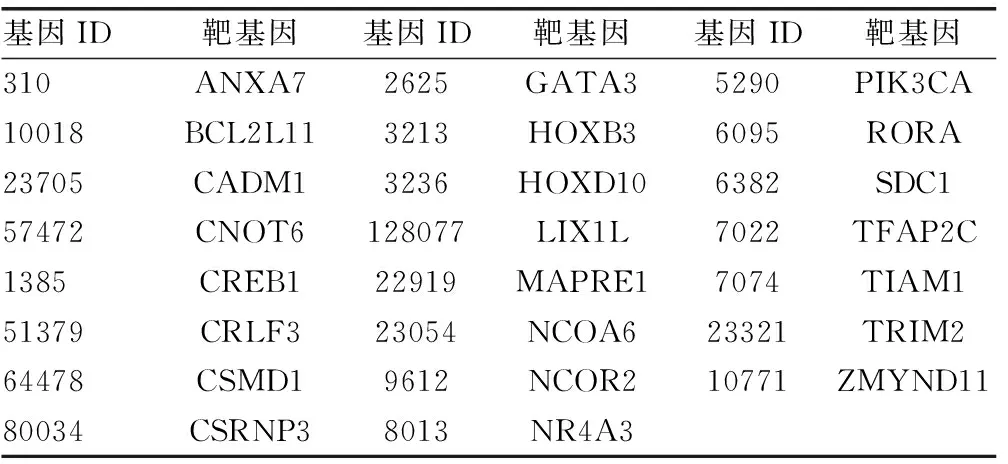
表2 交集的靶基因数据集
2.5 miR-10b靶基因编码蛋白的相互关系网络
miR-10b靶基因与慢性乙型肝炎诱导的HCC及其邻近的正常组织基因芯片GSE121248数据集GEO2R分析结果进行Venny分析获得573个共同基因,并将此573个基因上传在线工具STRING (https://string-db.org/),分析573个基因表达蛋白之间的相互作用,去掉无相互作用的蛋白质,获得501个相互作用的蛋白,将STRING分析结果导入Cystoscape后执行分析,选取节点度大于10共获93个蛋白基因,将此93个基因与miR-10b 4个网站预测23个交集的靶基因进行Venny 2.1分析获得CREB1、GATA3、NCOA6、NCOR2、PIK3CA、MAPRE1、TIAM1 7个核心蛋白基因。
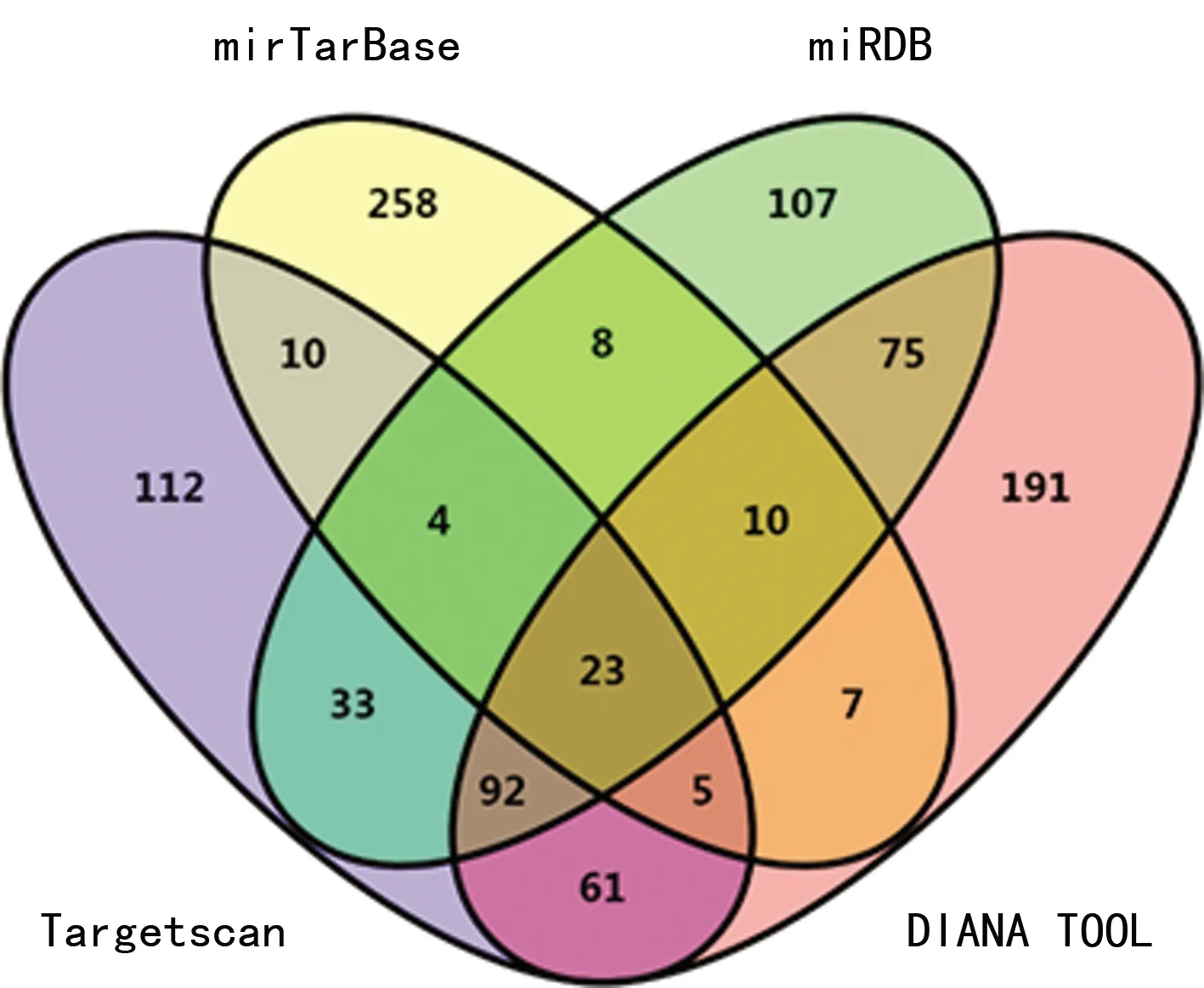
图2 miR-10b的靶基因预测
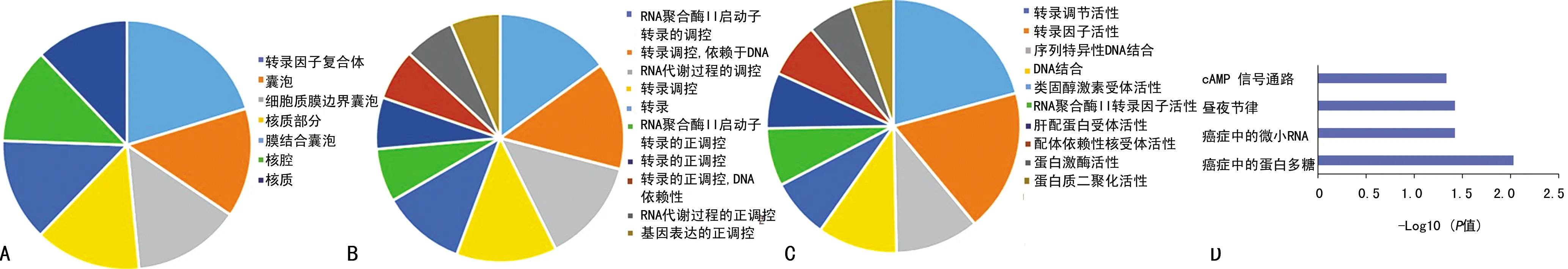
A:细胞组分分析;B:生物学过程分析;C:分子功能分析;D:KEGG pathway分析。
3 讨 论
尽管越来越多的研究表明,miR-10b在一些病毒性疾病的发病机制中具有关键作用,但其在HBV感染所致疾病中的作用仍鲜见文献报道。本研究通过分析NCBI数据库中的基因芯片数据发现,miR-10b在HBV感染肝细胞株HepG2.2.15中的表达明显升高。荧光定量PCR检测结果证实,HepG2.2.15细胞miR-10b的表达水平明显高于HepG2细胞。表明HBV感染可上调细胞miR-10b的表达。
MiR-10b在生物学方面具有重要功能。比对人类、小家鼠、猕猴、黑猩猩和牛等不同物种miR-10b序列的结果显示,其序列高度保守。实际上GO分析显示,miR-10b的靶基因一是生物过程方面与转录调控、RNA代谢和基因表达等相关;二是分子功能方面与转录调控、转录因子活性和DNA结合等相关;三是细胞组分方面与转录因子复合体、囊泡和核质部分等相关。KEGG信号通路分析提示,miR-10b的靶基因明显富集在癌症中蛋白多糖、癌症中miRNA、昼夜节律及cAMP信号通路等相关通路中。以GEO2R分析慢性乙型肝炎诱导的HCC及其邻近正常组织基因芯片GSE121248数据集差异表达基因与miR-10b靶基因进行Venny 2.1分析,获得了与HBV感染相关的miR-10b靶基因,进而STRING分析基因表达蛋白的相互作用确定了7个核心蛋白基因,分别为CREB1、NCOA6、NCOR2、PIK3CA、GATA3、MAPRE1和TIAM1。
CREB是正常细胞中许多重要信号传导通路的最终调节因子,在小鼠胰岛素分泌细胞INS-1中敲除CREB则上调caspase-3基因表达,促进细胞凋亡[10]。CREB可能通过诱导细胞保护因子——胰岛素受体底物-2(insulin receptor substrate-2,IRS-2)和神经元PAS结构域蛋白4(neuronal PAS domain protein 4,NPAS4)表达促进β细胞增殖[11]及存活[12-13]。此外,CREB可增强核心囊泡跨膜蛋白IA-2基因的表达促进胰岛素分泌[14]。NCOA6是一种转录共激活因子,NCOA6可调节胰腺β细胞中Nampt基因刺激胰岛素分泌[15]。磷酸肌醇-3激酶(phosphoinositide 3-kinase,PI3K)/磷脂酰3,4,5三磷酸(phosphatidylinositol 3,4,5-trisphosphate,PIP3)是胰岛素信号作用的重要信号途径[16-19],介导了所有细胞对胰岛素的反应。活化的PI3K亚基可解除糖原合成酶激酶3(glycogen synthase kinase 3,GSK-3)对糖原合成酶的抑制作用,促进肝糖原的合成,PI3K-C2γ敲除则导致实验小鼠肝脏糖原积累严重降低[20]。PI3K可通过促进细胞内囊泡中的葡萄糖转运蛋白4(glucose transporter 4,GLUT4)向细胞膜的转运,从而促进肌肉及脂肪组织葡萄糖的吸收[21-24]。miR-10b对CREB1和NCOA6的靶向作用可能降低胰岛β细胞的活性、下调胰岛素基因的表达及抑制胰岛素的分泌。miR-10b对PIK3CA的靶向作用可能会促进肝脏中糖原的分解,并阻止骨骼肌和脂肪组织中的葡萄糖摄取,从而导致机体糖稳态受损。这些研究结果显示,miR-10b参与了调节胰岛素分泌及葡萄糖摄取的生理过程。近年来,国内外研究表明,HBV感染可诱发2型糖尿病或增加2型糖尿病发病风险[25-26]。新近有研究发现,HBV感染与妊娠糖尿病相关,可增加妊娠糖尿病发病率[27-28]。慢性HBV感染合并非酒精性脂肪肝患者普遍具有胰岛素抵抗[29]。这些研究结果与miR-10b靶基因的生物信息学分析相吻合。miR-10b的其他靶基因编码蛋白如GATA3被报道在脂肪形成和炎症中具有重要作用[30]。新近有研究指出,其可作为胰岛素抵抗和2型糖尿病的潜在治疗靶标[31]。而miR-10b靶基因Tiam1和Mapre1编码蛋白TIAM1和MAPRE1分别被报道与肿瘤的侵袭性有关[32]和促进肝癌细胞的细胞周期进程[33]。
综上所述,HBV感染可上调细胞miR-10b的表达,而异常表达的miR-10b可能通过靶向沉默CREB1、NCOA6、NCOR2、PIK3CA、GATA3、MAPRE1、TIAM1等蛋白基因损害机体糖稳态调节,产生组织炎症及促进肿瘤发生、发展,从而在HBV感染的肝脏疾病中发挥着重要作用。因此,HBV感染对miR-10b表达的影响及其在肝脏疾病的作用值得深入研究。



