赵雪洋,李丹妮,王钰晨,郭 磊,王 丽,黄 杰,焦仪强,安小鹏,张希云, 张 磊*,宋宇轩*
(1.西北农林科技大学动物科技学院,杨凌 712100;2.永昌县畜牧兽医站,金昌 737200; 3.甘肃元生农牧科技有限公司,金昌 737100)
绵羊产羔性状是指母羊每胎次的产羔数,它是衡量绵羊繁殖力的重要指标[1]。提高绵羊产羔数可降低饲养成本,提高生产效益,促进绵羊养殖业的高效发展。绵羊产羔性状受多种因素的影响,包括遗传因素和环境因素[2-3]。遗传因素主要是指不同品种间的产羔数差异,环境因素主要是指饲养管理、配种控制、疾病防治等方面[4-7]。为了提高绵羊产羔性状,需要采取多种措施,包括优化饲料营养、调整配种时间、加强孕期护理等[8]。此外,利用分子遗传技术对绵羊产羔数相关基因进行挖掘和标记辅助选择,也是一种有效的方法[9-10]。
全基因组关联分析(genome wide association study,GWAS)是一种常用的分子遗传学方法,旨在鉴定复杂性状的遗传基础。通过GWAS分析,研究者可以深入了解绵羊的遗传特征和种群结构,发现与性状相关的基因和变异,为绵羊育种和遗传改良提供重要的信息和指导[11-14]。近年来,GWAS已成为提高绵羊繁殖性能的有效方法之一[15]。可以在全基因组范围内鉴定与繁殖性能相关的基因,进而深入了解繁殖性状的遗传机制和调控网络[10,16-18]。这些发现有望为改良和优化绵羊繁殖性能提供理论指导和科学依据。同时,GWAS还可以帮助筛选和鉴定出对繁殖性能有积极影响的遗传变异,进而为选育高产优质绵羊提供有力的选育手段[19]。总的来说,GWAS已经成为绵羊育种中不可或缺的工具之一。随着技术的不断进步和方法的不断创新,GWAS在绵羊育种中的应用前景将更加广阔。
现代数量遗传学在动物育种实践中的应用取得了巨大成功,如今它对一些遗传力高且呈连续性正态分布的数量性状仍然是必不可少的选择方法。但对于低遗传力性状,如母畜的产仔数等,选择反应并不理想。因此,可以从分子遗传水平上找到性状的遗传差异或与数量性状连锁的遗传标记,从而实现真正的基因型选择[20-22]。
本试验以东湖杂交奶绵羊为研究对象,对168只东湖杂交奶绵羊进行高深度全基因组重测序,再根据全基因组关联分析技术确定与产羔性状相关的关键基因,最后挑选关键基因的变异位点采用KASP分型技术进行SNP分型检测,并将其与产羔性状进行关联分析,探究其潜在的育种价值。
1 材料与方法
1.1 试验材料
试验动物来自西北农林科技大学金昌奶绵羊试验示范基地。选择体况良好、平均年龄一岁半的东湖级杂二代母羊748只,颈静脉采集5 mL血液于抗凝管中,并于-80 ℃保存。其中168只进行全基因组重测序,580只进行SNP分型检测并统计试验羊群不同胎次的产羔数据。
1.2 DNA提取与建库测序
本研究通过标准苯-酚氯仿法提取基因组DNA。将检测合格的DNA样品用超声波破碎仪打断至350 bp大小,再经末端修复、加A尾、加测序接头、纯化和PCR扩增等步骤进行文库的制备。文库检测合格后,使用Illumina高通量测序平台Nova-Seq 6000进行测序,生成150 bp的成对末端reads,共获得47 616 147个原始位点。
1.3 质量控制与变异检测
本研究利用fastp软件对原始的fastq格式文件进行过滤及质控。质控后的数据使用BWA-MEN对比到Oar_rambouillet_v1.0上,重复reads使用Picard软件进行排序。采用genome analysis toolkit (GATK, version 3.8)提取基因组高质量变异,获取变异数据的VCF集合。使用GATK进行过滤(参数:QD<2.0, FS>60.0, MQ<40.0, MQRankSum<-12.5, ReadPosRankSum<-8.0 and SOR>3.0)。使用GATK软件的SelectVariants模块分别提取SNP和InDel数据集合(参数:-restrict-alleles-to BIALLELIC,--remove-unused-alternates)。为了降低结果的假阳性,本研究将PLINK 1.9软件[23]获取的主成分分析结果作为协变量加入,此外还根据PLINK--missing 0.01参数和—maf 0.01参数进行了SNP位点的过滤,共得到27 750 039个位点。
1.4 全基因组关联分析
先使用GEMMA的-gk 2参数生成亲缘关系矩阵,然后结合生成的亲缘关系矩阵使用混合线性模型(MLM)进行GWAS分析(参数:gemma-bfile-k-lmm 1-p)。MLM模型结合了固定效应(配种方式和产羔季节)和随机效应。校正阈值线的确定为0.05/n(总位点数)。根据输出结果,后续使用R语言包CMplot进行曼哈顿图和QQ(Quantiles-Quantiles)图的绘制。
1.5 显着位点的注释
使用SnpEff软件构建绵羊参考基因组的本地注释数据库,并使用该软件对本研究构建的VCF集合进行注释,使用自己编写的perl脚本从注释后的VCF集合中提取注释信息。
1.6 注释基因的富集分析
本研究中使用北京大学构建的KOBAS 3.0数据库(http://bioinfo.org/kobas/retrieve/?taskid=5f7a3a18b1de4e53a517ffc7c7c86cfb)对注释得到的基因进行富集分析,以探究GWAS显着信号区域的基因在生物学网络中的作用和相互作用。
1.7 连锁不平衡分析
使用LDBlockShow软件进行连锁不平衡热图的绘制,突出显着性最强SNPs周边的连锁区域。
1.8 基因型检测
从NCBI(national center for biotechnology information)获取GRID2的序列信息(NC_040257.1),根据g.35685218A>G,和g.35685499C>T两个突变的位置,分别设计两条不同的上游引物和一条相同的下游引物。使用KASP进行特异性识别SNP基因型。
1.9 数据统计与分析
根据等位基因频率、基因型频率、纯合度和杂合度等参数的计算原理,自行编写Excel函数进行群体遗传学参数的计算,同时检验群体是否符合哈代-温伯格平衡原理。使用SPSS 20.0软件分析基因型与产羔性状之间的关系。线性分析模型为Y=μ+a+b+c+e,其中Y为性状的观察值,μ为均值,a为年季节效应,b为基因型固定效应,c为配种方式固定效应,e为随机误差。
2 结 果
2.1 全基因组关联分析结果
使用GEMMA软件的混合线性模型进行全基因组SNP的关联分析,设置显着性水平的校正阈值线为1.80×10-9,共检测到1 163个达到全基因组显着性水平的SNPs位点,所有达到显着性水平的位点均分布于绵羊6号染色体上,如图1A所示。通过SnpEff对所有显着位点进行注释,其中位于外显子区域4个,基因间区708个,内含子区域445个,其他区域6个。注释到与产羔性状相关的基因有BMPR1B、PDLIM5、PDHA2、UNC5C、GRID2、NFKB1、TSPAN5、STPG2、PPP3CA等。其中处于显着性前十的SNPs注释到了BMPR1B、GRID2、PDLIM5、PDHA2基因。
基于GATK提取的InDel集合利用GEMMA混合线性模型进行全基因组InDel关联分析,与SNP的结果相比,信号杂乱(图2),不具有较好参考价值,这也符合前人研究中多胎性状的主效位点主要为SNP的结果。
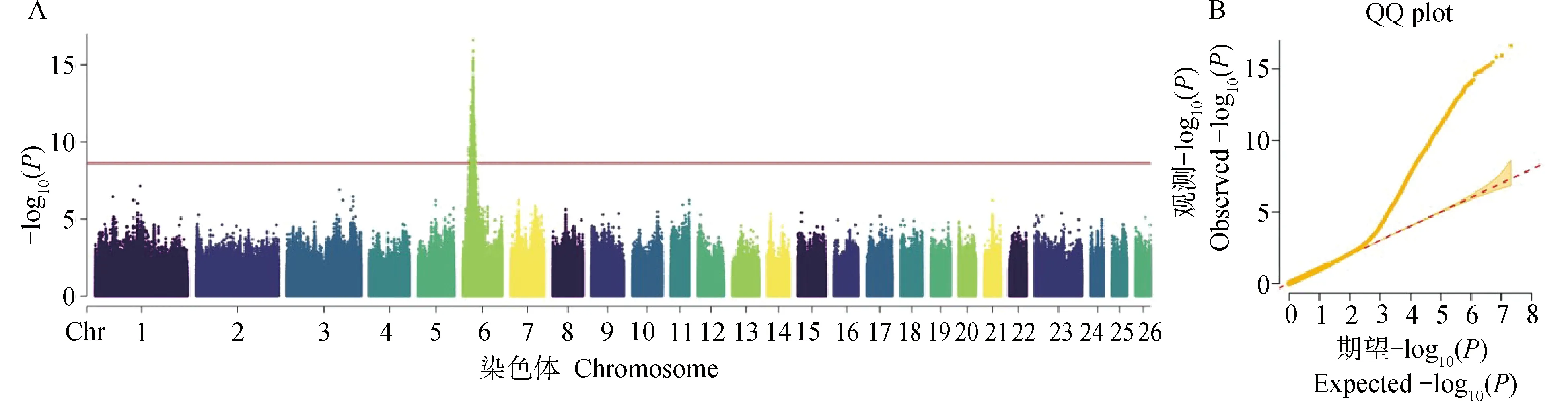
图1 SNP全基因组关联分析的曼哈顿图(A)和QQ-plot图(B)Fig.1 Manhattan map (A)and QQ-plot(B) of SNP genome-wide association study
2.2 KEGG富集分析
通过KOBAS对GWAS显着位点的注释基因进行KEGG富集分析后,共得到了17个校正P值达到显着性水平(correctedP<0.05)的通路,如图3所示,表1列出了KEGG富集分析的前20个通路。其中有多个信号通路已被证明与生殖过程有关。如Wnt信号通路、axon guidance、Th1和Th2细胞分化通路等。
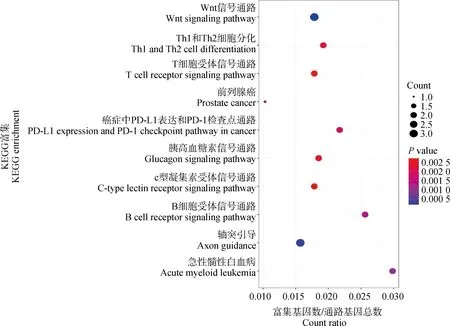
图3 KEGG富集分析气泡图Fig.3 Bubble diagram of KEGG enrichment analysis
2.3 连锁不平衡热图
根据BMPR1B基因在绵羊参考基因组的位置,选择信号最高点6∶34198746上下游10 kb区域、FecB(BMPR1B基因上A746G突变)突变位置上下游10 kb区域进行三角热图的绘制,如图4A、4B所示,并没有发现显着强连锁的区域。此外,为了研究候选基因GRID2两个SNPs位点和周边区域的连锁情况,选择g.35685218A>G和g.35685499C>T两个位置上下游10 kb区域进行三角热图绘制,发现周边SNP连锁情况相对较强,如图4C所示。
2.4 GRID2基因g.35685218A>G和g.35685499C>T的多态位点检测
通过KASP基因分型技术对GRID2基因的两个多态位点在560只东湖杂交奶绵羊群体中进行分型检测(图5),其中g.35685218A>G位点共检测到野生型(AA)的个体有211个,杂合型(AG)266个,突变纯合型(GG)83个。g.35685499C>T位点共检测到野生型(CC)的个体有220个,杂合型(CT)256个,突变纯合型(TT)84个。
2.5 GRID2基因g.35685218A>G和g.35685499C>T位点遗传多样性参数分析
本研究对560只东湖杂交奶绵羊GRID2的两个多态位点进行遗传多样性参数的计算,两个位点的等位基因频率和和基因型频率均十分相似(表2)。g.35685218A>G野生型(AA)基因型频率为0.377,杂合型(AG)基因型频率为0.475,突变纯合型(GG)基因型频率为0.148,A基因频率为0.614,G基因频率为0.386。g.35685499C>T野生型(CC)基因型频率为0.393,杂合型(CT)基因型频率为0.457,突变纯合型(TT)基因型频率为0.150,C基因频率为0.621,T基因频率为0.379。本研究中,g.35685218A>G位点的实际数值接近预测值(χ2<0.05),而g.35685499C>T位点的实际数值偏离预测值(χ2>0.05)。两个位点的多态信息含量均为中度多态性(0.25
2.6 GRID2基因g.35685218A>G和g.35685499C>T与产羔数关联分析
为了系统地研究GRID2基因g.35685218A>G和g.35685499C>T两个位点对产羔数的影响,本研究将不同胎次产羔数与基因型进行了关联分析,如表3所示。g.35685218A>G位点第一胎的野生型相较于突变杂合型平均产羔数增加了0.34只(P<0.05),相较于突变纯合型增加了0.64只(P<0.05)。第二胎的野生型相较于突变杂合型平均产羔数平均增加了0.57只(P<0.05),相较于突变纯合型增加了1.11只(P<0.05)。第三胎的野生型相较于突变杂合型平均产羔数平均增加了0.20只(P<0.05),相较于突变纯合型增加了1.06只(P<0.05)。
g.35685499C>T位点第一胎的野生型相较于突变杂合型平均产羔数增加了0.14只(P<0.05),相较于突变纯合型增加了0.50只(P<0.05)。第二胎的野生型相较于突变杂合型平均产羔数增加了0.41只(P<0.05),相较于突变纯合型增加了1.0只(P<0.05)。第三胎的野生型相较于突变杂合型平均产羔数增加了0.20只(P<0.05),相较于突变纯合型增加了1.15只(P<0.05)。
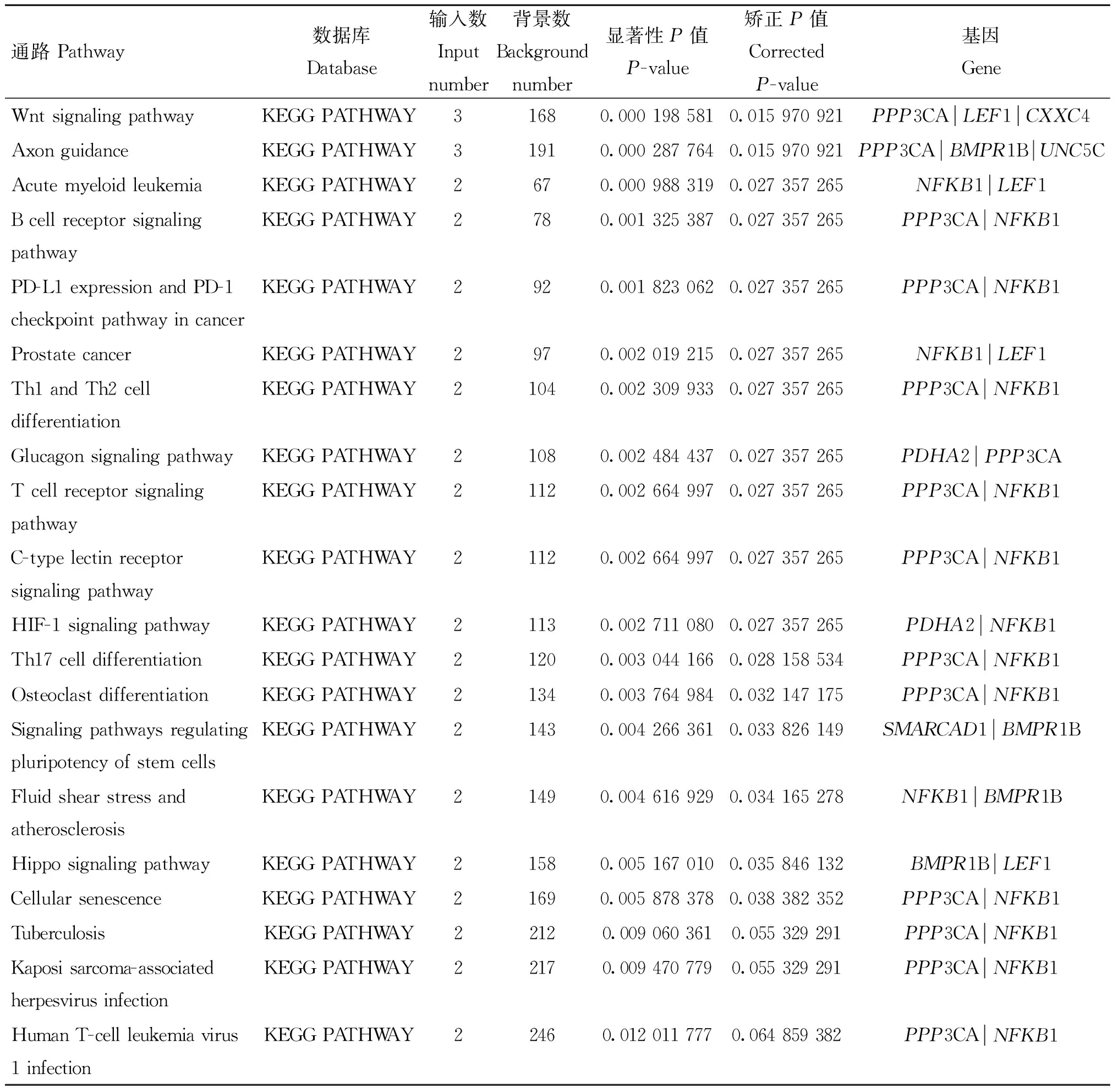
表1 KEGG富集通路Table 1 KEGG enrichment pathway
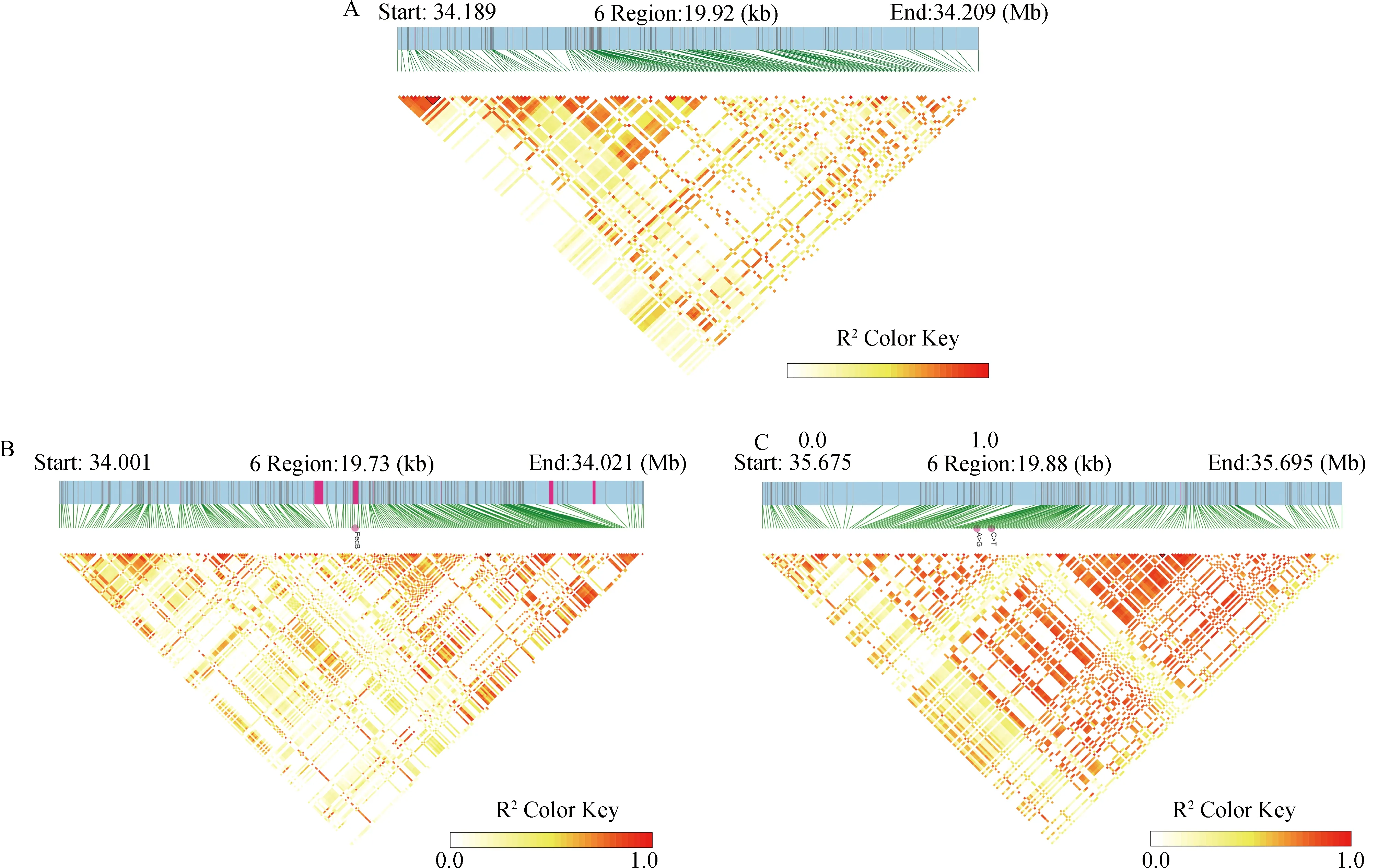
颜色从白色到红色,代表连锁程度从低到高。A.信号最高点6:34198746上、下游10 kb区域;B.FecB突变位置上、下游10 kb区域;C.g.35685218A>G和g.35685499C>T两个位置上、下游10 kb区域 The color ranges from white to red represent a low to high degree of linkage.A.The 10 kb region upstream and downstream of signal peak 6:34198746; B. The 10 kb region upstream and downstream of the FecB mutation site;C.The 10 kb region upstream and downstream of g.35685218A>G and g.35685499C>T图4 SNP连锁不平衡热图Fig.4 SNP linkage disequilibrium heat map
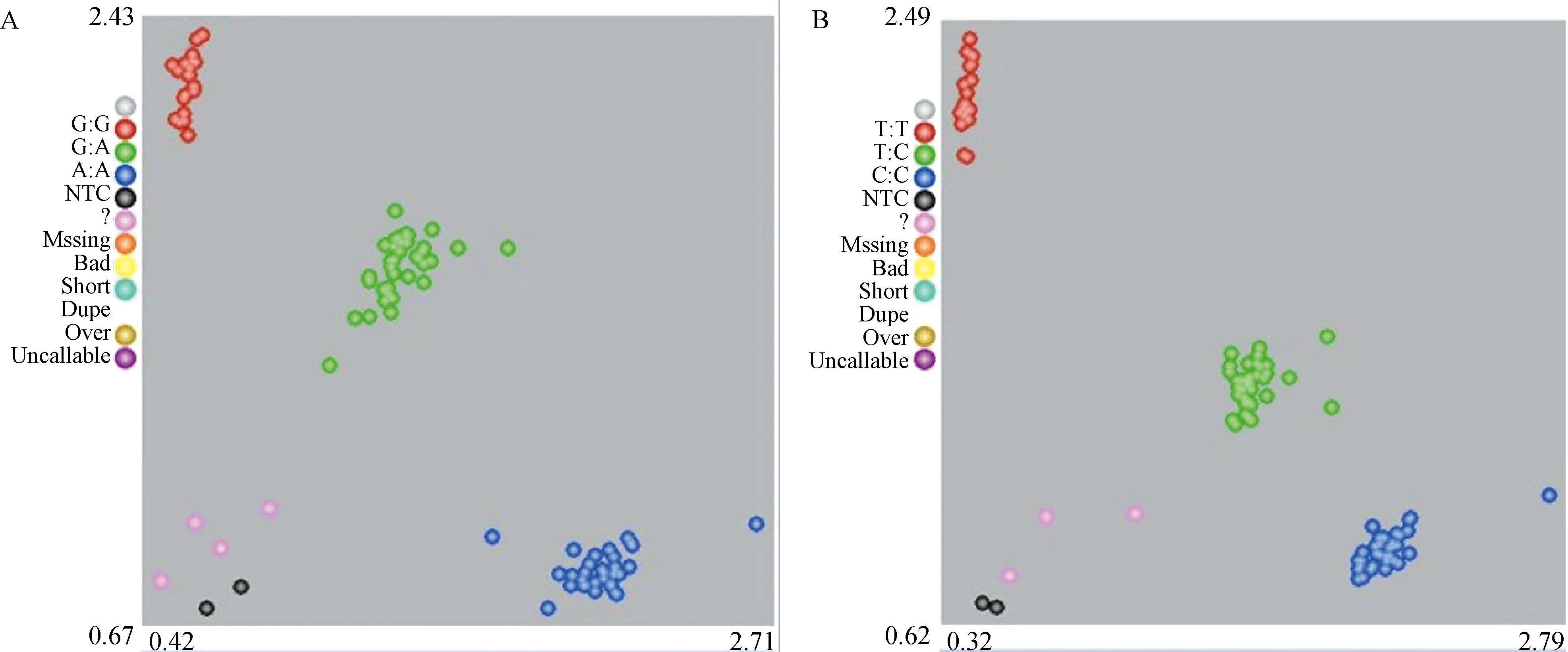
图5 GRID2基因g.35685218A>G(A)和g.35685499C>T(B)的KASP基因分型图Fig.5 KASP genotyping map of g.35685218A> G (A) and g.35685499C> T (B) in GRID2 gene
3 讨 论
本研究对168只东湖杂交奶绵羊的重测序数据与产羔性状进行了GWAS分析,共获得了1 163个达到显着性水平的SNPs位点。根据注释结果,发现了与产羔性状有关的多个基因和信号通路。其中,BMPR1B基因中FecB(A746G)错义突变导致氨基酸变化(Q249R),从而增加羊的排卵率[24],此突变对羊排卵具有加性效应[25-27],本课题组前期已经在奶绵羊群体中大量检测了FecB突变,研究结果显示,FecB突变显着影响东湖杂交二代羊的产羔数[28]。
根据KEGG富集分析的结果,同样有一些和繁殖过程有关的通路。Wnt信号通路是一个重要的调控机制,在细胞分化、增殖、凋亡和胚胎发育过程中发挥着关键作用。研究表明,Wnt信号通路在小鼠和人类卵巢中的活性会随着动情周期的变化而变化,表明其在生殖周期中起着重要作用[29]。另外一个与繁殖过程相关的通路是轴突导向通路(axon guidance),这个通路在胚胎发育期间,对于神经系统的形成和连接至关重要。研究发现,轴突导向通路的一些关键基因在胚胎期间还会表达于生殖细胞系中,这些基因对于生殖细胞系的发育也有重要的调控作用[30]。Th1和Th2细胞分化通路在免疫调节过程中发挥着关键作用,研究表明,妊娠期间,母体的免疫系统处于一种Th2倾向状态,Th1/Th2平衡的变化可能与早期流产、胎儿生长受限和先兆子痫等疾病有关[31]。此外,HIF-1还被认为在雌性哺乳动物中调节子宫内膜生长和修复,从而有助于维持正常的妊娠[32]。
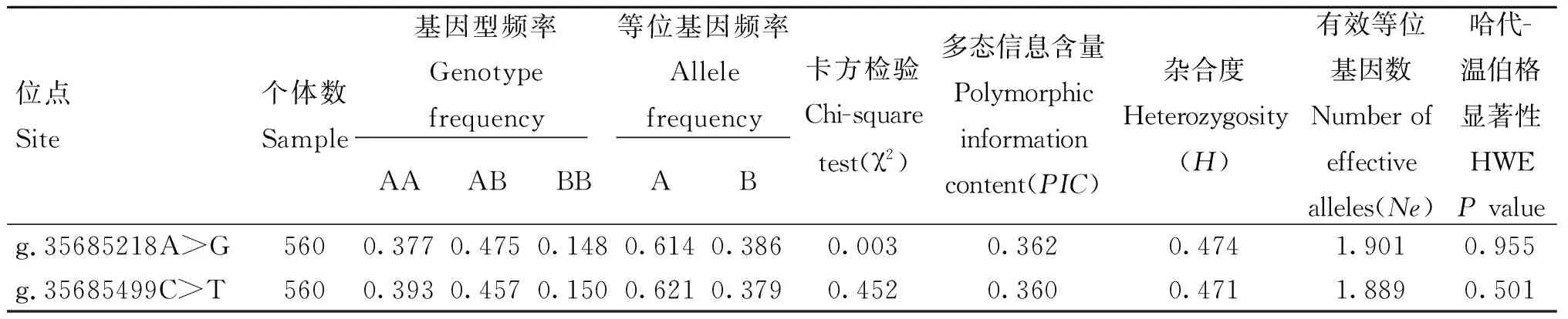
表2 GRID2基因g.35685218A>G和g.35685499C>T的遗传多态性参数Table 2 Genetic polymorphism parameters for the g.35685218A>G and g.35685499C>T loci in GRID2
同时,本研究也发现与东湖杂交奶绵羊产羔数相关的显着性SNPs位点中有几个位于GRID2基因,此基因在大脑皮层和小脑中广泛表达[33]。GRID2基因位于染色体4q22-q23上,包含约118 000个碱基对,并编码约3 930个氨基酸,编码蛋白质为Delta-2,在中枢神经系统中发挥重要作用,参与了许多神经功能的调节,包括学习、记忆、情绪和感知[34]。有报道显示,GRID2可能与动物繁殖性状有关,一项对31只大足黑山羊产羔数的全基因组选择信号研究发现,GRID2可能是影响产羔数的候选基因,LD分析表明,高产大足黑山羊表现出与低产山羊显着不同的连锁不平衡模式[35]。根据Chen等[36]对两个不同FecB基因型小尾寒羊不同生殖周期的垂体组织转录组研究发现,GRID2在不同基因型羊的垂体组织间差异表达,推测该基因可能介导了一些生殖相关激素的释放。此外,GRID2基因也被证明和猪卵母细胞的增殖和凋亡过程相关[37]。另外,6号染色体上存在大量的连锁区域,可能还存在一些其他类型的变异,例如结构变异(structural variation, SV)、拷贝数变异(copy number variation, CNV)等,受二代测序数据的限制,很难检测到这些变异,需要进一步的挖掘。并且,统计学分析发现GRID2基因g.35685218A>G和g.35685499C>T两个位点的野生型均处于优势状态,且两个位点在胎次增加时均值也增加。
4 结 论
本研究结果表明,通过全基因组关联分析确定了6号染色体上1 163个达到显着性水平的SNPs位点,注释到候选基因有BMPR1B、PDLIM5、PDHA2、UNC5C、GRID2、NFKB1、TSPAN5、STPG2、PPP3CA。其中GRID2基因2个SNPs(g.35685218A>G和g.35685499C>T)对东湖杂交奶绵羊产羔数有负效应,可以作为分子标记辅助选择的候选位点。



