韩浩园,李世凯,2,杨瑞巧,李曼曼,李 君,哈 斯,赵金艳,魏红芳,权 凯*
(1.河南牧业经济学院动物科技学院,郑州 450046;2.华南农业大学动物科学学院,广州 510642)
槐山羊是我国优良的地方品种,核心产区位于周口市沈丘县,群体分布覆盖整个豫东地区,是河南养羊业的第一名片,“槐山羊肉”、“槐山羊板皮(槐皮)”为沈丘县国家地理标志保护产品。槐山羊以皮质好、肉质鲜嫩、繁殖率高三大优势而闻名于世[1],槐山羊性成熟早,多胎多产,繁殖性能优异,能一年两产或两年三产,经产母羊产羔率平均为329.20%。2003年以来由于受到国际市场的冲击,波尔山羊等品种引入后对槐山羊大面积杂化,造成槐山羊群体血统含量参差不齐,生产力方向和生产性能也发生了巨大的变化,目前槐山羊多胎性状的功能基因和遗传机制尚不明晰。
目前,国内关于羊的高繁基因研究主要集中于绵羊[2-8],针对山羊品种的高繁殖力研究仅限于少数几个品种,前期基于DNA多态性研究,在陇东绒山羊、黔北麻羊、贵州黑山羊、辽宁绒山羊、陕北白绒山等品种中发现GJB6、DNAH1、NCOA1、FSHR等基因遗传变异与山羊繁殖力相关[9-14]。随着测序技术的革新,生命科学研究进入了后基因组时代,诞生了各种组学研究方法,其中转录组学研究是从转录水平上反映出基因的表达情况及其调控规律,是研究基因功能和筛选新基因的有效技术之一[15-16]。目前,在山羊中已陆续开展与繁殖性能相关的转录组研究,Ling等[17]对安徽白山羊卵泡期单、多羔的山羊卵巢进行转录组测序,通过比较分析信使RNA(mRNAs)、微RNA(miRNAs)和长非编码RNA(lncRNAs)的差异表达,发现在多羔组卵巢中miR-6404和miR-29c可能对山羊的繁殖性能有着重要调控作用。此外,大部分研究集中于卵泡不同发育阶段的基因表达模式,基于单细胞转录组,王俊杰[18]针对济宁青山羊卵泡不同发育阶段的卵母细胞和颗粒细胞,筛选出一部分与阶段发育相关联的功能性转录活动。Xu等[19]通过获取大足黑山羊不同发育阶段的完整卵泡转录组,发现128个mRNA、4个lncRNAs、49个miRNAs和290个环状RNA(circRNAs)在大、小滤泡中表达差异显着,差异mRNA富集到与卵泡发育相关的信号通路中。Liu等[20]通过生物信息学分析确定安徽白山羊卵泡期和黄体期卵巢中差异表达mRNA的表达模式,鉴定出3 770个差异表达mRNA,功能富集分析表明,HSD17B7、3BHSD和SRD5A28基因可能与孕酮的合成有关,RPL12、RPS13和RPL10基因可能与卵母细胞的生长和成熟有关。
目前,对于槐山羊繁殖性能功能基因及其调控机制的研究极少,本试验以高繁殖力品种槐山羊为研究对象,采样个体均来自沈丘县农牧科技研发中心,饲养标准与管理水平一致,依据经产母羊平均每胎产羔数分为单羔组和多羔组,利用转录组测序分析筛选与多羔性状相关的候选基因,可以为探讨山羊产羔性状的分子机理提供理论参考。
1 材料与方法
1.1 样本采集及建库测序
槐山羊卵巢组织采自沈丘县农牧科技研发中心,选取健康无病的3岁左右第3胎次母羊,试验个体体格大小相似,饲养标准一致,依据研发中心的槐山羊繁殖性能记录,采集3只平均每胎产羔数为1只的母羊为单羔组(S1~S3),采集3只平均每胎产羔数大于2只(分别为2.3、2.6和3.3只)的母羊为多羔组(M1~M3),屠宰后采集其卵巢组织,立即置于液氮中保存。利用TRIzol法提取卵巢组织总RNA,取RIN值>7.0,完整性良好的RNA送至派森诺进行建库测序,利用Illumina平台建库测序,采用paired-end 2×150 bp测序模式。
1.2 数据处理
检验合格样品经过上机测序,生成 FASTQ 的原始数据(raw data)。采用cutadapt去除3′端带接头的序列,去除平均质量分数低于Q20的reads,生成clean data。使用HISAT2(http://ccb.jhu.edu/software/hisat2/index.shtml )软件BWT算法将过滤后的reads比对到山羊参考基因组(Capra_hircus.ARS1.dna.toplevel.fa)上。
1.3 差异表达基因及功能富集分析
采用DESeq对基因表达进行差异分析,表达差异倍数|log2FoldChange|>1,显着性P<0.05的基因为差异表达基因(differentially expressed genes,DEGs)。使用R语言pheatmap软件包对差异基因的并集和样品进行双向聚类分析,根据同一基因在不同样品中的表达水平和同一样品中不同基因的表达模式进行聚类,采用euclidean方法计算距离,采用层次聚类最长距离法(complete linkage)进行聚类。本研究使用topGO进行GO富集分析,使用KAAS(http://www.genome.jp/tools/kaas/)中bi-directional best hit(BBH)参数进行KEGG功能注释,P<0.05为显着富集。
1.4 荧光定量PCR验证
通过实时荧光定量PCR(qRT-PCR)技术,以GAPDH为内参基因,挑选6个基因(ADCY8、FOS、PAK1、NR4A2、ADAMTS、NR4A1)进行验证。荧光定量PCR反应体系共10 μL,包括:2×SYBR qPCR master mix 5 μL,上、下游引物(表1)各0.2 μL,cDNA 0.1 μL,ddH2O 4.5 μL;反应程序为:95 ℃预变性3 min;95 ℃ 10 s,60 ℃ 30 s,40个循环。利用2-ΔΔCt方法计算基因表达量,以RNA-Seq方法的基因表达log2(Fold Change)值为横坐标,以实时荧光定量方法的log2(2-ΔΔCt)值为纵坐标,进行相关分析,并计算皮尔逊相关系数。
1.5 遗传变异分析
采用varscan程序获取SNP和InDel位点,过滤标准为:SNP位点碱基Q>20;覆盖该位点的reads数目>8;支持突变位点的reads数目>2;SNP位点的P<0.01。
2 结 果
2.1 下机及过滤数据
对每个样品的下机数据(raw data)分别进行统计,并对带接头、低质量的reads进行过滤,统计结果见表2。平均reads总数为41 163 239条,平均碱基总数为6 174 485 900 bp,平均clean reads数为37 908 182条,平均clean data碱基数为5 686 227 300 bp,clean data的reads和碱基占raw data的百分比为92.08%。
2.2 参考基因组数据比对
将过滤后的高质量序列与参考基因组序列进行比对,平均95.60%的clean data比对到参考基因组,其中96.21%的序列为特异比对,可用于后续分析,基本信息统计见表3。将比对到基因组上的reads分布情况进行统计,73.24%的reads定位到基因区域,26.76%的reads定位到基因间区,84.56%的reads定位到外显子区域,比对结果见表4。
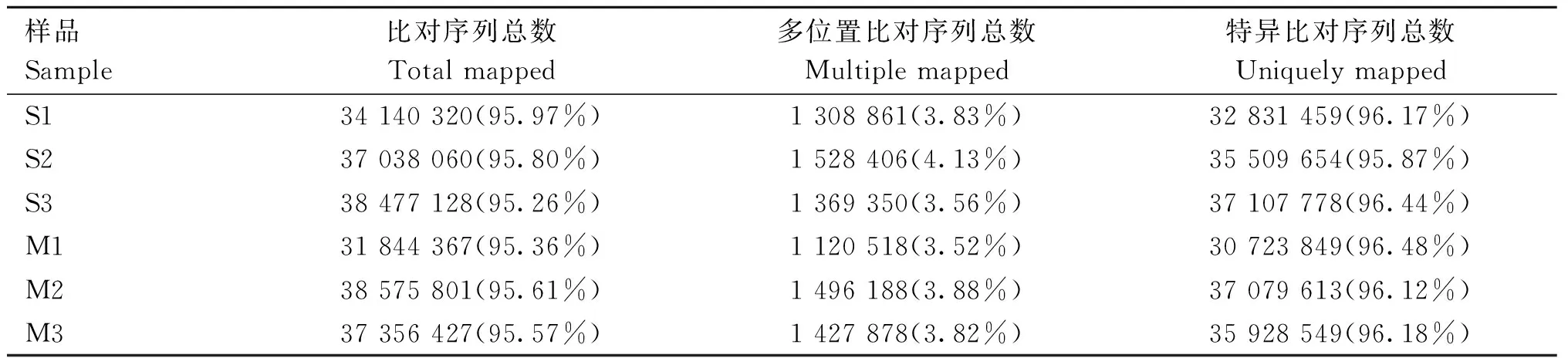
表3 卵巢组织转录组数据与参考序列比对结果Table 3 The mapping results between transcriptome data from ovarian tissue and reference sequences
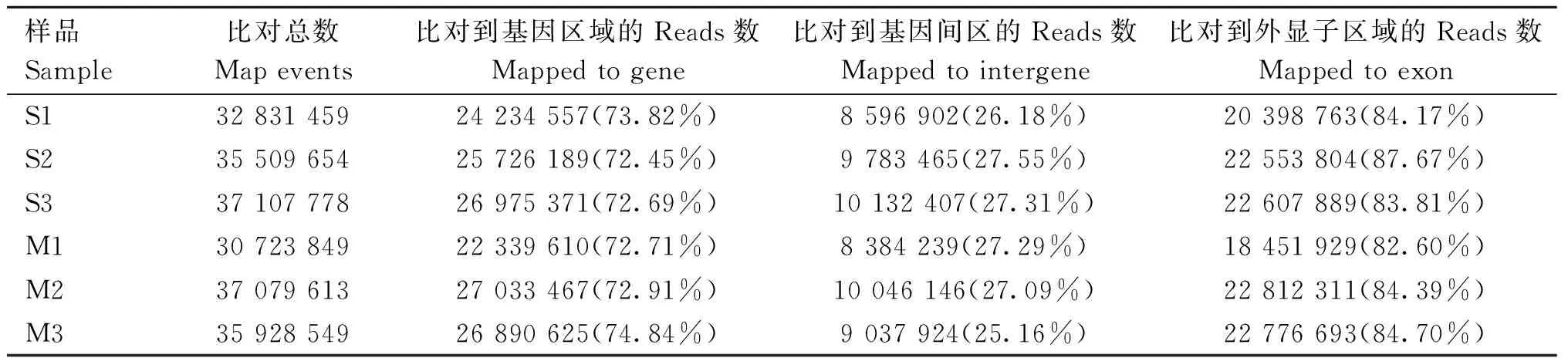
表4 转录组数据位置比对Table 4 RNA-Seq mapped events
2.3 差异表达基因分析
本研究在多羔与单羔组间共发现121个差异表达基因(|log2(Fold Change)|>1,P<0.05),包括30个上调基因和91个下调基因,差异表达基因火山图见图1,蓝色为多羔组相比于单羔组下调基因,红色为多羔组相比于单羔组上调基因。下调基因中显着性前5的基因为RXFP1、DUSP1、THSD7B、CCN1和MRAP2,上调基因中显着性前5的基因为ENSCHIG00000021945、SLF1、ENSCHIG00000001740、ND2和BRINP3(表5)。
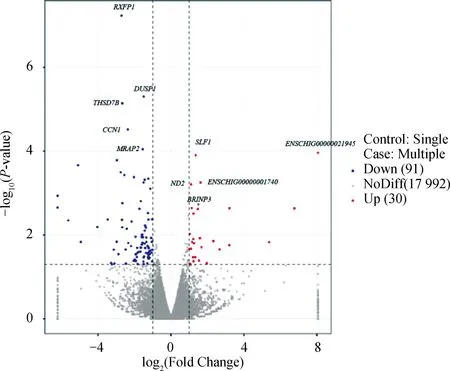
红点表示多羔组上调基因,蓝点表示多羔组下调基因Red dots represent up-regulated genes, blue dots represent down-regulated genes图1 差异表达基因火山图Fig.1 Volcano map of DEGs
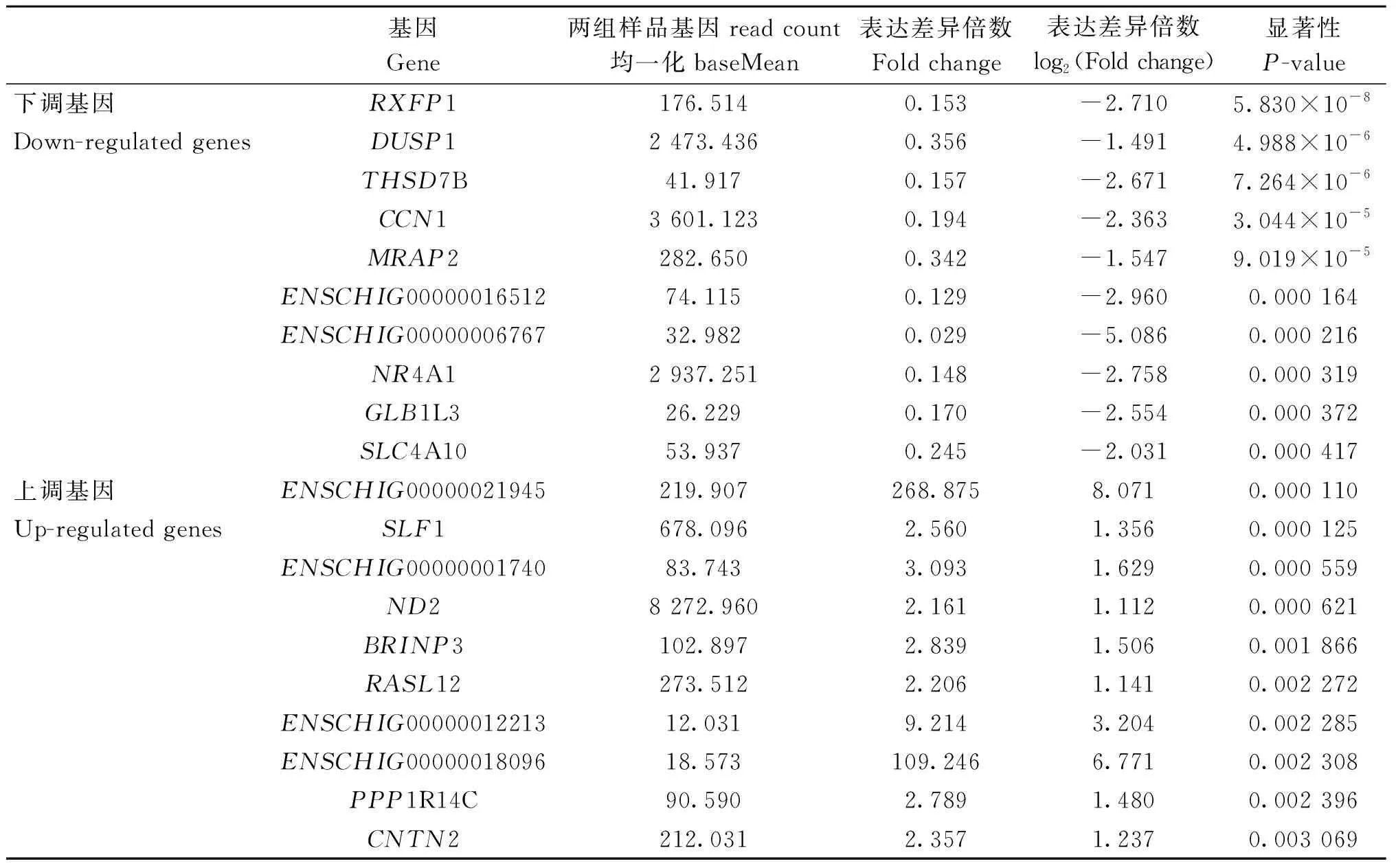
表5 多羔组与单羔组间差异表达基因(前10)Table 5 The top 10 DEGs between multi-lamb group and single-lamb group
2.4 聚类分析
基于差异表达基因的表达量,双向聚类分析结果表明单羔组(Single)S1~S3个体聚为一类,多羔组(Multiple)M1~M3个体聚为一类,由图2可见,单羔组和多羔组间的差异表达基因的表达模式差异明显,推测本研究筛选的差异基因表达量可能与槐山羊的产羔数相关。
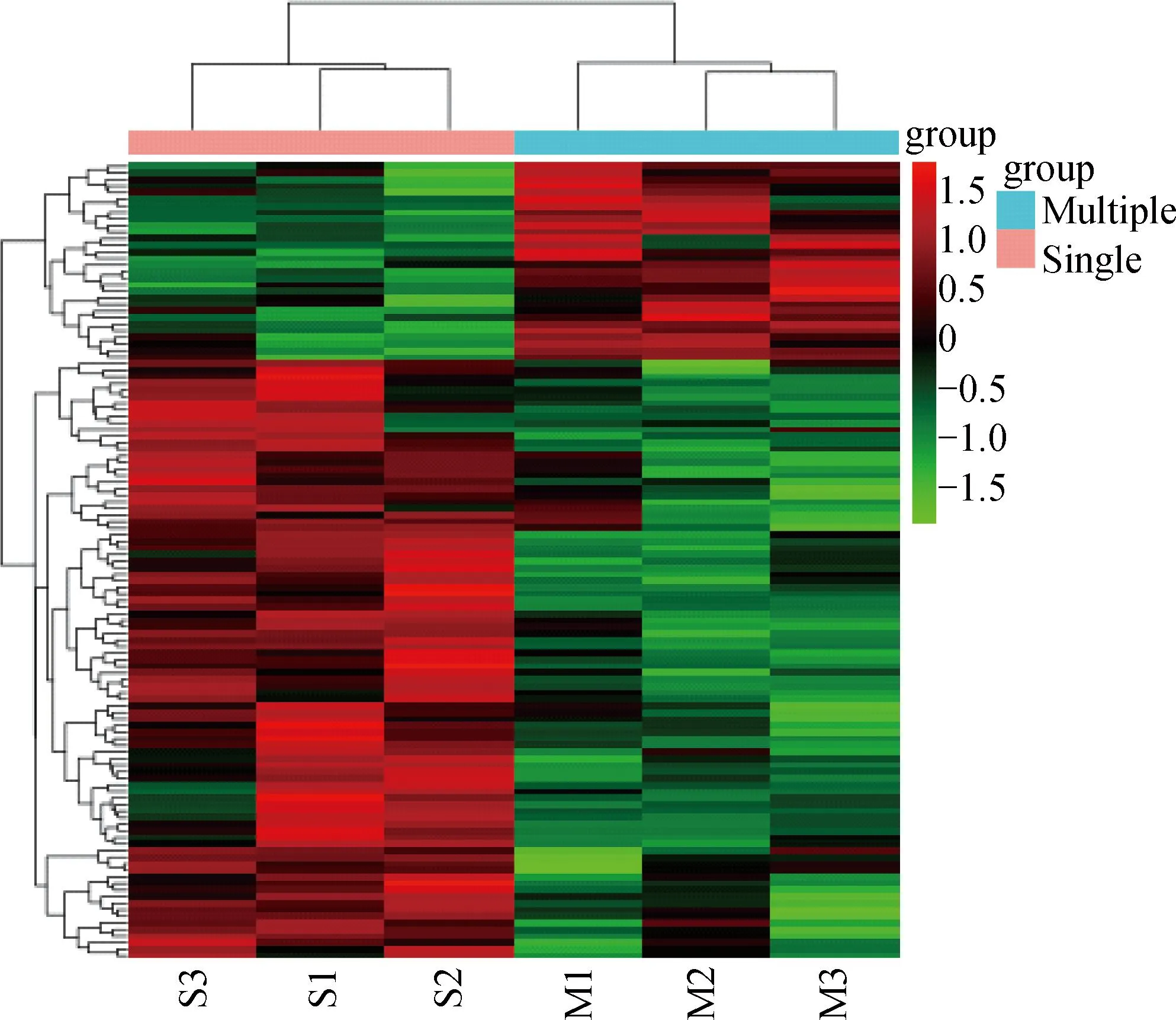
红色表示高表达基因,绿色表示低表达基因Red represents up-regulated genes, green represents down-regulated genes图2 差异表达基因双向聚类结果Fig.2 Two-way clustering results of DEGs
2.5 功能富集分析
对差异表达基因进行功能富集分析,GO分析共显着富集到440个功能条目(P<0.05),极显着富集到100个条目(P<0.01),图3显示了显着性最高的前20个GO条目,其中较多的差异基因富集到发育过程(developmental process)、解剖结构发育(anatomical structure development)、多细胞生物发育(multicellular organism development)和动物器官发育(animal organ development)等功能条目中(表6),PTX3、MMP13、PAK1、ADAMTS1、COL1A2、CCN1和SLC4A10等基因在单羔与多羔组间表达差异显着,且参与多条组织器官结构发育功能条目,可能影响母羊繁殖系统的发育过程。本研究发现,NR4A1和NR4A2基因富集到促肾上腺皮质激素释放激素应答(response to corticotropin-releasing hormone)和促肾上腺皮质激素释放激素刺激的细胞应答(cellular response to corticotropin-releasing hormone stimulus)2个功能条目中(表6),促肾上腺皮质激素释放激素在外周与生殖功能相关,推测NR4A1和NR4A2基因可能通过参与促肾上腺皮质激素释放激素应答,进而调控槐山羊的产羔数。
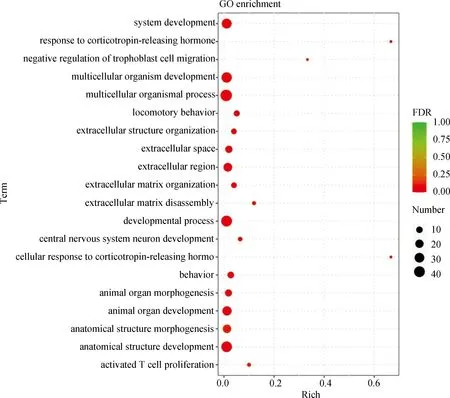
图3 GO富集分析气泡图Fig.3 The bubble diagram of GO enrichment analysis
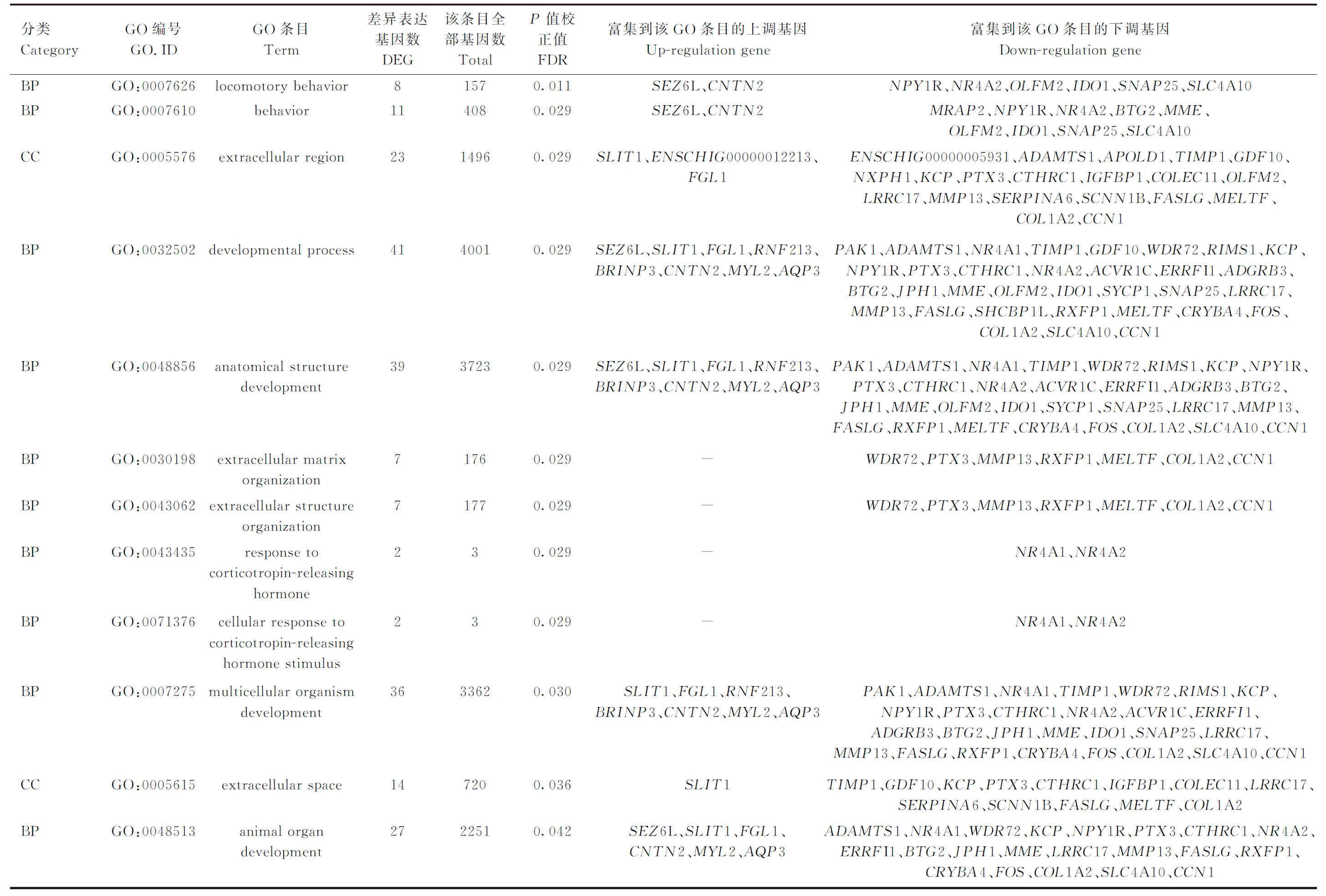
表6 GO富集分析Table 6 GO enrichment analysis
GO有向无环图结果表明(图4),生物过程中的发育过程(developmental process)、多细胞生物发育(multicellular organismal process)和行为(behavior)条目显着富集,且其下属功能条目均被显着富集,其中,发育过程(developmental process)、多细胞生物过程(multicellular organismal process)、解剖结构发育(anatomical structure development)、多细胞生物发育(multicellular organism development)、系统发育(system development)功能条目均包括超过30个差异表达基因,本结果说明槐山羊产羔数高低可能与其细胞生物过程、解剖结构发育及系统发育有关。

图4 生物过程中部分条目的GO有向无环图Fig.4 The GO directed acyclic graph based on a part of terms in biology process
KEGG分析将差异表达基因共富集到15条通路中(图5),其中6条信号通路与内分泌系统有关(表7),包括甲状旁腺激素的合成、分泌和作用(parathyroid hormone synthesis, secretion and action)、松弛素信号通路(relaxin signaling pathway)、醛固酮合成与分泌(aldosterone synthesis and secretion)、脂肪细胞中脂肪分解的调节(regulation of lipolysis in adipocytes)、皮质醇的合成与分泌(cortisol synthesis and secretion)、催产素信号通路(oxytocin signaling pathway),在多羔组与单羔组比较中发现FOS、MMP13、NR4A1、NR4A2、ADCY8基因差异表达,且富集在多条内分泌相关的KEGG通路中(表7),本结果说明槐山多羔性状可能与激素分泌及上述5个基因的表达情况有关。
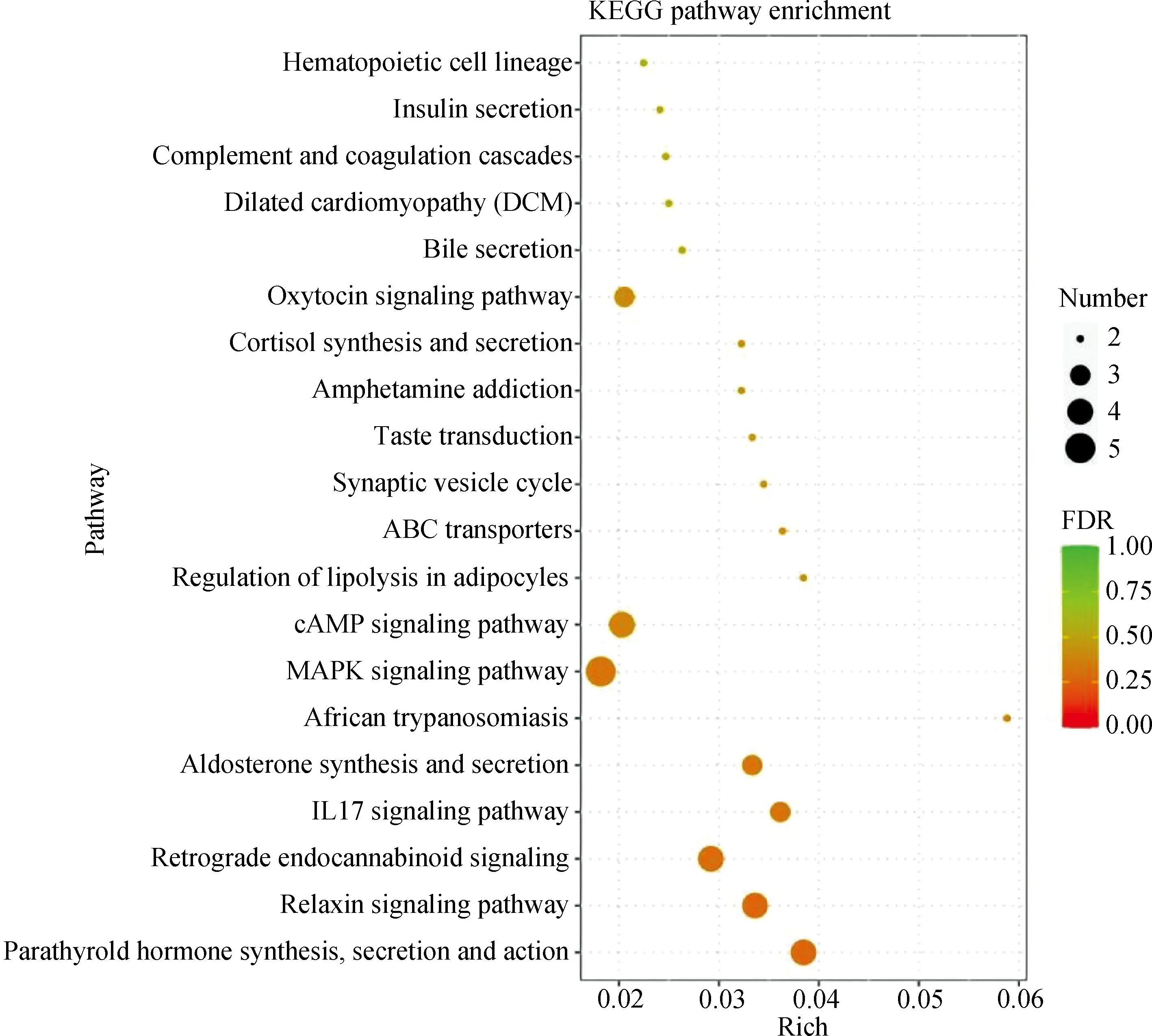
图5 KEGG富集分析气泡图Fig.5 The bubble diagram of KEGG enrichment analysis
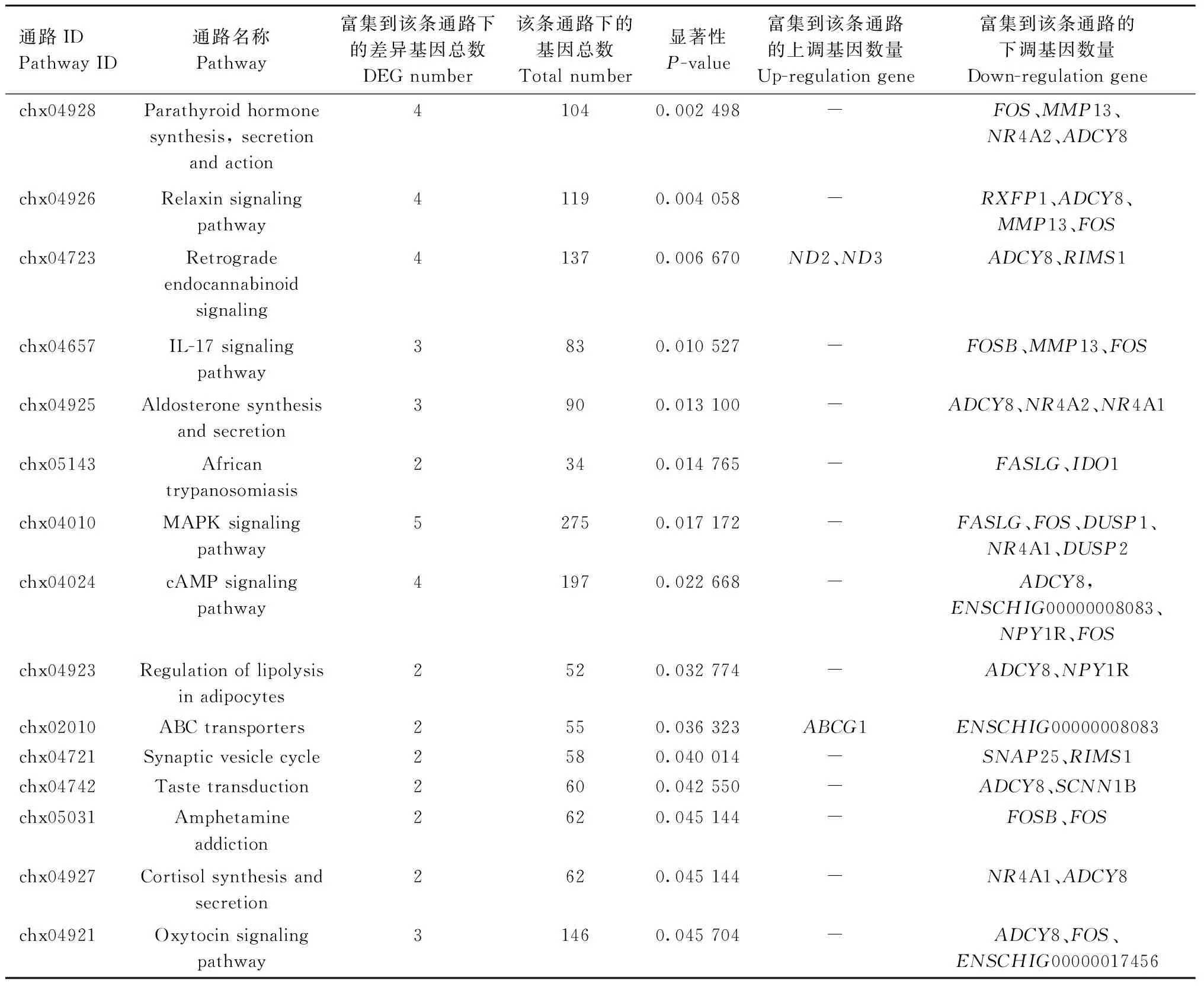
表7 KEGG功能富集分析Table 7 KEGG functional enrichment analysis
2.6 实时荧光定量验证
以GAPDH基因为内参基因,用实时荧光定量法检测单羔组和多羔组槐山羊卵巢组织中的ADCY8、FOS、PAK1、NR4A2、ADAMTS、NR4A1基因表达量,结果显示6个基因在槐山羊多羔组中显着下调,荧光定量PCR结果与RNA-Seq结果一致,分别以RNA-Seq及qRT-PCR的单羔、多羔组间的表达量差异倍数为横、纵坐标,两种方法得到的6个基因表达量的皮尔逊相关系数R2为0.971 7,显着性P<0.01(图6),证明RNA-Seq的结果准确可靠。
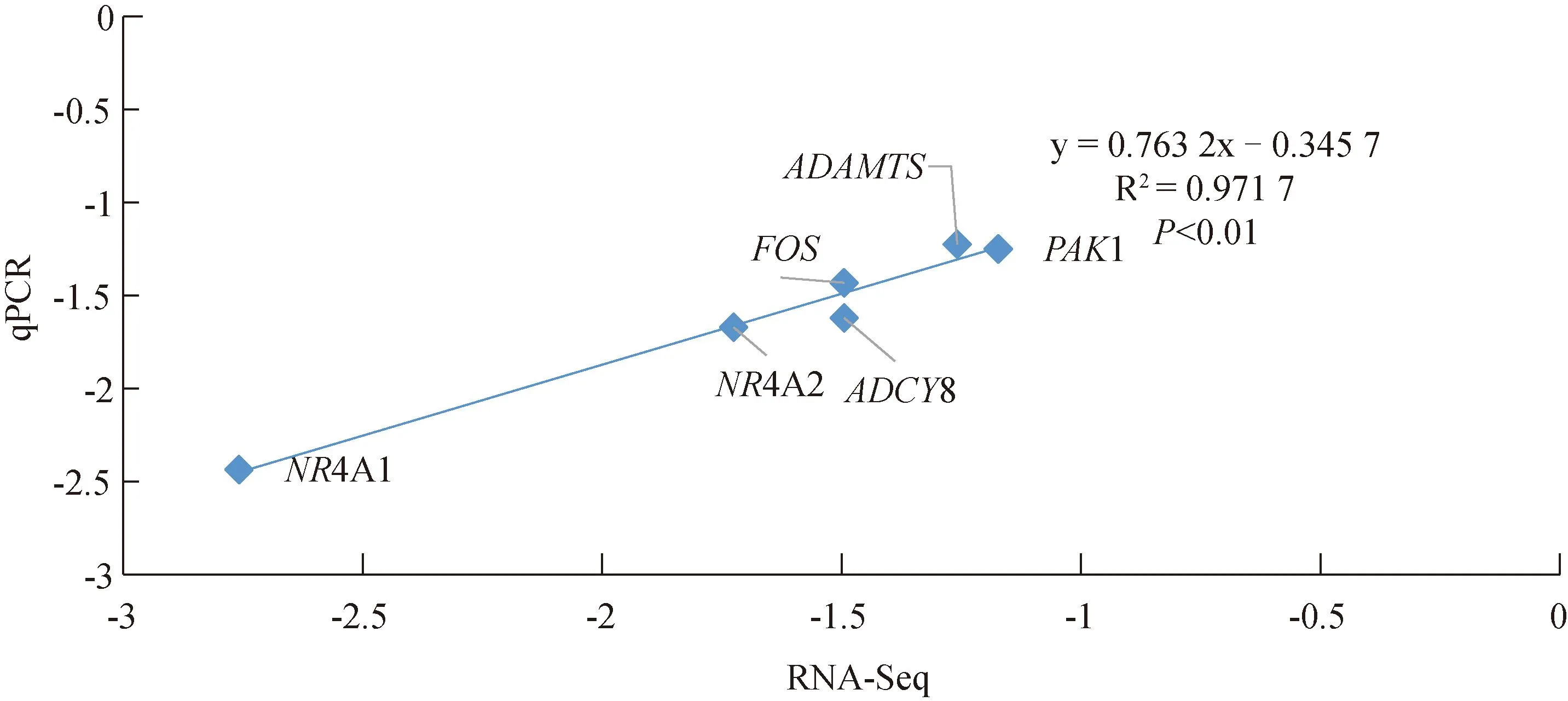
图6 实时荧光定量PCR验证结果Fig.6 The validation results of quantitative real-time PCR
2.7 遗传变异筛选
SNP/Indel筛选结果表明6个个体的SNP数为82 358~106 639个,Indel数为8 856~10 844个(表8)。在上述结果中发现的与槐山羊产羔数相关的11个基因(PTX3、MMP13、PAK1、ADAMTS1、COL1A2、CCN1、SLC4A10、FOS、NR4A1、NR4A2、ADCY8)中共发现236个SNPs,其中39个SNPs位于外显子区域,7个SNPs为错义突变,4个位于ADAMTS1基因,1个位于PTX3基因,2个位于CCN1基因(表9)。

表8 SNP/Indel筛选结果Table 8 The screen results of SNP/Indel
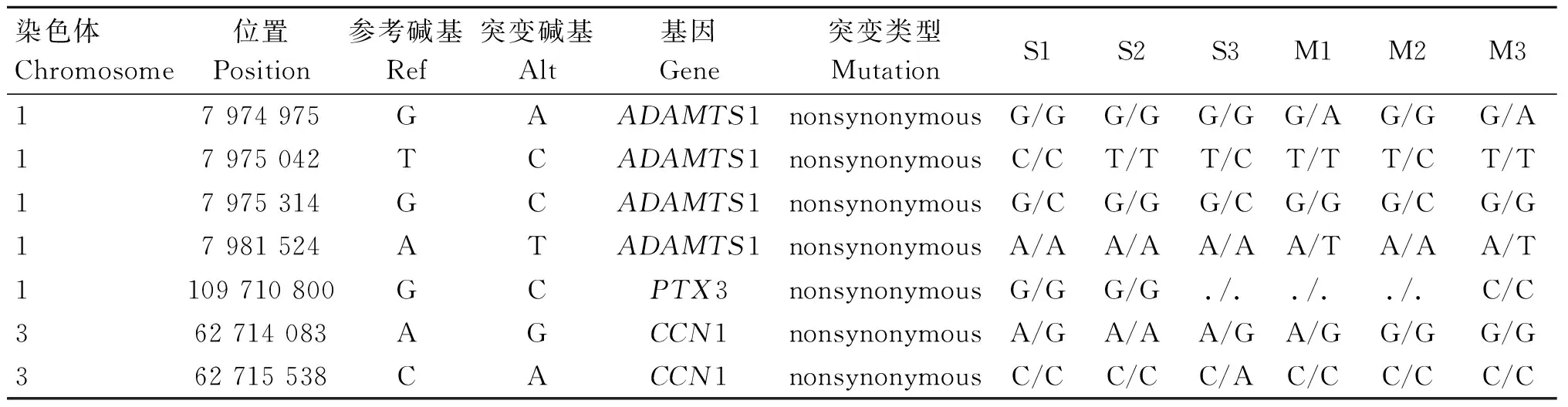
表9 与槐山羊产羔数相关基因的非同义突变Table 9 The nonsynonymous mutations related to lambing numbers of Huai goats
3 讨 论
繁殖性能包括生育力、生产力和繁殖力3个方面,它们分别指的是母畜每年的妊娠率、每胎的产羔数和每年的产羔数,排卵率和窝产羔数是繁殖性能的直接体现。国内外关于山羊的研究报道相对匮乏,相关重视程度和技术发展远远比不上绵羊,与产羔数相关的研究主要集中于黔北麻羊、济宁青山羊和绒山羊等品种中,对于槐山羊尚无相关报道。基于转录组测序技术,本研究构建了6个单、多羔槐山羊卵巢组织mRNA文库,共发现了121个差异表达基因,其中多羔组上调基因30个,下调基因91个,富集到多条组织器官结构发育功能条目及多条内分泌系统相关激素分泌信号通路。在单、双羔燕山绒山羊卵巢组织转录组测序中发现的差异表达基因富集于繁殖过程相关通路中,包括松弛素信号通路、cAMP 信号通路、TGF-beta信号通路、MAPK信号通路和雌激素信号通路等[21]。在济宁青山羊的转录组分析中也发现类固醇代谢和胰岛素通路可能参与了产羔数性状的进化选择[18],证明这些信号通路与山羊的产羔数相关。本研究筛选出PTX3、MMP13、PAK1、ADAMTS1、COL1A2、CCN1、SLC4A10、ADCY8、NR4A1和NR4A2等主要候选基因,而燕山绒山羊中筛选的基因包括ADAMTS16、GABRA1、TIMP3、TRPC1、SOS2、WNT2、SLC6A3、PRLHR和SERPINB11等,说明不同品种间控制产羔数的调控基因可能存在差异。在Pelibuey羊的卵巢组织研究中同样筛选出NR4A1基因可能与羊繁殖相关[22],与本研究结果一致。
本研究在单羔与多羔组间发现的差异表达基因包括PTX3、MMP13、PAK1、ADAMTS1、COL1A2、CCN1、SLC4A10、ADCY8、NR4A1和NR4A2等。已有研究表明其中PAK1、PTX3、MMP13、ADAMTS1、ADCY8、NR4A1和NR4A2基因均在卵巢组织中表达,且与卵泡发育、排卵及类固醇激素合成相关。如在对济宁青山羊研究中发现PAK1基因与山羊产羔数相关[18],与本研究结果一致。PTX3基因可通过抑制PI3K/AKT/mTOR和Erk1/2信号通路,从而调控奶山羊卵巢颗粒细胞的增殖、凋亡,并调控类固醇合成关键酶CYP19A1和CYP11A1的表达,进而作用于颗粒细胞类固醇激素的生成[23]。MMP13胶原酶则参与了卵泡壁的降解,在卵泡形成和排卵中具有重要功能[24-26]。ADAMTS1基因mRNA和蛋白在优势卵泡周围富集,参与子宫和卵巢的形态发生、子宫内膜蜕膜化、卵子的发生和排卵过程,促进卵泡的发育和成熟[27-28]。也有研究发现,ADCY8基因参与卵巢类固醇生成、G蛋白和雌激素信号通路,与卵母细胞减数分裂、孕激素介导的卵母细胞成熟有关[29]。
此外,本研究发现NR4A1和NR4A2基因参与促肾上腺皮质激素释放激素应答,促肾上腺皮质激素释放激素在外周与生殖功能相关,推测NR4A1和NR4A2基因可能通过促肾上腺皮质激素释放激素应答,进而调控槐山羊的产羔数。前人研究发现NR4A1基因作为一个孤核受体的转录因子,编码类固醇-甲状腺激素超家族,同样调节关键类固醇激素合成酶基因的转录[30-31],而孤儿核受体NR4A2与细胞生长或凋亡密切相关,可能参与卵巢上皮性癌的发生与发展[32]。结合本研究结果,推测PAK1、PTX3、MMP13、ADAMTS1、ADCY8、NR4A1和NR4A2基因表达于山羊卵巢组织,且调控山羊卵泡发育及产羔数。
本研究在与槐山羊产羔数相关的11个基因(PTX3、MMP13、PAK1、ADAMTS1、COL1A2、CCN1、SLC4A10、FOS、NR4A1、NR4A2、ADCY8)中共发现236个SNPs,其中编码蛋白发生改变的SNPs为7个,位于ADAMTS1、PTX3和CCN1基因中,本研究所发现的遗传变异位点与产羔数的关系需要进一步验证。目前尚无这些基因的SNP与山羊产羔数关系的研究。已发现的与中国本地山羊高繁性能有关的SNPs研究主要集中在FSHR、NCOA1、RBP4和DNAH1等基因[9]。在长白猪群体的NR4A1基因第5内含子发现了A/G突变,多态性分型结果表明胎次产仔数呈现GG>AG>AA的趋势[33],该基因SNP在山羊产羔数中的作用有待进一步研究及验证。
4 结 论
本研究构建了多羔及单羔共6个个体的mRNA文库,发现PTX3、MMP13、PAK1、ADAMTS1、COL1A2、CCN1、SLC4A10、FOS、NR4A1、NR4A2、ADCY8等11个基因显着差异表达,且参与到组织器官的结构发育及内分泌系统相关激素分泌,可能通过调控山羊卵泡发育、排卵及类固醇激素分泌,进而影响山羊产羔数。本研究为解析山羊多羔性状遗传机制提供了理论依据。



