祝雪丽,张龙超,王立贤,蒲 蕾,刘 欣*
(1.中国农业科学院北京畜牧兽医研究所,北京 100193;2.天津农学院动物科学与动物医学学院,天津 300384; 3.天津市绿色生态饲料重点实验室,天津 301800)
生猪养殖在我国畜牧业中占有较大的比重,仅2021年年底全国生猪出栏量已达4亿多头[1]。过去几十年来,为了提高生产效率,猪的选择主要集中在胴体瘦肉率和生长性状上[2]。近年来,多以体脂、肌内脂肪作为猪肉品质的研究对象[3]。然而猪生长速度提高的同时,却降低了猪的肉质性状,导致适口性差,缺少风味,降低了消费者的满意度。肌内脂肪(IMF)与皮下脂肪(BF)是家畜的两个主要脂肪库[4],同时多项研究表明,食肉最佳的肌内脂肪含量为2%~3.5%[5]。因此,如何在稳定皮下脂肪含量、提高瘦肉率的情况下,提高肌内脂肪含量,是当今猪业生产中需要解决的问题。
本研究所关注的水通道蛋白,目前已在哺乳动物中发现13种(AQP0~AQP12),按功能可以分为传统的水通道蛋白[6]和甘油水通道蛋白(可以通透中性溶质如甘油、其他小的非离子物质和水)[7]。水通道蛋白9(aquaporin9,AQP9)属于甘油水通道蛋白一类,于1998年首次在人类白细胞和大鼠肝脏中被发现[8],较其他水通道蛋白更易通透水、甘油等小分子溶质[9]。另在小鼠研究中发现AQP9主要在肝脏中表达,参与甘油转运,是血浆甘油转运的主要通道[10]。AQP9促进肝脏的甘油摄取并代谢为脂肪合成中间产物3-磷酸甘油,将脂解产生的甘油运输进肝脏,将肝脏中3-磷酸甘油运输进血浆参与脂肪合成,有效的将脂解与脂肪合成通过甘油连接起来[11]。同时,甘油在血浆与肝脏间转运的增强,也促使机体内AQP9表达量的增加[12]。另有研究表明,AQP9是氧化物酶体增殖物激活受体γ(PPARγ)的靶基因,PPARα、PPARγ作为典型的成脂相关基因,其表达量均影响AQP9的表达[13]。综上,可以推断AQP9可能是影响动物脂肪沉积的潜在基因。
核糖体蛋白(ribosomal protein, RP)是真核生物核糖体的重要组成成分,参与蛋白质的合成[14],是核糖体小亚基的组成蛋白,属于核糖体蛋白家族成员[15]。RPS10的甲基化对核糖体的蛋白质合成、细胞增殖过程的调节具有重要作用[16]。有诸多研究报道了RPS10与猪的生产性状相关,在对中国东乡斑点猪的两个外部性状(斑点毛色和面部类型)进行全基因组关联分析(GWAS)中也发现RPS10是影响面部变异的潜在基因[17]。Lü等[18-19]在研究中指出,RPS10可能与猪的骨骼生长及体型有关。将恩施黑猪的基因组库与中国野猪的全基因组结合,鉴定了嵌入到恩施黑猪基因组中的400多个蛋白质编码基因,并将包括RPS10在内的5个基因列为与猪肉脂肪沉积相关的候选基因[20]。因此,可以推测RPS10对猪的脂肪沉积有影响。
本研究所采用的北京黑猪是我国着名的培育品种,由通县猪等本地猪与大白猪杂交形成[21],其肌内脂肪含量可达到3%,肉质优良,风味独特深受消费者欢迎[22]。然而北京黑猪因含地方猪血统,背膘较厚,因此需对其背膘厚开展研究。在本课题组的前期研究中,通过对背膘厚极端差异组进行转录组分析,确定了AQP9基因为脂肪沉积相关的候选基因[23]。对大白、民猪杂交群体6~7肋间背膘厚进行GWAS,注释到SSC7上多个SNPs,并在连锁不平衡(LD)区间发现了RPS10基因[24]。上述两基因虽已被确定为影响背膘厚度的候选基因,但其在北京黑猪的多态性及与背膘厚的相关性尚不清楚。本研究通过对AQP9和RPS10基因进行突变位点检测,并开展基因型与背膘厚度性状的关联分析,以期筛选到影响北京黑猪背膘厚的候选功能位点,为今后北京黑猪背膘厚度性状的分子选育提供标记,并为背膘厚的遗传机制研究提供参考。
1 材料与方法
1.1 组织样本及表型数据收集
本研究所用413头北京黑猪均来自北京黑六牧业科技有限公司,屠宰后使用游标卡尺测量试验猪右侧胴体背部脂肪不同位置(肩部、6~7肋间、胸腰结合处、腰荐结合处)的背膘厚度,从左侧胴体采集背部脂肪样品于液氮速冻后,-80 ℃冰箱保存。
1.2 DNA提取
使用DNA提取试剂盒(QIAamp DNA Mini Kti)从组织样本中提取基因组DNA,利用超微分光光度计(IMPLEN)检测DNA浓度,并用1.5%的琼脂糖凝胶电泳进行DNA质量评估,检测合格的DNA样品存放于-20 ℃冰箱中。
1.3 RNA提取
对组织样品进行研磨,并使用RNA提取试剂盒(TaKaRa MiniBEST Universal RNA Extraction Kit)进行RNA提取,随后通过超微分光光度计(IMPLEN)检测其浓度及质量。对于合格的RNA,用反转录试剂盒(PrimeScript RT reagent Kit)进行反转录,反转好的cDNA样品置于-20 ℃冰箱保存备用。
1.4 引物设计及合成
利用美国国家生物技术信息中心(National Center for Biotechnology Information, NCBI,http://www.ncbi.nim.nih/)的基因序列对AQP9(Gene ID: 100127153)和RPS10(Gene ID: 100155444)的启动子及外显子区域共设计22对引物,部分引物见表1,设计好的引物均由北京六合华大基因科技有限公司合成。
1.5 聚合酶链式反应(PCR)及基因分型
对413头北京黑猪进行PCR扩增,反应体系:(2×Phanta Max Buffer 12.5 μL,DNA 1 μL,dNTP 0.5 μL,上、下引物各1 μL,聚合酶 0.5 μL,RNase-free水 8.5 μL)。PCR反应程序如下:95 ℃预变性5 min;95 ℃变性30 s,57~62 ℃退火30 s,72 ℃延伸30~90 s,35个循环;72 ℃延伸2 min。PCR产物经过琼脂糖凝胶电泳检测后,由北京六合华大基因科技有限公司进行Sanger测序。
1.6 AQP9和RPS10基因表达量的检测
利用NCBI(http://www.ncbi.nim.nih/)的基因序列对AQP9和RPS10基因设计荧光定量PCR引物,引物见表2,设计好的引物由北京六合华大基因科技有限公司合成。
以GAPDH基因为内参,反转录后的cDNA为模板,开展实时荧光定量PCR,检测AQP9、RPS10基因的表达。采用20 μL反应体系:10 μL TB Green,0.4 μL PCR Forward Primer,0.4 μL PCR Reverse Primer,0.4 μL ROX Reference Dye П,2 μL模板,6.8 μL 无菌水。RT-qPCR反应条件为:预变性阶段(95 ℃ 30 s);扩增阶段(95 ℃ 5 s,60 ℃ 34 s,共40循环);建立熔解曲线阶段(60~95 ℃,每10 s缓慢升温0.5 ℃)。

表1 PCR引物信息Table 1 PCR primers information
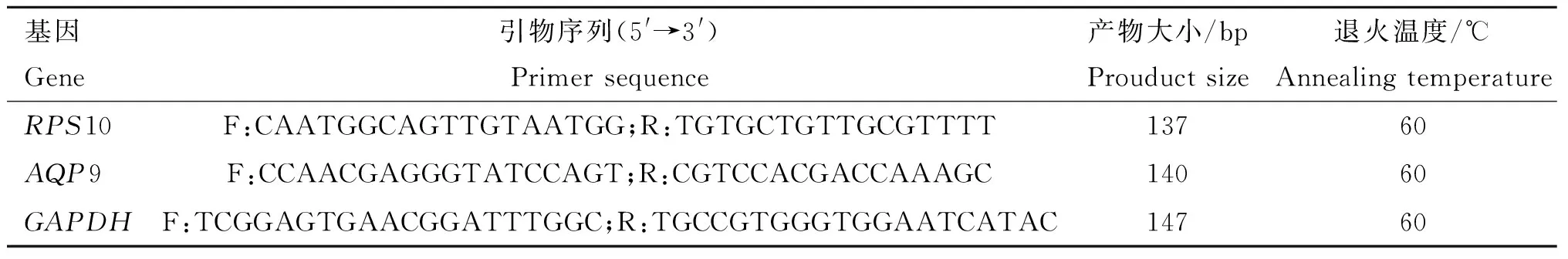
表2 RT-qPCR引物信息Table 2 RT-qPCR primers information
1.7 数据统计分析
利用Excel 2016对基因型频率和各位点的等位基因频率进行统计。分别计算各突变位点的多态信息含量、杂合度及哈代-温伯格平衡检验。使用SAS9.4软件进行不同基因型个体间背膘厚的差异显着性分析(P<0.05),采用混合线性模型,加入体重作为协变量,利用协方差分析法,具体模型如下:
Yij=μ+Gi+bW+eij
其中,Yij表示性状测定值;μ表示群体均值;Gi表示基因型效应;W表示体重协变量效应;b为协变量的回归系数;eij为随机误差。
采用2-ΔΔCt法计算AQP9、RPS10基因的相对表达量(以GAPDH基因为内参),采用T检验评估不同基因型间定量表达结果差异的显着性(P<0.05)。
1.8 突变位点功能预测
使用转录因子结合谱数据库(A database of transcription factor binding profiles, JASPAR)预测AQP9基因启动子区转录因子结合位点,及突变前后转录因子的变化。使用miRDB(miRDB-Custom Prediction)分析RPS10基因3′UTR区潜在的miRNA结合位点。
2 结 果
2.1 表型数据整理
对413头北京黑猪的肩部背膘厚、6~7肋间背膘厚、胸腰结合处背膘厚、腰荐结合处背膘厚、四点平均背膘厚的测定结果进行统计分析,均值分别为3.86、3.07、2.57、2.74、3.07 cm(表3)。

表3 北京黑猪不同部位背膘厚度性状的表型值统计Table 3 Statistics of phenotypic values of backfat thickness traits at different parts of Beijing black pigs
2.2 AQP9和RPS10基因SNP位点基因型频率及群体遗传特征
通过对AQP9和RPS10基因的启动子区及外显子区进行PCR扩增,最终在AQP9和RPS10基因共发现12个突变位点(表4和表5)。其中AQP9中有4个突变位点位于启动子区,而RPS10中有8个突变位点,包括7个突变位置在3′UTR区,1个在CDS区。AQP9和RPS10基因突变位点的基因型频率和群体遗传特性,见表4和表5。全部12个SNPs的基因型频率变化范围在1%~87%,等位基因变化频率为7%~92%。该群体中rs80862457 C>T、rs80795904 C>G、rs80932213 T>C、rs3469834461 C>T、rs3469834461 C>A显示了中等多态性 (0.25
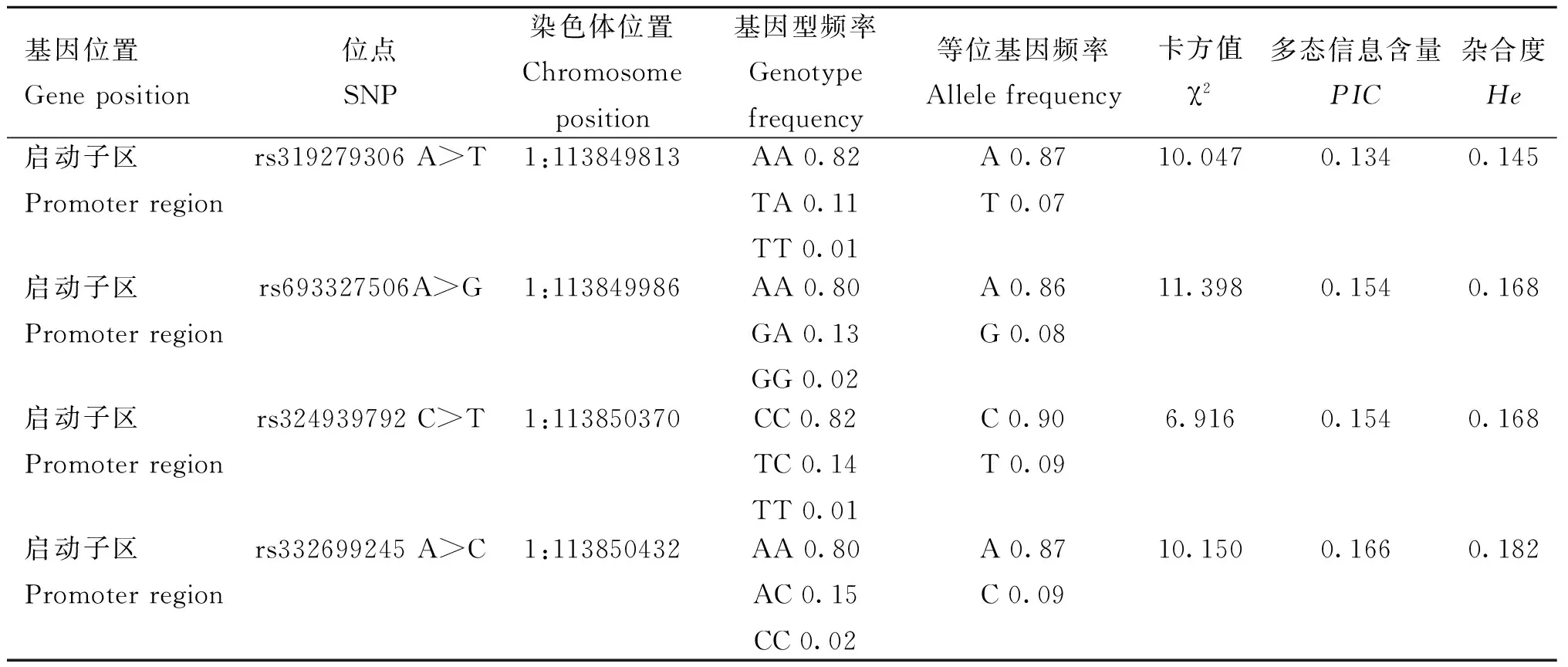
表4 AQP9基因多态位点群体遗传特性Table 4 Population genetic characteristics of AQP9 SNPs
2.3 AQP9、RPS10基因多态性和不同部位背膘厚性状的关联分析
将AQP9和RPS10基因筛选出的12个SNPs位点与猪的肩部背膘厚、6~7肋间背膘厚、胸腰结合处背膘厚、腰荐结合处背膘厚、四点平均背膘厚进行关联分析(表6和表7)。在AQP9的4个启动子区突变中,rs332699245 A>C与胸腰结合处背膘厚度显着关联(P<0.05),其余3个位点与各个部位背膘厚度关联均不显着。在RPS10的8个SNPs中,7个位于3′UTR区的突变位点中,有4个SNPs与背膘厚度显着相关(P<0.05),分别是:rs80862457 C>T与胸腰结合处背膘厚、腰荐结合处背膘厚均显着关联;rs80795904 C>G和rs80932213 T>C与6~7肋背膘厚显着关联;rs3469834461 C>T与肩部背膘厚和四点平均背膘厚均显着关联。另位于CDS区的位点rs336938272 A>C与6~7肋背背膘厚、腰荐结合处背膘厚、四点平均背膘厚均显着关联(P<0.05)。
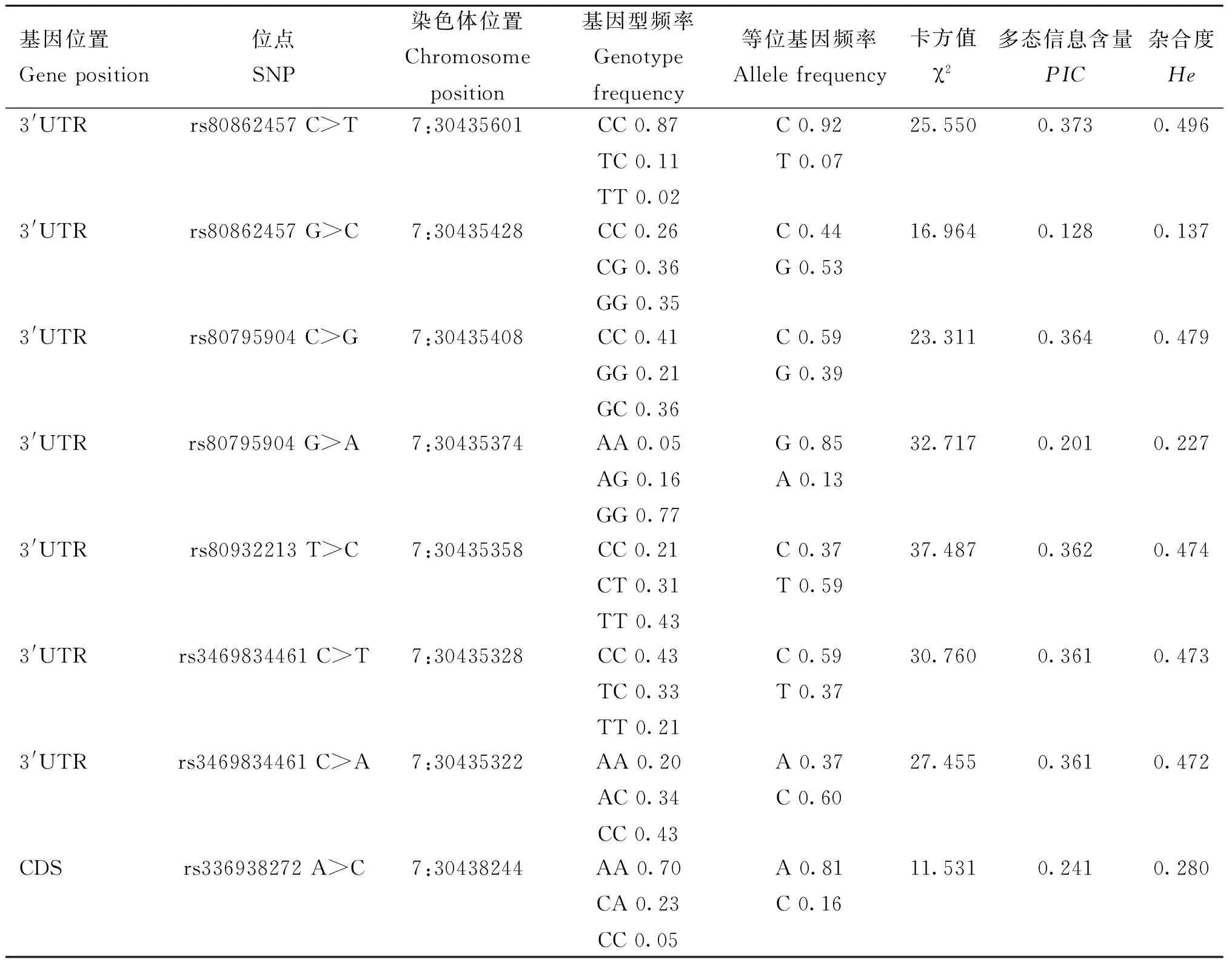
表5 RPS10基因多态位点群体遗传特性Table 5 Population genetic characteristics of RPS10 SNPs
2.4 SNPs位点突变功能分析
使用JASPAR (https://jaspar.genereg.net/)分析AQP9基因启动子区与背膘厚显着关联的SNP rs332699245 A>C潜在的转录因子(Transcription Factor,TF)结合位点。结果显示,rs332699245 A>C位于转录因子SPI1的结合位点处。
使用miRDB(miRDB-Custom Prediction)预测RPS10基因位于3′UTR区的4个与背膘厚显着关联的突变位点(rs80862457 C>T、rs80795904 C>G、rs80932213 T>C、rs3469834461 C>T)中潜在的miRNA结合位点,发现rs80862457 C>T为miR-6777-5p的结合位点。
2.5 AQP9 rs332699245 A>C和RPS10 rs80862457 C>T不同基因型个体的基因表达差异分析
根据rs332699245 A>C位点AA型和AC型个体背膘厚差异显着,利用RT-qPCR技术检测AA与AC型个体背膘组织中AQP9基因的表达情况。结果显示,AA基因型个体的AQP9表达量显着高于AC型个体(P<0.05),见图1。
根据rs80862457 C>T位点AA和CC型个体背膘厚差异显着,利用RT-qPCR技术检测了CC与TT型个体背膘组织中RPS10基因的表达情况。结果显示,TT基因型个体的RPS10表达量显着高于CC型个体(P<0.05),见图2。
3 讨 论
背膘厚对猪的胴体性状、繁殖性能、肉质性状等均有影响,是评价胴体瘦肉率、育肥效果、繁殖性能等重要性状的指标。关红民等[25]研究表明,背膘厚与胴体瘦肉率呈高度负相关,可以作为衡量瘦肉率的指标。育肥猪背膘厚增加可以提高背最长肌的肌内脂肪含量、风味和多汁性,从而影响猪肉品质[26]。此外,背膘厚在各品种间存在显着差异[27],是重要的数量性状[28]。本研究所用北京黑猪在pH、保水力、嫩度等肉质指标上相比于长白猪、大约克猪等外来瘦肉型猪种有较大优势[29],但存在中国地方猪种背膘较厚、生长速度较慢的问题[30]。因此,对北京黑猪背膘厚遗传机制的研究是必要的。
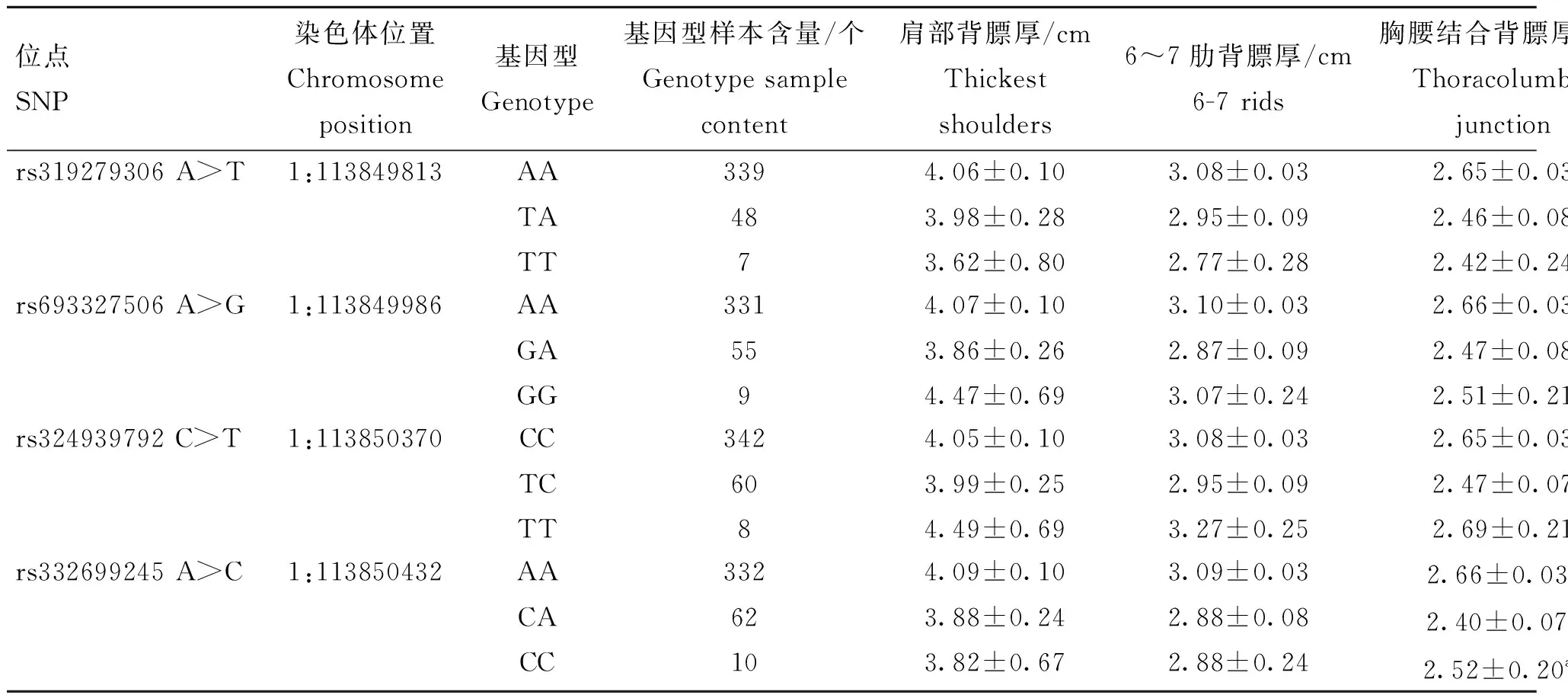
表6 AQP9基因SNPs与背膘厚度性状的关联分析Table 6 Association analysis of AQP9 gene SNPs with backfat thickness

表7 RPS10基因SNPs与黑猪背膘厚度性状的关联分析Table 7 Association analysis of RPS10 gene SNPs with backfat thickness
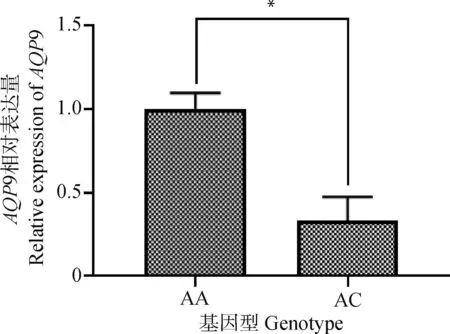
*.P<0.05。下同 *.P<0.05. The same as below图1 AQP9基因相对表达量Fig.1 Relative expression of AQP9 gene
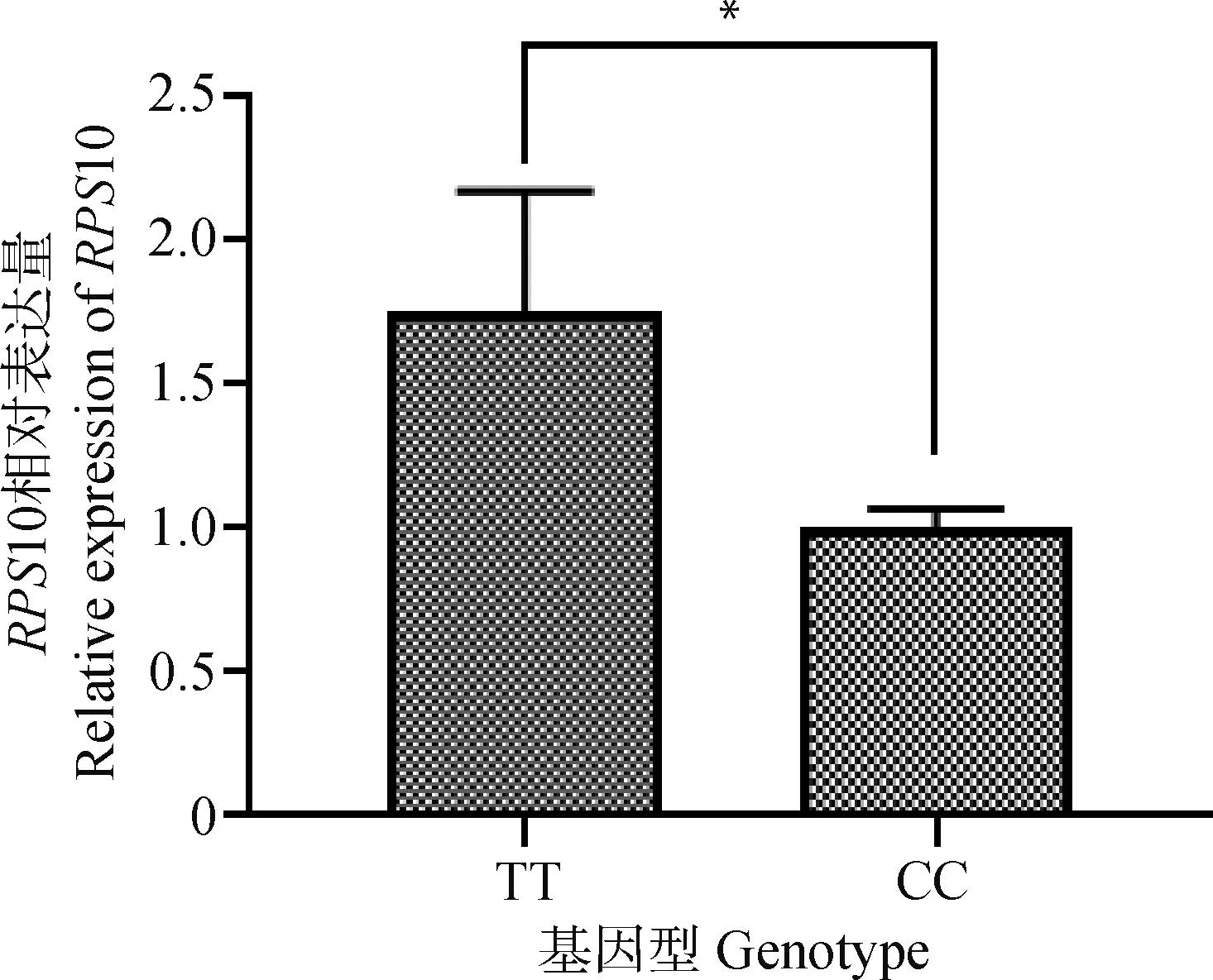
图2 RPS10基因相对表达量Fig.2 Relative expression of RPS10 gene
目前,研究人员在不同猪种上均发现了与背膘厚显着相关的SNPs。吴正常等[31]在丹系大白猪上利用55K SNP芯片对背膘厚性状进行全基因组关联分析(GWAS),筛选出2个与背膘厚显着相关的SNPs,发现4个候选基因;Zhu等[32]对一个大白、长白杂交群体进行全基因组关联分析,在7条染色体上检测到25个与背膘厚显着关联的SNPs;刘欣[33]在576头长白猪、民猪的杂交群体中发现与6~7肋背膘厚度显着相关的35个SNPs位点。本课题组前期研究中也发现了AQP9、RPS10均为影响北京黑猪背膘厚的候选基因,但并未明确上述两基因多态性与北京黑猪背膘厚的相关性关系,因此,本研究对AQP9、RPS10不同多态位点与背膘厚度进行了分析,为之后北京黑猪背膘厚的分子育种提供了理论依据。
AQP9在维持体内能量代谢与平衡、脂肪合成以及个体的生长发育等方面都具有至关重要的作用。本研究在北京黑猪群体中对AQP9基因的4个SNPs位点与背膘厚度性状进行关联分析,结果发现仅有rs332699245 A>C位点与胸腰结合处背膘厚显着相关。基因表达差异分析表明,rs332699245 A>C中AQP9基因mRNA水平表达量AA基因型个体显着高于AC基因型个体。脂肪的生成及分化受到多种转录因子(transcription factor,TF)的影响,转录因子(TF)是人类中存在的最大的蛋白质组之一,TF直接控制基因表达。AQP9启动子区的rs332699245 A>C被预测为TF结合位点,该位点的突变导致出现新的转录因子SPI1的结合位点。转录因子沙门氏菌致病岛1(SPI1)是ETS家族的重要成员,它首先在弗里德氏小鼠红细胞白血病中被鉴定为一个促癌基因[34]。SPI1是一种造血转录因子,经鉴定可促进糖酵解过程,通过促进有氧糖酵解促进癌症进展,该转录因子通过影响糖酵解,进而影响脂肪的合成[35]。但其在猪上的功能尚不清楚,仍需后续研究。
据报道,RPS10与猪的生长性状(肌肉生长、脂肪沉积)有强的相关性[20]。本研究在北京黑猪群体中对RPS10基因的8个位点与各部位背膘厚度性状进行关联分析,并发现5个(rs3469834461 C>T、rs80932213 T>C、rs80795904 C>G、rs80862457 C>T、rs336938272 A>C)SNPs与背膘厚显着关联。基因表达差异分析表明,rs80862457 C>T不同基因型个体背膘组织中RPS10基因mRNA表达量在TT基因型个体显着高于CC基因型个体。在RPS10的3′UTR区域的突变位点(rs80862457 C>T)通过预测被确定为潜在的miR-6777-5p的结合位点。miRNAs是真核生物中具有竞争性和靶向调控作用的非编码RNA[36],长度约为19~25个核苷酸[37],大多数的miRNA位于蛋白编码基因的3′UTR区域[38]、内含子区域,参与生物胚胎发育、脂质代谢、神经调节等多种调控过程[39]。miRNA主要是通过与靶基因mRNA碱基配对达到使mRNA降解或阻碍蛋白质翻译,调控个体的生理活动[40]。有研究表明,miRNA是肌肉和脂肪组织代谢平衡的关键调节因子[41],也可作为脂代谢调节的潜在媒介[42],主要通过抑制脂代谢靶基因的翻译从而影响脂肪、胆固醇的合成等过程。在脂肪生成过程中可以加速或抑制脂肪细胞分化,调节脂肪发育[43],miR-122-5p、miR-142-3p、miR-143等已被报道是脂肪沉积的调节因子[44-45]。miR-6777-5p在人类青光眼的临床症状中被观察到,并被证明参与了青光眼发病机制的调控[46]。但其在猪上的功能研究尚不完全,仍需后续完善。
4 结 论
通过对413头北京黑猪开展AQP9和RPS10基因多态性与背膘厚性状的关联分析,共发现12个SNPs位点,其中AQP9基因内发现4个SNPs,RPS10基因内发现8个SNPs。关联分析结果表明,AQP9基因内1个SNP(rs332699245 A>C)与背膘厚显着关联;RPS10基因内5个SNPs(rs3469834461 C>T、rs80932213 T>C、rs80795904 C>G、rs80862457 C>T、rs336938272 A>C)与背膘厚显着关联。AQP9基因rs332699245 A>C和RPS10基因rs80862457 C>T不同基因型个体间基因表达差异显着。本研究结果为猪AQP9、RPS10基因调控背部脂肪含量的分子机理提出了新见解,以期探索调控性状变异的候选基因功能位点,为进一步阐述性状变异的遗传机理奠定基础,为北京黑猪的分子遗传育种提供理论参考。



