严晓春,习海娇,李金泉,王志英,苏 蕊
(内蒙古农业大学动物科学学院,呼和浩特 010018)
内蒙古绒山羊是经过长期自然选择和人工选育形成的优良畜种,其生产的羊绒纤维纤细、柔软、有光泽,用它制成的羊绒制品精美绝伦,手感密实柔软,温暖舒适,享誉海内外[1]。近年来,随着国内外绒毛市场的发展,内蒙古绒山羊作为自治区的特色产业,具有很好的产业优势,随着时代的进步,人民生活水平不断提高,市场对绒山羊产品的需求发生了改变,因此,需要制定更为合理的育种方案提升绒山羊绒毛产量。
SSGBLUP法最早由Legarra等[2]提出,其核心在于利用基因组、系谱和表型数据构建H矩阵进行育种值估计。在大多数物种上,例如猪[3-5]、鸡[6-8]、牛[9-11]等,均通过比较BLUP、GBLUP和SSGBLUP模型估计育种值的准确性,证明了SSGBLUP的选育效果相对较好。Zhao等[12]利用GBLUP 和SSGBLUP方法对杜洛克公猪的5种精子形态异常进行基因组预测,发现SSGBLUP的预测能力高于GBLUP。Lourenco等[13]在安格斯牛群体中的研究结果表明,SSGBLUP方法估计育种值准确性方面优于BLUP方法。Gao等[14]在黄羽肉鸡群体的研究中,发现SSGBLUP方法优于BLUP和GBLUP方法。近年来,绒山羊的基因组选择开展了一定的工作,但尚未对用于SSGBLUP法H矩阵的尺度参数进行测试与优化。
本研究充分利用内蒙古阿尔巴斯型绒山羊的系谱数据、基因型数据以及羊绒性状的表型和环境数据记录,建立动物混合线性模型,对SSGBLUP法中H矩阵的两个参数ω和τ设定不同的参数组合,进行遗传参数和基因组育种值的估计,采用五倍交叉验证法对基因组选择准确性进行评价,筛选并确定用于内蒙古绒山羊羊绒性状基因组育种值估计的SSGBLUP法H矩阵构建最适参数。本研究旨在通过确定SSGBLUP法中H矩阵中ω和τ的最适参数组合,提高内蒙古绒山羊绒毛性状的基因组选择准确性,加快群体遗传改良,进一步提高牧民的经济效益。
1 材料与方法
1.1 系谱及表型数据
本研究所用系谱数据来源于内蒙古亿维白绒山羊有限责任公司内蒙古绒山羊(阿尔巴斯型)群体,共24 791个体(由216父本和5 257母本交配产生),所有个体系谱清晰,记录准确详实。所研究性状包括绒长、绒细、产绒量,共2 256个个体包含从1岁到8岁的24 023条表型记录,根据数据特征,剔除平均数加减3倍标准差两尾的数据,其余保留用于后续分析。
1.2 基因型数据
本研究所用基因型数据为课题组前期积累的基于Illumina GGP_Goat_70K BeadChip芯片测序的内蒙古绒山羊(阿尔巴斯型)2 299只个体的基因型数据[15],获得的原始数据进一步使用PLINK软件v1.90b6.21对其进行质量控制,质控标准为[15]:最小等位基因频率小于0.05、Hardy-Weinberg平衡P值小于10-5、位点缺失率大于10%、个体缺失率高于10%。根据以上标准,去除不合格个体以及位点,最终保留了2 256个个体共50 728个SNPs位点。
1.3 遗传参数及基因组育种值的估计
基于已获得的系谱、表型和基因型数据,同时结合环境效应(测定年份:2011年至2021年;性别:公畜和母畜;场:1~12;个体年龄:1~8岁),建立重复力动物模型,利用SSGBLUP法对各绒毛性状进行遗传参数和基因组育种值的估计,模型方程式如下:
y=μ+Xb+Zu+Wp+e
其中,y为具有基因型信息个体的性状观测值,μ为性状观测值均值,b为性状的固定效应,u为个体加性遗传效应,p为个体永久环境效应,X为固定效应的结构矩阵,Z为个体加性效应的结构矩阵,W为个体永久环境效应的结构矩阵,e为性状的残差效应。
对于SSGBLUP方法,通常使用H矩阵建立亲缘关系矩阵,H矩阵是将系谱关系矩阵A阵和基因组关系矩阵G阵结合而形成。H矩阵[16]的构建如下:
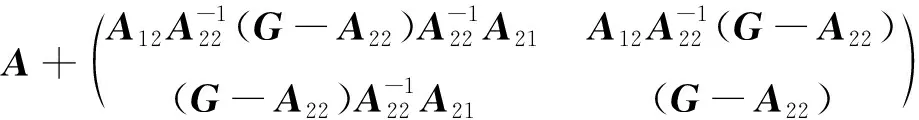
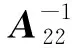
如何将A和G有效结合提高基因组育种值估计准确性已经从理论上进行了探讨[17, 21],重点关注两个尺度参数τ 和 ω,用于缩放基因组信息,使其与谱系信息兼容。本研究通过设置H逆矩阵的不同尺度参数τ(0.1~1)和ω(0.1~1)共26个组合,利用SSGBLUP法对每一个组合下个体的绒毛性状进行遗传参数和基因组育种值的估计。其中,引入尺度参数τ 和 ω后H逆矩阵的公式可转换为[22-24]:
1.4 基因组育种值估计准确性评价
本研究利用五倍交叉验证法评价内蒙古绒山羊(阿尔巴斯型)个体绒毛性状的基因组育种值预测效果,即将研究群体随机分为5组,选择其中一组具有基因型的个体作为验证群体,其他具有基因型和表型数据的个体作为参考群体,共重复循环5次。通过计算验证群体的校正表型值和估计育种值的相关除以遗传力的开方来进行基因组育种值准确性评价。
其中,cov(a,p)是性状估计育种值和校正表型值的协方差,h2是性状的估计遗传力。
2 结 果
2.1 内蒙古绒山羊绒毛性状表型记录统计分析
本研究共统计了2 256只个体绒毛性状的表型记录,对性状的最大值、最小值、平均值、标准差和变异系数进行描述性统计,结果见表1。由表可知,绒长、产绒量和绒细的平均值分别为6.25 cm,740.30 g和15.23 μm;产绒量性状的标准差和变异系数最大,分别为215.19 g和29.07%,绒细性状的标准差和变异系数最小,分别为0.81 μm和5.32%。
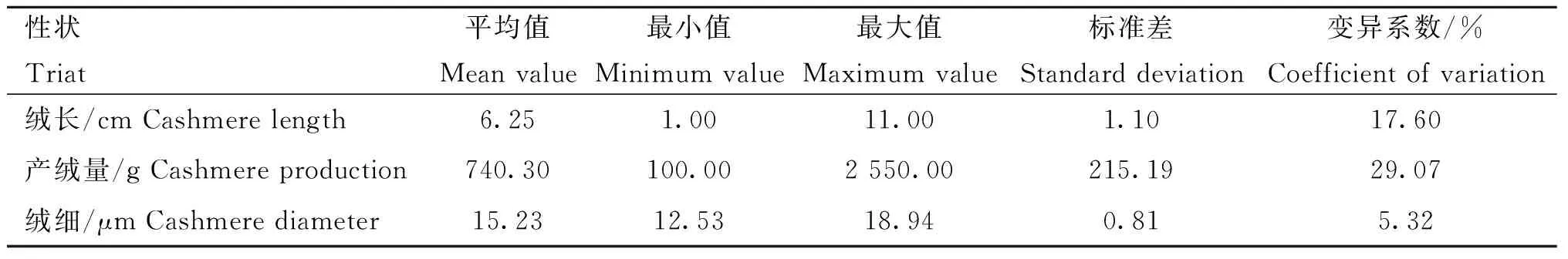
表1 内蒙古绒山羊绒毛性状表型记录的基本统计Table 1 The basic statistics of phenotype records of fleece traits in Inner Mongolia cashmere goats
2.2 不同参数组合下内蒙古绒山羊绒长遗传参数及基因组选择育种值准确性评价
对于不同尺度参数组合,SSGBLUP法估计的内蒙古绒山羊绒长性状的基因组育种值估计准确性见表2和图1。研究结果表明,基因组育种值估计准确性在不同参数组合存在较大差异。随着ω的增加和τ的减少,内蒙古绒山羊基因组育种值估计准确性逐渐增加。由于一个性状的遗传力通常是稳定的,因此结合前期ABLUP和GBLUP法估计绒长遗传参数的结果认为[15],当τ为0.3、ω为0.9时,SSGBLUP法用于内蒙古绒山羊绒长性状的遗传评估具有一定的准确性,其育种值估计的准确性为0.702 8。该条件下,对应绒长性状的遗传力和重复力均为0.05。
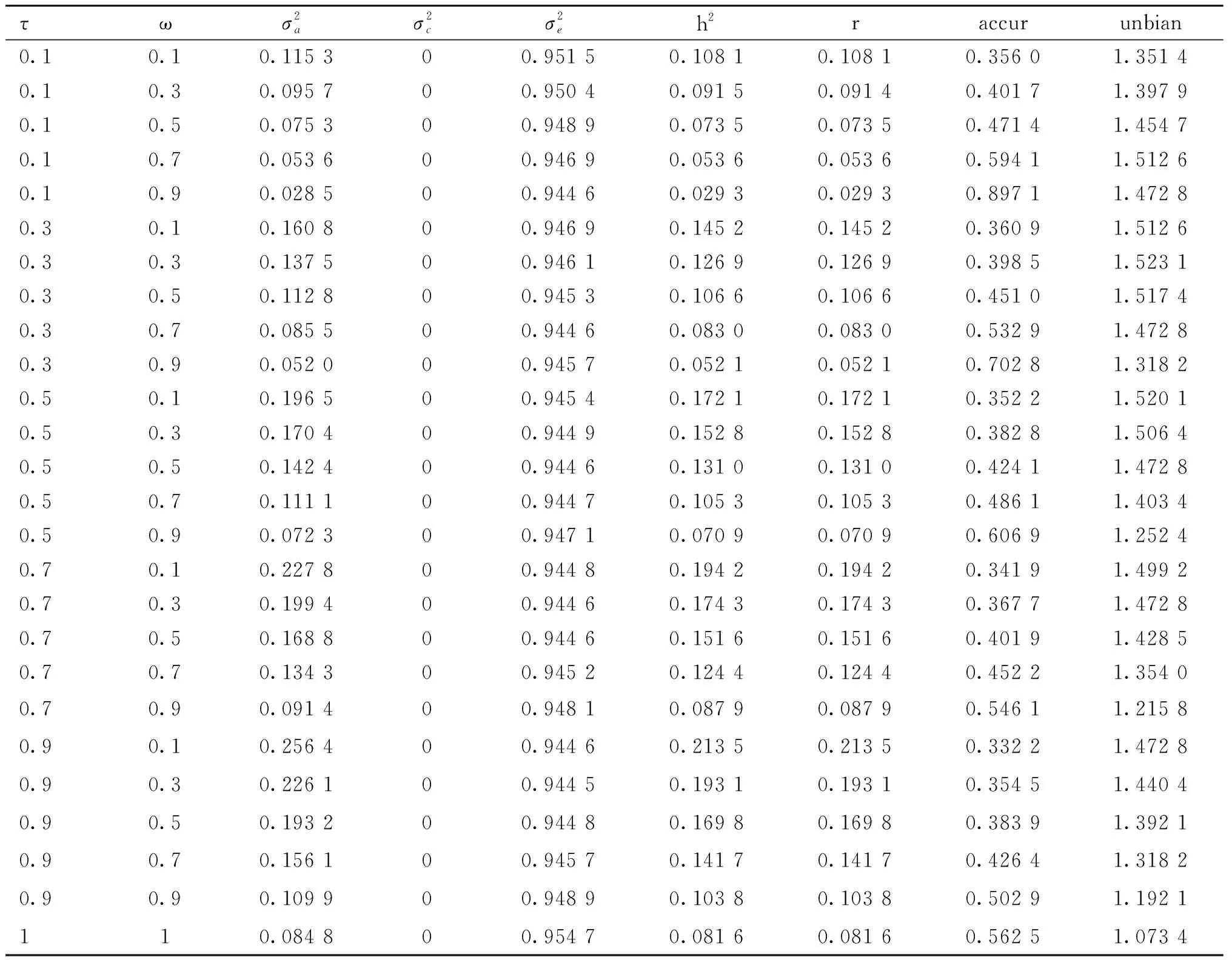
表2 不同参数下内蒙古绒山羊绒长的遗传参数和基因组育种值估计准确性Table 2 Genetic parameters and accuracy of GEBV of cashmere length in Inner Mongolia cashmere goats with different parameters
2.3 不同参数组合下内蒙古绒山羊绒细遗传参数及基因组选择育种值准确性评价
对于不同尺度参数组合,SSGBLUP法估计的内蒙古绒山羊绒细性状的基因组育种值估计准确性见表3和图2。研究结果表明,基因组育种值估计准确性在不同参数组合存在较大差异。随着ω的增加和τ的减少,内蒙古绒山羊绒细性状的基因组选择准确性逐渐增加。由于一个性状的遗传力通常是稳定的,因此结合前期ABLUP和GBLUP法估计绒细遗传参数的结果认为[15],当τ为0.3、ω为0.9时,SSGBLUP法用于内蒙古绒山羊绒细性状的遗传评估具有一定的准确性,其育种值估计的准确性为0.668 2。该条件下,对应绒细性状的遗传力为0.22,重复力为0.24。
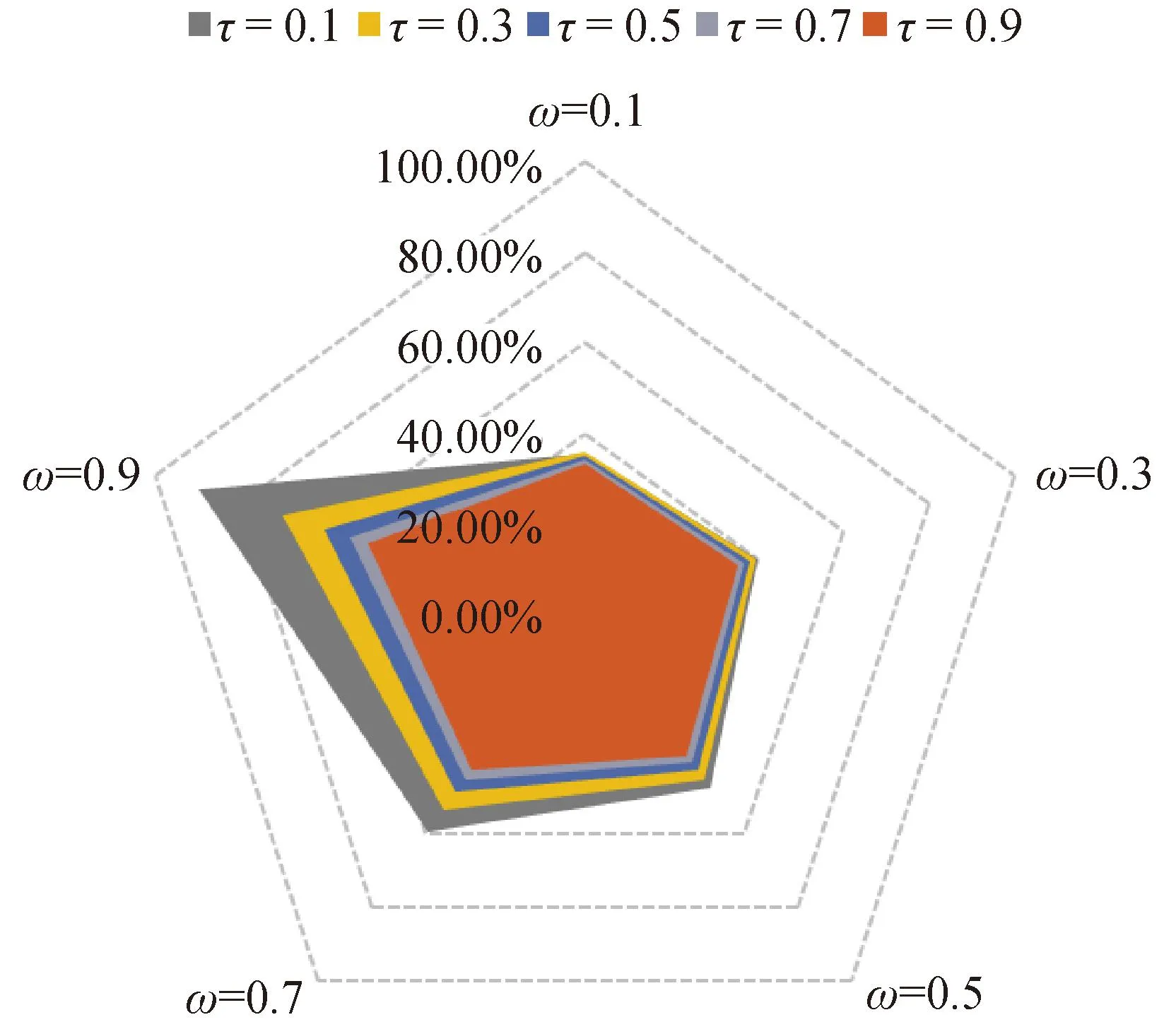
图1 不同参数下内蒙古绒山羊绒长性状基因组育种值估计准确性Fig.1 Accuracy of GEBV of cashmere length in Inner Mongolia cashmere goats with different parameters
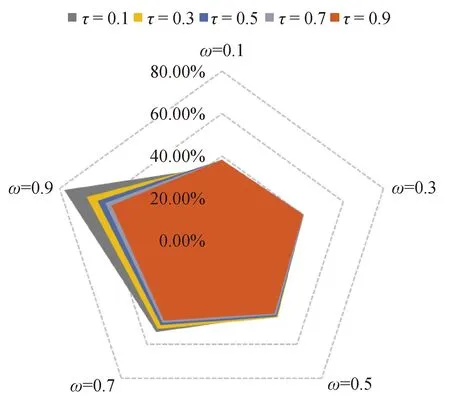
图2 不同参数下内蒙古绒山羊绒细性状基因组育种值估计准确性Fig.2 Accuracy of GEBV of cashmere diameter in Inner Mongolia cashmere goats with different parameters
2.4 不同参数组合下内蒙古绒山羊产绒量遗传参数及基因组选择育种值准确性评价
对于不同尺度参数组合下,SSGBLUP法估计的内蒙古绒山羊产绒量性状的基因组育种值估计准确性见表4和图3。同样,研究结果表明基因组育种值估计准确性在不同参数组合存在较大差异。随着ω的增加和τ的减少,内蒙古绒山羊基因组育种值估计准确性逐渐增加。结合前期ABLUP和GBLUP法估计产绒量的遗传参数的结果认为[15],当τ为0.3、ω为0.9时,SSGBLUP法用于内蒙古绒山羊绒长性状的遗传评估具有一定的准确性,其育种值估计的准确性为0.713 1。该条件下,对应产绒量性状的遗传力为0.15,重复力为0.26。
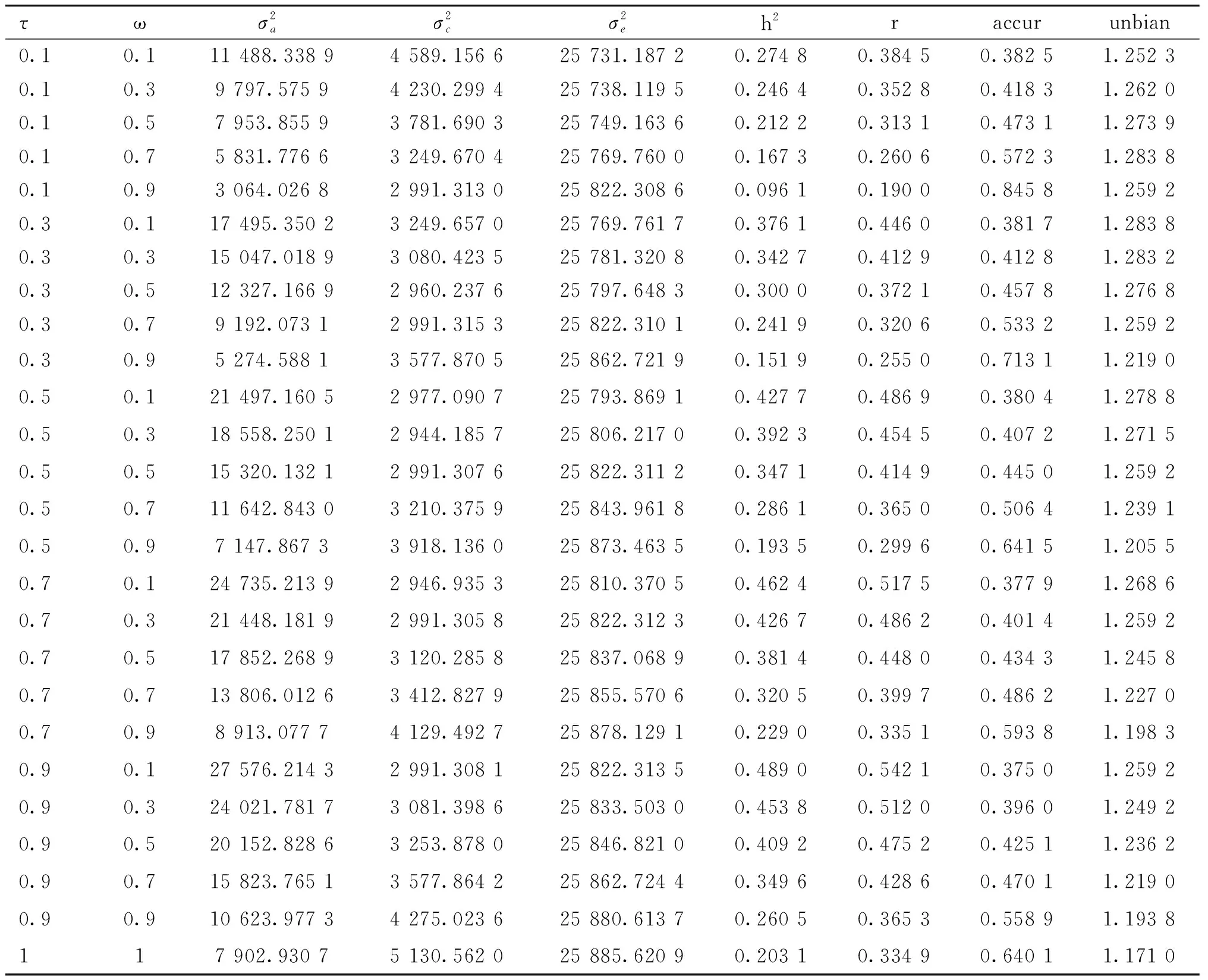
表4 不同参数下内蒙古绒山羊产绒量的遗传参数和基因组育种值估计准确性Table 4 Genetic parameters and accuracy of GEBV of cashmere production in Inner Mongolia cashmere goats with different parameters
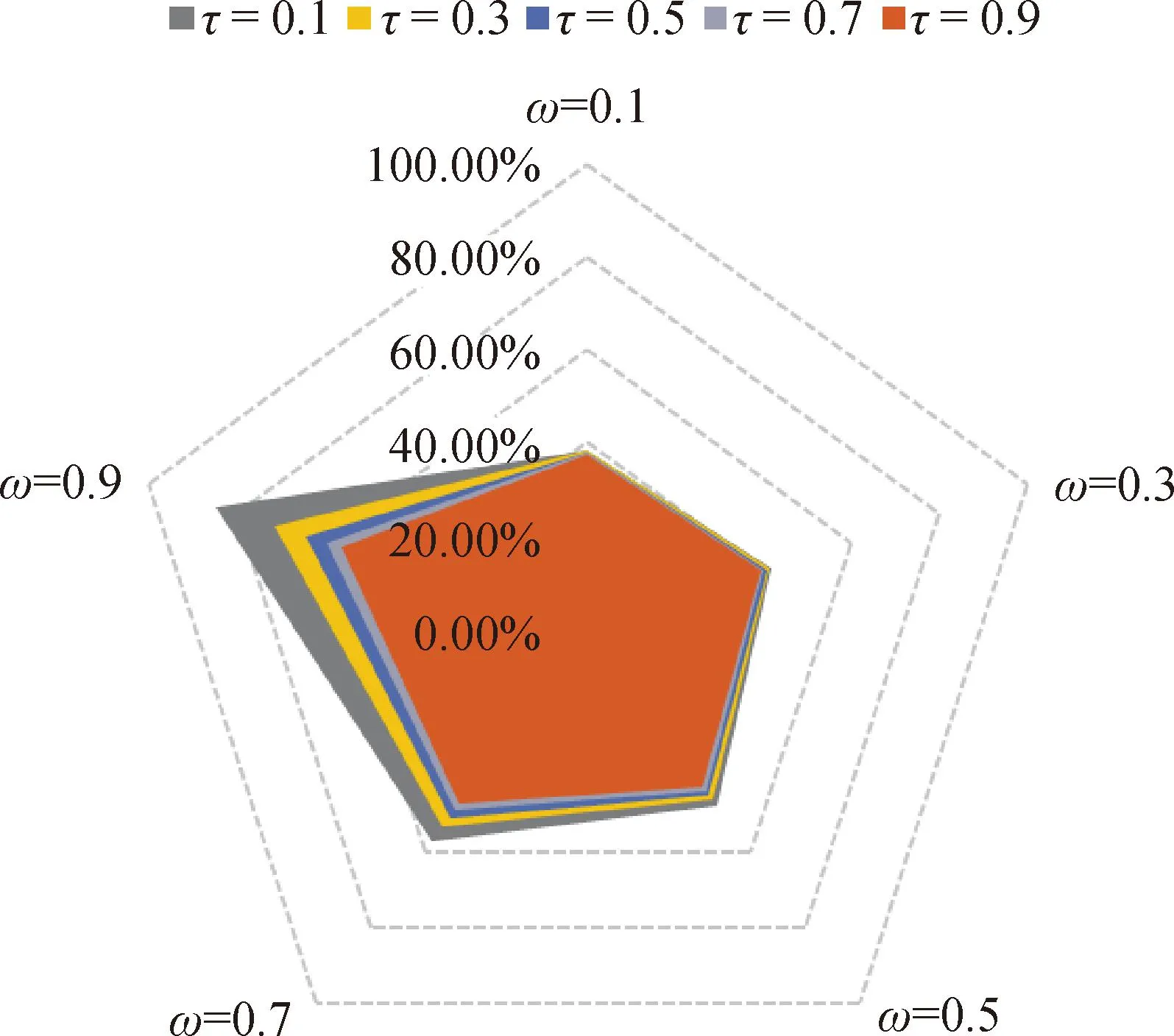
图3 不同参数下内蒙古绒山羊产绒量性状基因组育种值估计准确性Fig.3 Accuracy of GEBV of cashmere production in Inner Mongolia cashmere goat’s with different parameters
3 讨 论
SSGBLUP是一种基于一步法的遗传评估和选择方法,可以将系谱信息和基因组信息整合起来,并使用多个来源的信息来估计性状基因组育种值的模型,对基因组和系谱信息进行权重设置,得到每个个体的基因组育种值的预测值。因此,SSGBLUP法可以更准确地估计遗传参数,提高育种效果预测的准确性。一般来说,SSGBLUP比 GBLUP 的准确性更高。基因组育种值的准确性也与参考群体规模和系谱数据完整性呈正相关,当参考群体规模和系谱数据完整性增加时,基因组育种值的准确性也随之增加。并且SSGBLUP法对数量性状的基因组选择具有显着优势。Gao等[25]在芬兰红牛群体中的研究结果表明,SSGBLUP法比传统BLUP法准确性提高了2%~3%。Guillaume等[26]在荷斯坦奶牛群体中的研究结果表明,SSGBLUP方法估计育种值准确性优于BLUP和GBLUP 方法估计的育种值准确性。大量使用SSGBLUP方法对不同动物(如肉牛[27-30]、奶山羊[31-32]、奶羊[33]猪[34-35])的体尺性状进行育种值准确性评估的研究结果,均表明SSGBLUP方法的预测准确性与GBLUP方法相比更高。在奶山羊中,SSGBLUP在产奶性状、乳用性状和体细胞评分方面的表现优于BLUP或GBLUP法,并且在乳用性状的基因组预测准确性方面提高了61%~96%[36]。此外,Oget等[33]研究表明,对于乳用绵羊的乳产量和乳蛋白含量性状,SSGBLUP法比基于家系BLUP法的准确性更高。
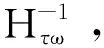
本研究结果表明,绒长性状的遗传力为0.05,属于低遗传力性状。绒细和产绒量性状的遗传力分别为0.22和 0.15,属于中等偏低遗传力性状,与王志英[38]对内蒙古绒山羊绒毛性状单性状动物模型估计的绒长和绒细的结果相似。遗传力分别是0.17 和0.34,分别为低遗传力和中等遗传力,与王凤红[15]SSGBLUP法估计内蒙古绒山羊绒长和绒细的遗传力结果一致。
4 结 论
本研究以内蒙古绒山羊绒毛性状为研究对象,通过设置SSGBLUP法H矩阵的尺度参数进行遗传参数和基因组育种值估计,发现τ为0.3、ω为0.9时,绒长、绒细和产绒量的遗传力估计值相对可靠,对应的基因组育种值估计准确性分别为0.702 8、0.668 2和0.713 1,是目前估计准确性最高的方法,可用于后续开展绒毛性状的基因组选择,加快绒山羊种群的遗传改良,缩短世代间隔。



