姚 松,梁 鹏,涂清鲜,姜黔峰
(1.遵义医科大学第三附属医院 心内科,贵州 遵义 563000;2.遵义医科大学 第一临床学院,贵州 遵义 563099;3.贵州航天医院,贵州 遵义 563000)
急性心肌梗死目前仍是全球主要的死亡原因之一,全球每年发病人数约有700万人且呈逐年上升趋势[1]。由于冠状动脉粥样斑块形成,供血不足诱发心肌缺血缺氧继而心肌坏死,发生心律失常、心源性休克或者心力衰竭等,可危及生命并给社会和家庭造成严重经济负担[2]。尽管急性心肌梗死在诊断手段与治疗途径上取得了突破性进展,但是其确切发病机制尚需进一步阐明。
近年来,多种形式的细胞死亡已证实与心血管疾病相关[3],铁死亡是急性心肌梗死后新发现的程序性细胞死亡方式之一,然而急性心肌梗死的铁死亡机制尚未阐明。动脉粥样硬化患者体内铁水平升高,铁过载可以促进氧化应激、脂质过氧化等病理过程的发生,脂质过氧化及铁沉积是动脉粥样硬化斑块后期的重要特征[4],心肌细胞铁死亡介导的氧化应激,胰岛素抵抗为心肌梗死的重要病理特点[5]。有研究发现,铁蛋白缺失的小鼠心肌细胞内心脏铁含量下降,氧化应激升高,铁调节基因SLC7A11的过表达可以阻断铁死亡,减少心肌细胞损伤[6]。铁死亡抑制剂通过减少线粒体活性氧的产生,对受损心肌起保护作用,证明了铁死亡对心肌的显着影响。铁死亡相关基因有可能成为心血管疾病新的生物标志物或者治疗靶点,然而,仍有许多铁死亡基因尚未被发现,因此需要更多的研究来探索铁死亡相关基因。
随着生物数据检测技术的不断发展,高通量测序平台及研究方法的产生已广泛应用于分子生物学领域,生物信息学已迈入大数据时代,大量的数据已经在公共数据库平台上发布,整合这些数据库可以更加深入地研究分子机制。目前应用生物信息学技术研究急性心肌梗死后铁死亡基因机制较少,为此,本研究采用数据挖掘和数据分析的方法,对急性心肌梗死患者与正常人的差异表达基因(DEGs)进行筛选,然后将这些DEG与Ferroptosis数据集相交以获得铁死亡差异基因(FerroptosisDEG)。另外,为明确关键生物标志物,从分子水平明确急性心肌梗死发病机制,对可能在急性心肌梗死中发挥主要功能的重要miRNA进行了研究。筛选急性心肌梗死后铁死亡基因,为临床诊治急性心肌梗死提供新思路。
1 材料与方法
1.1 数据来源 通过美国国立生物技术信息中心(National center of biotechnology information,NCBI)的基因表达数据库下载数据集GSE66360的基因表达数据,最终纳入49例急性心肌梗死患者和50例正常人群。
1.2 铁死亡相关基因的确定 根据 FerrDb 数据库(http://www.zhounan.org/ferrdb/)中提供的铁死亡相关基因,将 259 个基因作为后续分析用的待选基因。
1.3 差异表达基因的筛选及 GO 和 KEGG 富集分析 首先使用GEO 数据库自带的 GEOR2 在线网络分析工具对数据集 GSE66360进行数据分析,筛选|log2FC|≥1且调整后的P值 <0.05的基因作为候选差异表达基因,差异基因用火山图表示。并将其与259个铁死亡相关基因取交集,使用在线工具Venny2.1.0生成Ferroptosis DEGs的Venn图。使用 DAVID工具对 27 个 Ferroptosis DEGs进行基因本体(gene ontology,GO)和通路富集分析(kyoto encyclopedia of genes and genomes,KEGG);GO 分析包括生物学过程(biological process,BP)、细胞成分(cellular component,CC)和分子功能(molecular function,MF)。
1.4 蛋白质相互作用网络的构建及关键基因的筛选 使用在线数据库 STRING(https://string-db.org/),对组织中蛋白质间的相互作用进行分析,构建Ferroptosis DEGs的蛋白质相互作用 (protein-protein interaction,PPI) 网络。根据MCODE算法计算的得分,将PPI网络分为一个簇。再根据MCC、DMNC、BETWEEN、DEGREE、EPC算法分析取交集做为关键基因,利用GeneMania工具来预测关键基因与其功能相似的基因功能和可能的相互作用网络。
1.5 基因-miRNA相互作用网络 使用 miRWalk 2.0 预测目标关键 miRNA,并构建与关键基因-miRNA 相互作用网络。对 MiRTarBase 和 mirWalk 数据库的预测结果取交集,筛选了靶向2个以上基因的 miRNA。
1.6 样本采集 本研究经遵义医科大第三附属医院医学伦理委员会批准,编号为伦审[2022]-161号,共有 10 名 AMI 患者和10 名对照者参加了该研究。在排除有恶性肿瘤、严重感染、肝肾功能不全等可能影响检测结果的因素后,收集了入院后第一次血液检查的结果。AMI的诊断标准基于最新的临床指南(2022)。
1.7 酶联免疫吸附测定 用 ELISA 试剂盒检测正常对照组及急性心肌梗死组的TLR4和ATF3表达水平。步骤如下:① 设置标准品孔和样本孔,标准孔各加不同浓度的标准品50 μL。②分别设空白孔(空白对照孔不加样品及酶标试剂,其余各步操作相同)、待测样品孔。在酶标包被板上待测样品孔中先加样品稀释液40 μL,然后再加待测样品10 μL(样品最终稀释度为5倍)。将样品加于酶标板孔底部,尽量不触及孔壁,轻轻晃动混匀。③37 ℃温育 30 min后弃去液体,甩干,每孔加满洗涤液,静置 30 s后弃去,如此重复 5次。④加入酶标试剂 50 μL,空白孔除外,再次温育和洗涤。⑤先加入显色剂 A 50 μL,再加入显色剂 B 50 μL,轻轻震荡混匀,37 ℃避光显色 15 min。⑥每孔加终止液 50 μL,终止反应。⑦以空白孔调零,450 nm 波长依序测量各孔的吸光度(OD 值)。
1.8 统计学分析 比较AMI 组和对照组之间的统计学差异,使用GraphPad Prism 9 软件进行秩和检验进行统计分析,P<0.05 为差异有统计学意义,并完成接受者操作特征(ROC)曲线的构建和ROC曲线下面积(AUC)的计算。
2 结果
2.1 急性心肌梗死中铁死亡相关差异表达基因的鉴定 GEO2R是一种在线分析工具,用于进行差异表达分析。比较急性心肌梗死患者和正常人的表达谱以鉴定 DEG。使用以下标准选择:|log2FC| ≥ 1 且调整后的P<0.05,共筛选出差异基因 658个,其中上调基因 463 个,下调基因194个。差异基因用火山图表示(图1)。还从 Ferroptosis 数据库中获得了1个包含 259 个基因的数据集,并将其与GSE 66360相交以识别 Ferroptosis DEGs。使用在线工具Venny2.1.0生成DEGs的Venn图(图2),并对筛选出来的Ferroptosis DEGs进行分类(表1)。
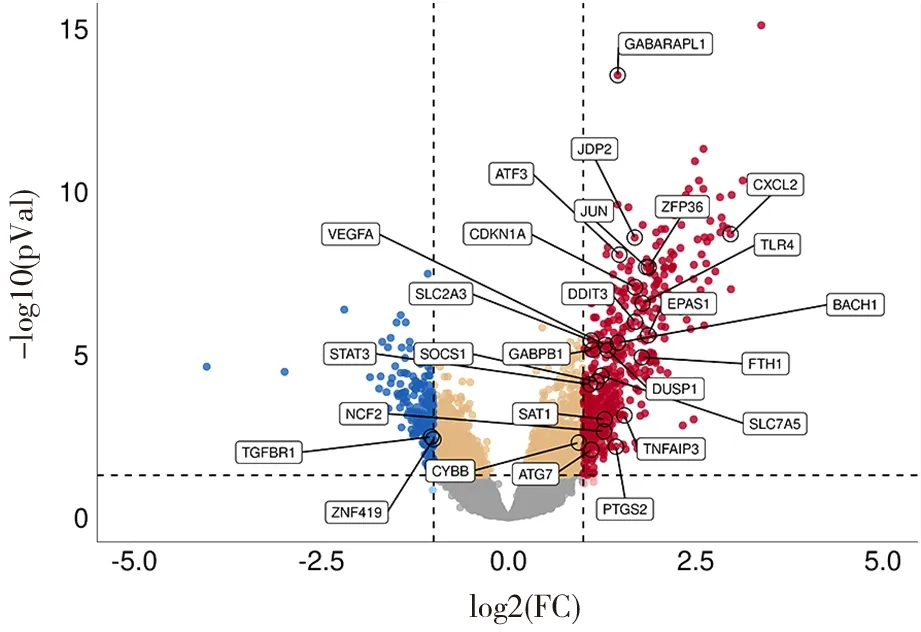
图1 铁死亡相关差异基因
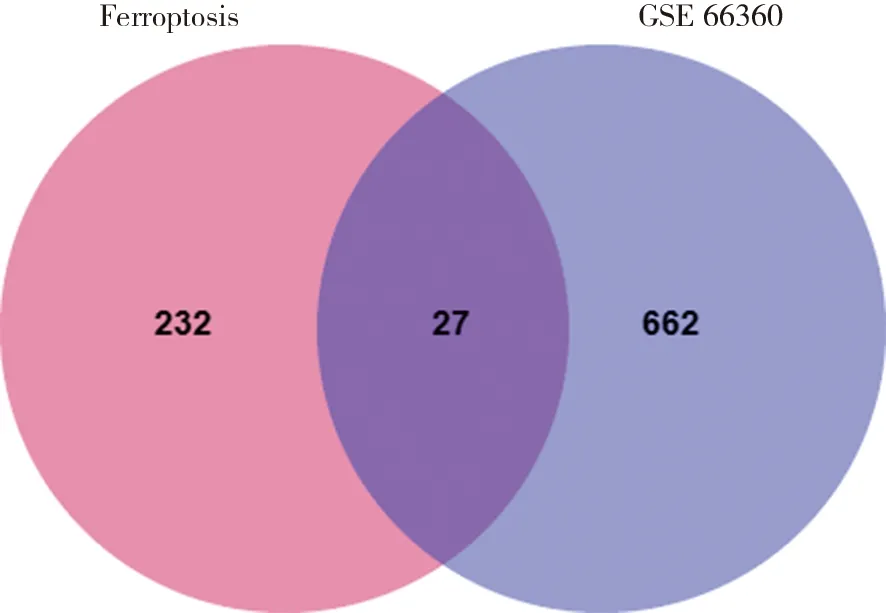
图2 铁死亡相关交集基因

表1 Ferroptosis DEGs基因所属的基因调节类别
2.2 差异表达基因功能富集分析 为了探索ferroptosis DEGs潜在的生物学功能,通过DAVID在线工具进行GO富集分析和KEGG通路分析,这些基因涉及生物学过程主要是对RNA聚合酶Ⅱ启动子转录调控、炎症反应的调节、凋亡过程的正调控;分子功能包括蛋白质结合、DNA结合、转录因子结合等,细胞组分显示这些基因主要参与细胞核、细胞质、质膜、染色质等。此外,KEGG通路分析的主要途径是坏死凋亡、脂质氧化和动脉粥样硬化等。然后将27个ferroptosis DEGs上传到 Metascape进行GO富集分析和KEGG通路分析,所得结论基本一致(图3)。上述结果表明,Ferroptosis DEGs和AMI之间存在密切的相关性,主要介导炎症、氧化应激、坏死和凋亡、脂质与动脉粥样硬化。
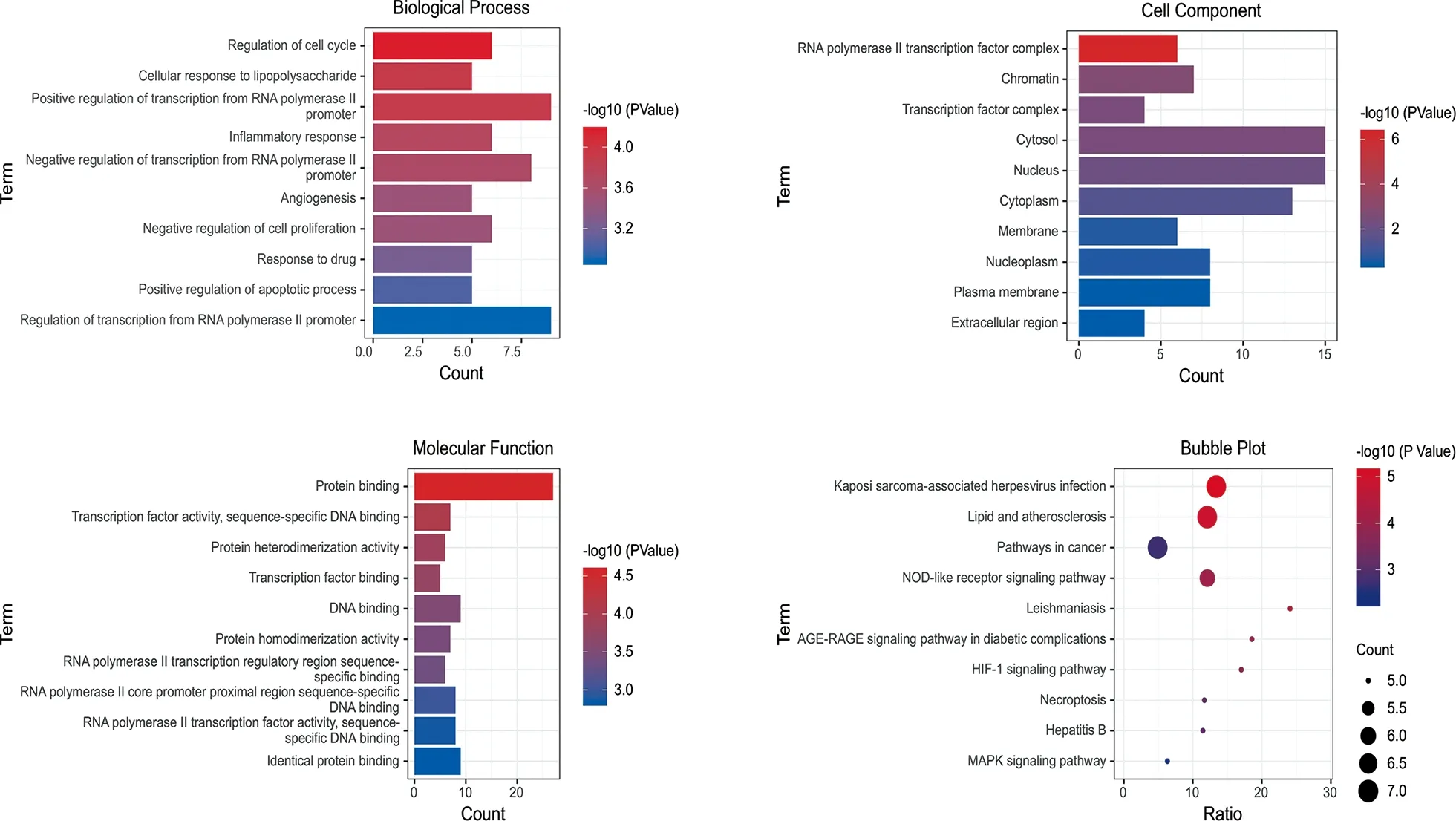
图3 GO、KEGG 功能富集分析结果
2.3 PPI网络构建、模块化分析 为了探索基因的相互作用,使用 STRING 数据库构建了蛋白质-蛋白质相互作用网络。然后,将可视化交互分数设置0.4为阈值,作为构建 STRING PPI 网络所需的最低交互分数。PPI 网络由27个节点和 79条边组成(图4)。27个基因中有4个与其他分子无关,也没有形成分子网络。使用 Cytoscape 中的 MCODE 插件,确定了1个模块(图5),包括 9个节点和 68 条边,cytohubba插件中MCC、DMNC、MNC、Degree和EPC 5种算法获得的前10个hub基因(表2),通过维恩图获取了5种算法中重叠的中心基因,包括TLR4和ATF3(图6)。
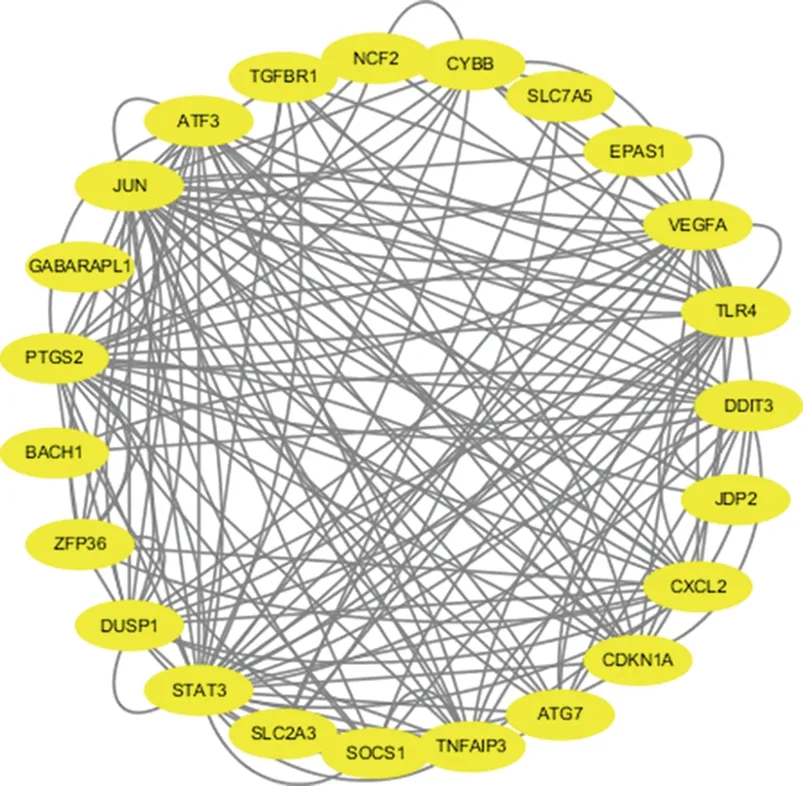
图4 PPI构建相互作用网络
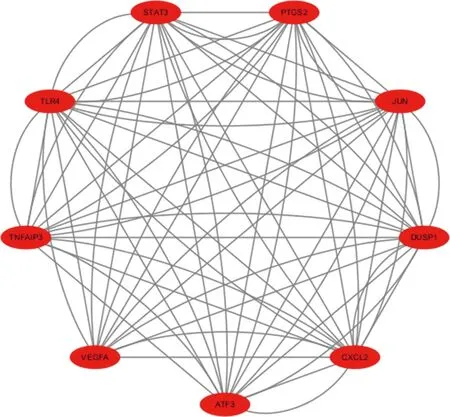
图5 mcode网络模块化分析
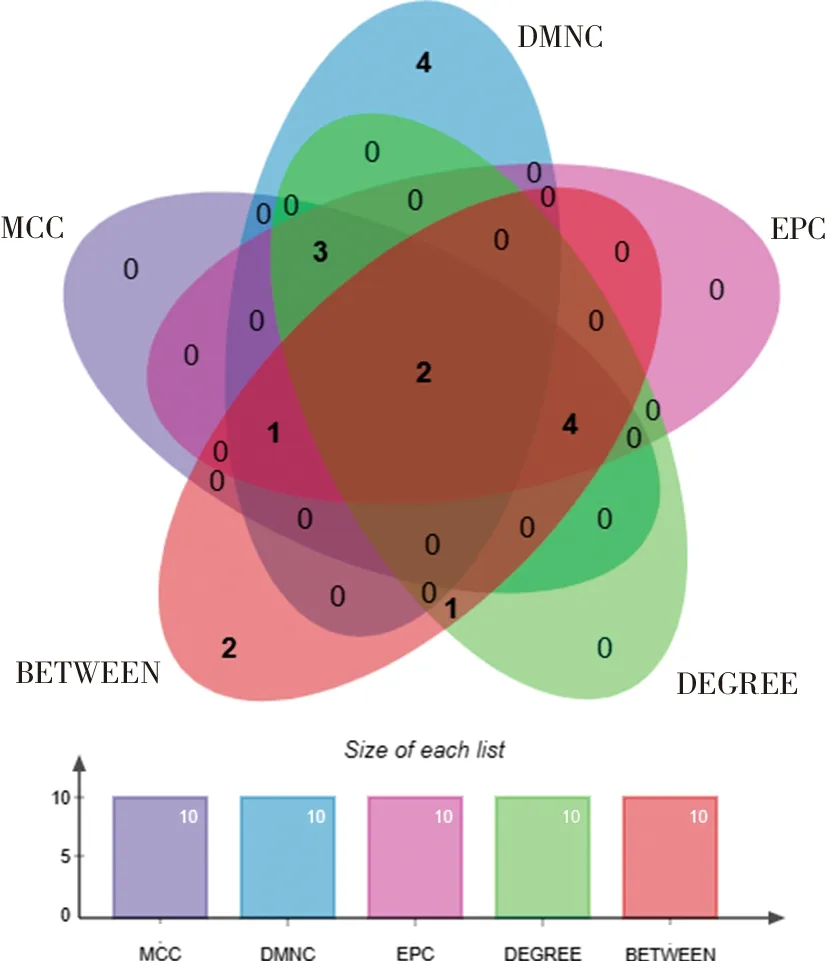
图6 5种算法中重叠的中心基因
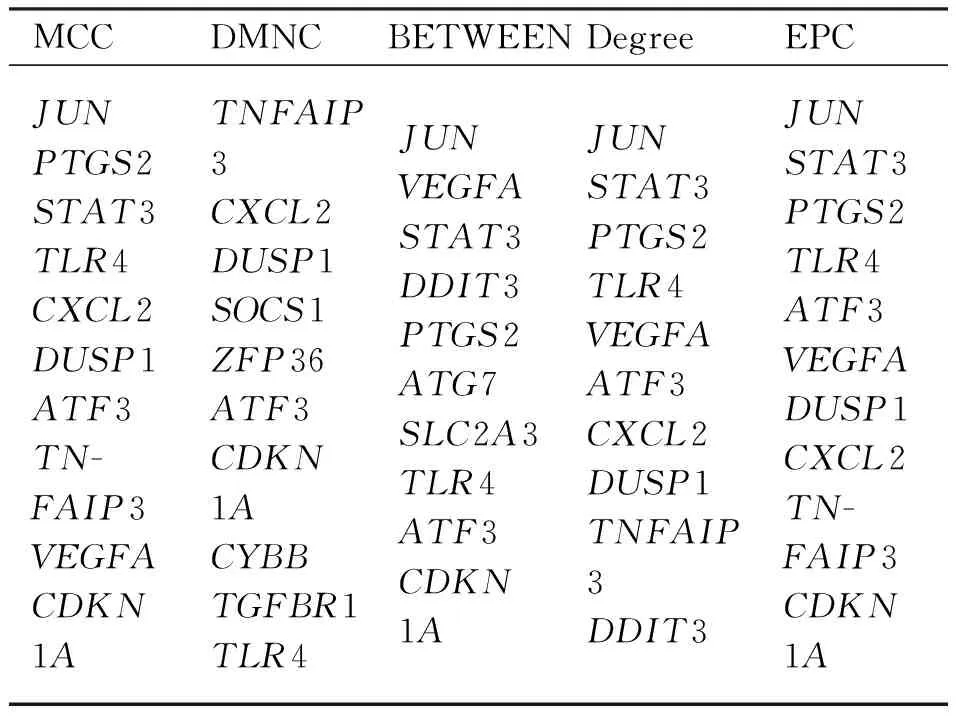
表2 5种算法获得的前10个hub基因
2.4 中心基因的蛋白质-蛋白质相互作用(PPI) 通过使用“GeneMania”工具来预测关键基因功能和可能的相互作用网络,构建了1个由 22 个基因组成的假定蛋白质-蛋白质相互作用网络,其中包括中枢基因TLR4和ATF3。PPI 网络总共包含 569个链接,网络上的基因本体分析表明,22个基因功能集中在细胞对应激反应、DNA模板转录对应激的反应、炎症反应、坏死凋亡、氧化应激上,与GO富集及KEGG富集分析相吻合,表明AMI的发病机制中可能发生氧化应激、炎性反应及坏死调亡等变化(图7)。
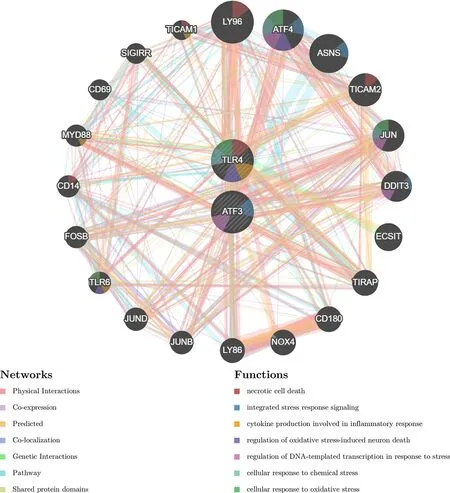
图7 预测中心基因的功能相似的基因PPI网络
2.5 潜在 miRNA 靶标调控网络的预测 筛选了簇 1 中的 13 个基因,并使用 miRWalk 2.0数据库检索了靶向真实中心基因的miRNA,发现hsa-mir-155-5p同时关联TLR4和ATF3基因,推测其可能是急性心肌梗死发病机制和发展中最关键的miRNA(图8)。
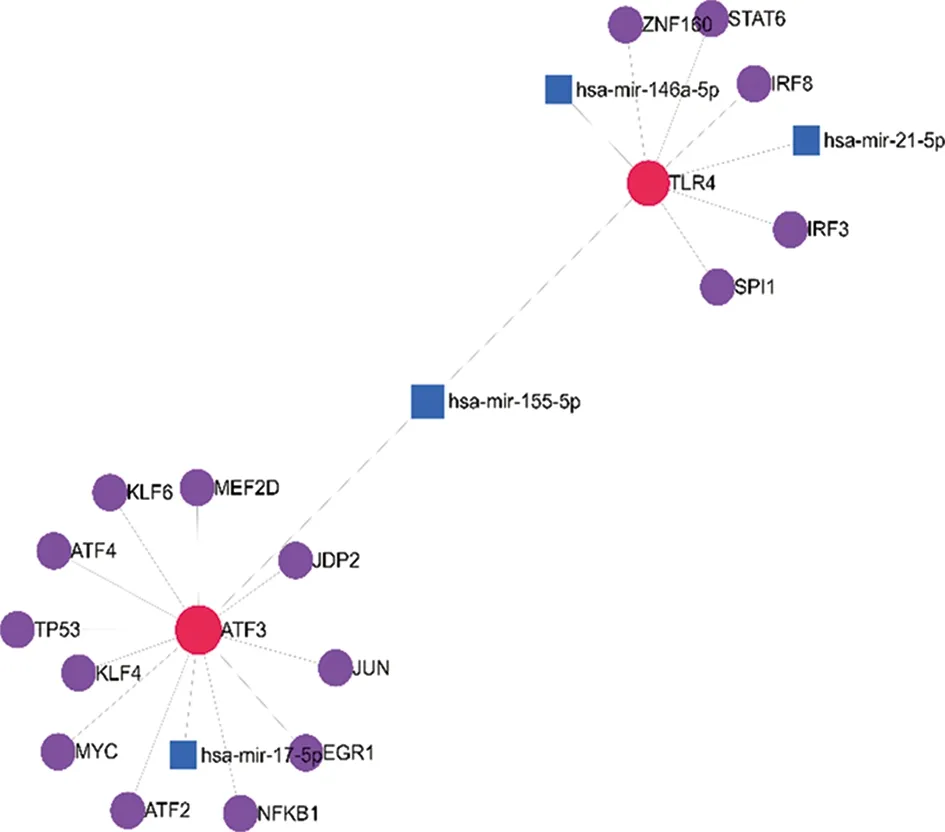
图8 关键基因与其靶向 miRNA 之间的相互作用网络
2.6 验证血清TLR4和ATF3浓度 通过ELISA验证急性心肌梗死患者与正常对照组之间血清中2种蛋白质的浓度,结果表明TLR4和ATF3表达水平明显高于正常组。为了评估TLR4和ATF3区分 AMI 组和对照组的能力,绘制了 ROC 曲线。根据结果TLR4的AUC值0.9700,ATF3的AUC值0.9850,表明鉴定的关键基因TLR4和ATF3展示了作为 AMI 潜在生物标志物的强大辨别能力(图9)。
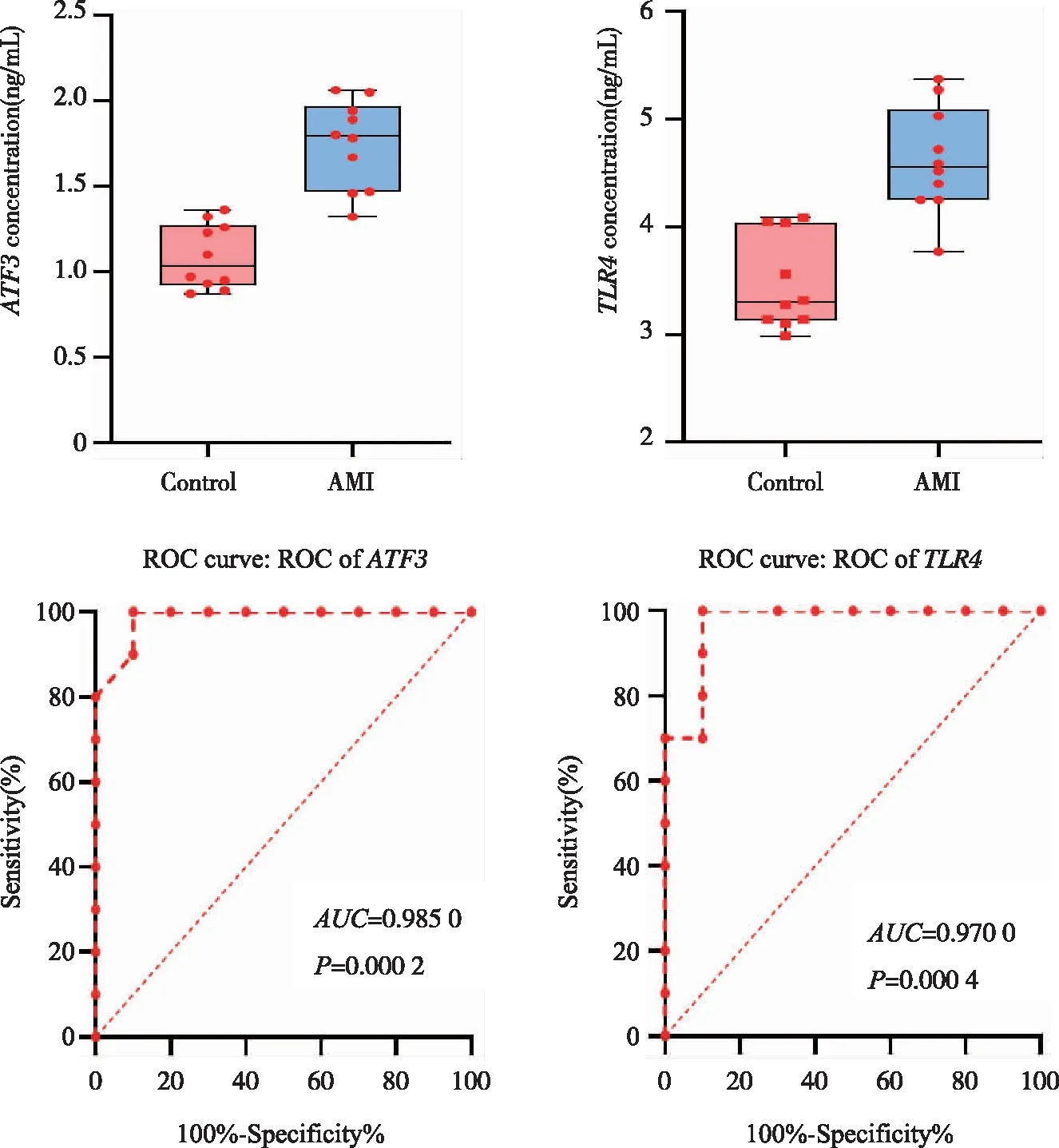
图9 TLR4和ATF3 箱式图与ROC曲线
3 讨论
急性心肌梗死是一种基因和环境相互作用而产生的疾病,可导致严重的心血管不良事件,尽管在诊断手段与治疗途径上取得了突破性进展,但是全世界发病率和死亡率都很高。早期及时的诊治对于改善急性心肌梗死预后至关重要,微阵列芯片技术的迅速发展和广泛应用为理解AMI发病机制提供了新的线索。急性心肌梗死后触发多种因素导致心肌细胞死亡,铁死亡已在急性心肌梗死中被发现,在AMI的发病过程中,铁死亡的作用不容忽视。因此,为了找到新的靶点或生物标志物,对铁死亡相关的关键基因研究就显得非常重要,这或许有助于从铁死亡方面进一步阐明AMI的发病机制。
本研究鉴定了急性心肌梗死中铁死亡相关的关键基因,从GEO数据库中下载了GSE66360数据集并与FerrDb 数据库取交集得到27个Ferroptosis DEGs,富集分析显示这些基因主要参与氧化应激反应和炎症反应、坏死凋亡以及脂质过氧化、动脉粥样硬化信号通路,这些结果与以往研究一致,脂质驱动的慢性炎症反应参与AMI的病理生理过程。利用STRING数据库,本研究构建了PPI网络并鉴定了2个关键基因TLR4和ATF3,这些基因也富含炎症相关途径,已有研究发现急性心肌梗死后铁死亡过程可通过激活TLR4信号通路,诱导炎性介质产生。此外,hsa-mir-155-5p调控网络可对急性心肌梗死患者TLR4、ATF3基因产生影响。本研究从生物信息学分析角度对急性心肌梗死病理机制提供有效的参考。
铁死亡最早于2012年被报道为细胞死亡的一种形式[7],主要通过铁依赖性的方式来调控细胞死亡,受脂质和氨基酸及其他各种代谢途径的调控[8-9],涉及脂质活性氧积累,引起氧化应激和炎症,促进动脉粥样硬化斑块和血管内皮的破坏[10]。氧化应激是动脉粥样硬化、心肌梗死、心律失常等心血管疾病的重要病理特征[11],铁死亡抑制蛋白(FSP1)是氧化还原酶,生成抗氧化剂,对氧化应激起抑制作用,证实氧化应激对铁死亡起重要作用[12]。NRF2靶基因在阻止脂质过氧化物形成及铁死亡发生发展中起关键作用,介导谷胱甘肽过氧化物酶4(GPX4)的合成,对细胞氧化应激反应及铁代谢过程有调节作用,证明氧化应激对铁死亡的影响显着[13]。
炎症和细胞死亡在急性心肌梗死发病机制中起关键作用,动脉粥样硬化是由脂质驱动的慢性炎症,血管内皮细胞、平滑肌细胞和巨噬细胞等参与其病理生理过程[14],Toll样受体4(TLR4)触发炎症途径的激活。已有研究发现,急性心肌梗死后铁死亡过程通过TLR4信号通路激活,并诱导炎性介质产生[15]。既往研究显示,急性冠状动脉综合征(ACS)组单个核细胞TLR4明显高于稳定型心绞痛 (SAP)组、冠状动脉慢血流(CSF)组及健康对照(HC)组,TLR4过度表达及其炎症反应可能与ACS的发生密切相关[16]。已有证据表明TLR4激活诱导脑缺血患者氧化应激反应,应用TLR4抑制剂阻断缺氧缺血性脑损伤(HIBD)的活性氧(ROS)产生,进一步降低了神经元炎性反应及铁死亡,表明TLR4在铁死亡中发挥重要作用[17]。心力衰竭时TLR4对其进展具有促进作用,敲除TLR4基因,左心室重塑有所改善,表明TLR4成为抑制心力衰竭患者心肌细胞铁死亡的治疗靶点[18]。
TLR4可用作炎性小体活化的辅助因子,由NF-κB活化诱导的促炎细胞因子,在小鼠横纹肌溶解模型上检测到炎症性趋化因子、脂质过氧化升高、抗氧化防御降低、肾小管细胞铁死亡,姜黄素可抑制TLR4/NF-κB轴降低炎症及氧化应激,从而实现对铁死亡的抑制作用,缓解肾脏损伤[19]。另一项研究发现,敲低TLR4可明显降低缺血-再灌注大鼠心脏中铁及脂质过氧化程度,减少心肌细胞中铁的死亡[20]。在本项研究中发现,TLR4是导致铁死亡的关键基因,这和以往生信分析结果相符[21]。
活化转录因子3(ATF3)是ATF/CREB转录因子家族成员,大量研究表明它的表达是由各种细胞应激迅速诱导,其中就包括DNA损伤、氧化应激与细胞损伤,广泛涉及调节细胞增殖、凋亡和炎症的作用[22-24]。有研究报道,ATF3与细胞应激反应有关,并通过抑制SLC7A11的表达而促进细胞铁死亡[25];此外,ATF3还通过促进过氧化氢和铁来促进神经胶质瘤细胞铁死亡[26],ATF3可通过活化c-CASP3,抑制SLC7A11、SOD2等促进鼻咽癌细胞的凋亡、铁死亡与ROS,这提示ATF3在铁死亡与凋亡构成的混合细胞中可能起着介导作用[27]。ATF3参与动脉粥样硬化的发生、发展,促进铁死亡作用可能成为未来治疗的靶点[28]。
miRNA生物学功能具有多样性,一种miRNA可具有多种靶基因,广泛涉及高血压、动脉粥样硬化、心力衰竭和其他心血管疾病[29]。本研究建立了miRNA靶点网络,以探讨急性心肌梗死后铁死亡机制的潜在miRNA。此网络提示hsa-mir-155-5p在急性心肌梗死时可能调控TLR4、ATF3基因的表达。有研究显示,miR-155通过靶向一氧化氮合酶(eNOS),加速动脉粥样硬化血管平滑肌增殖与迁移[30]。一项横断面调查研究显示,高血压组miR-155含量显着高于白大衣高血压组及正常高血压组,提示miR-155对血压升高有积极影响[31]。本研究结果表明在急性心肌梗死患者中可能存在miR-155靶向TLR4和ATF3基因的连接链,需进行进一步的实验验,证具体的分子机制。
本研究描述的2个铁死亡相关基因被明确标记为铁死亡驱动因素,在本组急性心肌梗死患者中得到验证。ROC曲线显示,TLR4和ATF3的AUC值>97%,表明TLR4和ATF3具有诊断潜力。本研究也存在一定的局限性,首先,仅通过生物信息学分析来检测相关功能,这需要更多的实验及临床试验来验证;其次,样本量太少,且外周血样本都是从本院采集的,应在多中心和大规模临床研究中确定。
综上所述,本文通过生物信息学分析得到了从铁死亡角度探索急性心肌梗死机制的关键基因,并进行了验证,证明了生物信息学分析的可靠性,这些结果为后续通过生物信息学分析联合基础实验探索铁死亡调节机制在急性心肌梗死发病机制中的作用提供了方向。



