郝若晨,唐敏嘉,刘光亮,张 艳,Muhammad Shoaib,尚若锋,曹宗喜*,蒲万霞*
(1.海南省农业科学院畜牧兽医研究所 海南省热带动物繁育与疫病研究重点实验室,海口 571100;2.中国农业科学院兰州畜牧与兽药研究所/农业农村部兽用药物创制重点实验室/甘肃省新兽药工程重点实验室,兰州 730050;3.中国农业科学院兰州兽医研究所,兰州 730046)
近十年来乳制品的生产和消费大幅增加,并存在强劲增长的势头,同时乳制品污染成为全世界非常关注的问题。乳制品含有丰富的营养成分,包括碳水化合物、蛋白质和矿物质,可以促进微生物的生长[1]。200多种已知的病原体可以通过食物传播,其中有几种肠杆菌科细菌属于对人类和动物会造成严重影响的重要食源性病原体,如产生毒素的沙门菌(Salmonella)、志贺菌(Shigella)和大肠埃希菌(Escherichiacoli,E.coli)[2]。食品中的致病性E.coli污染是一个公共卫生问题,这些细菌会造成严重的肠道疾病[3]。
细菌的致病力与其携带的毒力基因种类与数量高度相关。毒力基因种类繁多,包括黏附素、铁摄取系统、脂多糖、多糖荚膜和侵袭素等,这些毒力基因通常位于致病岛(pathogenicity islands,PAIs)、质粒和其它移动遗传元件(mobile genetic elements,MGEs)上[4]。高致病性毒力岛(high-pathogenicity island,HPI)是一种重要的毒力基因,其核心功能区由fyuA和irp2组成,因此fyuA和irp2是检测HPI的标志性基因[5]。ibeB是重要的致病因子[6],与免疫逃避相关的毒力基因主要包括荚膜、补体抗性蛋白和外膜蛋白酶等,ompA、ompT、traT、cvaC和iss分别编码外膜蛋白、外膜蛋白酶、补体抗体蛋白、定殖因子以及抗吞噬作用因子[7]。铁是许多细菌生长所必需的,摄铁系统是细菌重要的生存机制,iroN编码铁载体受体,iucD编码NADPH依赖性赖氨酸N(6′)-单加氧酶,该酶与铁载体生物合成蛋白相关[7]。
质粒是能够在细胞之间进行自我传递的自主DNA分子,携带的基因对宿主的生长或生存是不必要的[8]。几乎所有的细菌都有质粒,它们可以垂直传播到后代,也可以水平传播。质粒通过转座子或插入序列等MGEs获得新基因,并能够在广泛的宿主中复制,使它们成为肠杆菌科细菌的耐药性(antimicrobial resistance,AMR)和致病性传播的完美载体[9]。六种主要的质粒家族已被证明可以介导肠道细菌物种之间的抗菌素耐药性传播,即Inc F、Inc A/C、Inc L/M、Inc N、Inc I和Inc HI2[10]。由于毒力因子可增加细菌存活率并可能影响抗生素耐药性表达[11],因此,对不同细菌宿主中毒力基因和质粒特征的鉴定是了解细菌致病力和耐药性的基础。
生物被膜是由细菌在生长过程中为适应生存环境而黏附于物体或活性组织表面并包被其自身而产生的细胞外多糖基质形成的,是细菌的特殊存在形式。它的形成一方面促进细菌逃避机体免疫系统的作用,阻碍细胞吞噬或减少吞噬作用与氧活性降低后的应激反应[12];另一方面可以阻止或延缓药物的渗透,生物被膜内细菌的生理学特点影响了细菌对药物的敏感性[13-14]。生物被膜结构可以破坏人类的防御系统,为微生物提供庇护所,导致免疫逃避和细菌耐药性[15]。E.coli、肺炎克雷伯菌(Klebsiellapneumoniae,K.pneumoniae)、Salmonella、铜绿假单胞菌、金黄色葡萄球菌、炭疽杆菌等大多数细菌能产生生物被膜[16]。美国国立卫生研究院表示80%的慢性感染与生物被膜形成相关[17],因此检测细菌生物被膜形成能力是了解其致病力和耐药性的另一个主要的生物学特性。
了解特定地理区域内流行菌株的遗传关系对于疾病的预防是至关重要的。肠杆菌科细菌基因间重复一致序列-聚合酶链式反应分型(enterobacterial repetitive intergenic consensus-PCR, ERIC-PCR)技术是一种用于细菌流行病学分析和基因分型的分子方法,基于基因间重复共有序列进行PCR扩增,由条带数目及大小进行分型,可以评估菌株的亲缘关系和遗传多样性。我国要将海南省建设为面向全球的自由贸易试验区,食品安全问题就显得尤为重要,但海南省奶牛养殖业欠发达,目前很少有研究聚焦于牛奶生产环境中被细菌污染相关的风险因素。
因此本研究从海南省两个奶牛场采集鲜乳、工人手臂、挤奶设备、牛舍护栏、粪便和商品奶样本,共52份。对其进行了样本中肠杆菌科细菌的分离与鉴定、肠杆菌科细菌质粒型的检测、肠杆菌科细菌生物被膜表型的检测以及肠杆菌科细菌基因间重复序列分子分型,旨在探明海南省肠杆菌科细菌在生乳、商品奶及奶牛养殖环境的分布及分型情况,分析菌株的遗传关系。
1 材料与方法
1.1 试验材料
PCR和全自动紫外凝胶成像仪(美国Applied Biosystems公司);移液器、高速离心机和紫外分光光度计(德国Eppendorf公司);电泳槽和高压电泳仪(北京六一仪器厂);多功能酶标仪(美国Gene公司);琼脂糖购自西班牙Biowest公司;刚果红培养基(Congo Red Agar,CRA)购自上海中秦化学试剂有限公司;Premix TaqTM、10×Taq Buffer(Mg2+)和Taq酶(5 U·μL-1)均购自宝生物工程(大连)有限公司(大连TaKaRa公司)。
1.2 样品采集
样品于2021年采集自海南省两个奶牛养殖场,其中A牛场养殖规模为200头,采集健康奶牛鲜乳10份、工人手臂拭子4份、挤奶设备拭子4份、粪便5份和商品奶8份;B牛场养殖规模为100头,采集健康奶牛鲜乳9份、工人手臂拭子3份、牛舍护栏拭子7份和粪便2份。
奶样采集时,先将乳房擦拭消毒,丢弃前3把奶,分别采集每头牛4个乳区的奶3~4 mL,混合后为一份样品;采集工人手臂、牛舍护栏以及挤奶设备源样品时,先将灭菌棉签蘸取适量生理盐水,然后用棉签分别在工人手臂、牛舍护栏以及挤奶设备处涂抹,放入有培养基的采样管中;粪样采用五点采样法收集。
1.3 细菌分离鉴定和去重
称取粪样100 g,加入400 mL灭菌BHI肉汤,混匀,吸取5 mL上清液接种于100 mL灭菌BHI液体培养基中,37 ℃ 180 r·min-1增菌15 h。吸取1 mL奶样,接种至9 mL BHI肉汤中进行增菌。无菌条件下蘸取增菌液,在MAC平皿上进行单划线接种,37 ℃静置培养18 h,挑取形态不一致的单菌落接种至MAC上培养,直至菌落形态单一。采用水煮法提取细菌基因组DNA,以纯化菌株的DNA为模板,进行ERIC-PCR扩增,ERIC-PCR反应体系为25 μL:10×Taq Buffer(Mg2+)2.5 μL、dNTP 2 μL、Taq酶1 μL、ERIC引物各0.5 μL(10 μmol·L-1),模板DNA 1.5 μL,ddH2O 17 μL。反应条件:94 ℃预变性5 min,94 ℃变性45 s,52 ℃退火1 min,72 ℃延伸5 min,35个循环;72 ℃最后延伸10 min。产物在2.0%琼脂糖凝胶上进行电泳并拍照,将分离自同一样品中条带数目、大小完全一致的菌株视为重复,仅保留一株。以纯化非重复菌株DNA为模板,扩增其16S rDNA序列,PCR反应体系(25 μL):Premix TaqTM12.5 μL,上下游引物各1 μL(10 μmol·L-1),模板1 μL,加ddH2O至25 μL。反应条件:94 ℃预变性5 min;94 ℃变性30 s,退火30 s,72 ℃下1 kb·min-1延伸,30个循环;72 ℃延伸5 min。扩增产物在1.0%琼脂糖凝胶上进行电泳并拍照记录结果。将符合预期大小的PCR产物送上海生工生物有限公司进行测序,在NCBI中将测序结果进行BLAST比对分析,按相似性≥99%判定,确定分离菌株的种属地位。经分离、纯化与鉴定的非重复性肠杆菌科细菌科菌株,于-70 ℃超低温冰箱保存菌种。
1.4 肠杆菌科细菌的毒力基因检测和质粒分型
根据参考文献合成了11种毒力基因的引物,引物由北京擎科生物有限公司合成,引物序列、扩增片段大小及退火温度见表 1。对49株肠杆菌科细菌菌株进行ibeB、iucD、iroN、fyuA、irp2、ompA、ompT、traT、iss、cvaC和tsh毒力基因的检测。与铁转运相关的fyuA和irp2基因采用双重PCR检测,反应体系为20 μL:fyuA和irp2基因的上下游引物各0.5 μL(10 μmol·L-1),Premix TaqTM10 μL,模板1 μL,再补充去离子水至20 μL。其余毒力基因的检测均使用单反应PCR方法,反应体系与反应条件与“1.3 ”中16S rDNA扩增体系一致。以实验室保存的携带相应特异性基因的菌株作为阳性对照,灭菌超纯水为空白对照。在1%的琼脂糖凝胶上电泳,然后使用全自动凝胶成像系统拍照并记录结果。
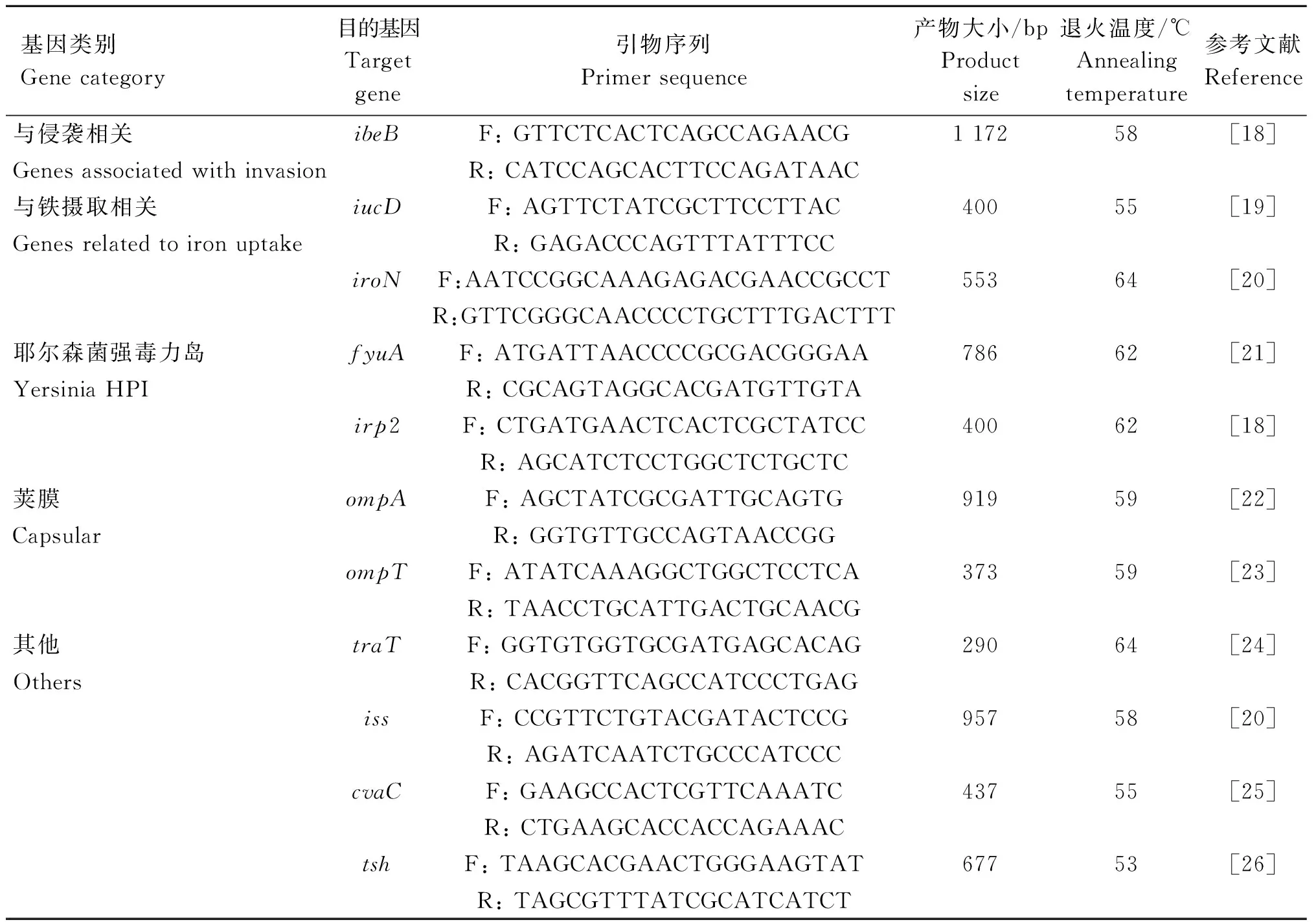
表1 毒力基因引物序列Table 1 Primer sequences of virulence genes
采用Carattoli等[27]开发的基于肠杆菌科主要质粒不相容性组的复制子分型(PCR-based replicon typing,PBRT),分析所分离肠杆菌科菌株的质粒携带情况。根据参考文献合成了识别parA-parB、iterons和RNAI 等目标位点的HI1、HI2、I1、X、L/M、N、FIA、FIB、W、Y、P、FIC、A/C、T、FII、F、K和B/O等18种常见的复制子引物[27],通过PCR方法对上述菌株携带的质粒型进行检测。该方法包括5组多重PCR和三个单重PCR反应,其引物序列、扩增片段大小及退火温度见表2。以细菌总DNA为模板,使用可识别Inc复制子的18种引物进行基于PCR的复制子分型。
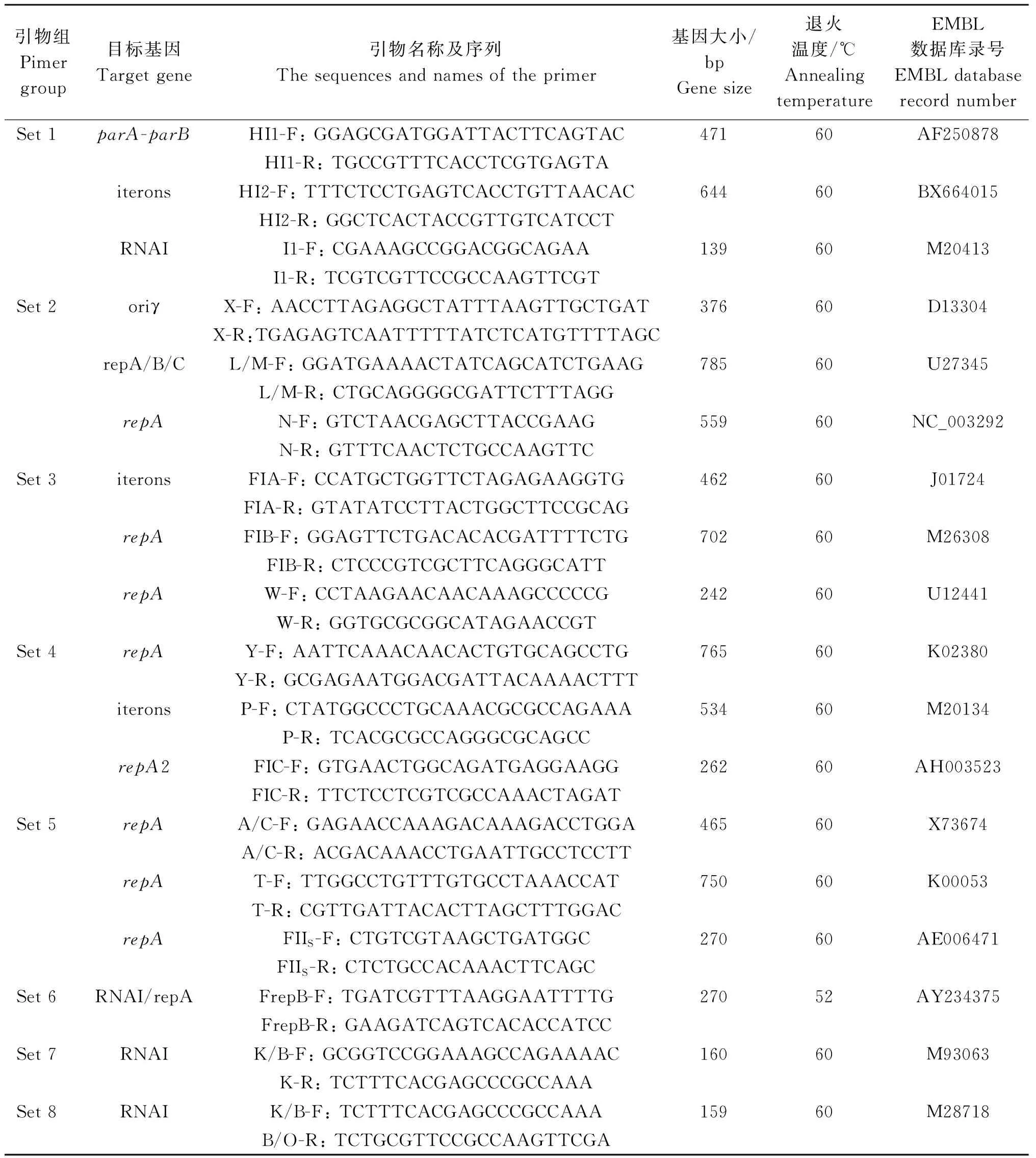
表2 质粒分型引物信息Table 2 Plasmid typing primers information
质粒分型基因检测的反应体系:上下游引物各0.4 μL(10 μmol·L-1),DNA模板1 μL,Premix TaqTM10 μL,ddH2O 8.2 μL,总体积为20 μL。质粒分型PCR的set 1~5和7~8反应条件:预变性94 ℃ 5 min;94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸1 min,35个循环;最后72 ℃延伸7 min。质粒分型PCR set 6的反应条件:预热变性94 ℃ 5 min;94 ℃变性30 s,52 ℃ 退火30 s,72 ℃延伸1 min,35个循环;最后72 ℃延伸7 min。
1.5 菌株生物被膜定性检测
复苏菌株,将复苏后的菌株划线接种于CRA上,37 ℃培养22~24 h,观察菌落在培养基上的颜色和菌落形态。如果菌落在CRA上呈干燥、黑色、光亮结晶和周边培养基褪色,指示为生物被膜阳性菌株;菌落在CRA上呈湿润、红色和周边培养基不褪色,指示为生物被膜阴性菌株。
1.6 肠杆菌科分离株的ERIC-PCR分子分型
以所分离菌株的基因组DNA做模板,利用ERIC引物对其进行PCR扩增,扩增产物在2%的琼脂糖凝胶上进行电泳,使用DL5000 DNA Marker,电泳后用紫外凝胶成像系统观察并拍照记录,并通过BioNumerics软件进行遗传进化关系分析。
2 结 果
2.1 细菌分离鉴定
52份样本中有31份样本中能获得可培养的菌落,总分离率为59.6%(31/52),共分离77株菌。分别为A牛场分离率64.5%(20/31),分离得到57株菌;B牛场分离率52.4%(11/21),分离得到20株菌。粪样的分离率最高,为100.0%(7/7);其次为鲜乳样,分离率为78.9%(15/19),其中A牛场为90.0%(9/10),B牛场为66.7%(6/9);工人手臂源样本的分离率为57.1%(4/7),其中A牛场为75.0%(3/4),B牛场为33.3%(1/3);挤奶设备或牛舍护栏源样本的分离率为36.4%(4/11),其中A牛场为50.0%(2/4),B牛场为28.6%(2/7);商品奶样本的分离率为12.5%(1/8)(表3)。整体而言,A牛场的分离率较B牛场高,需要提到的一点是,B牛场无商品奶。
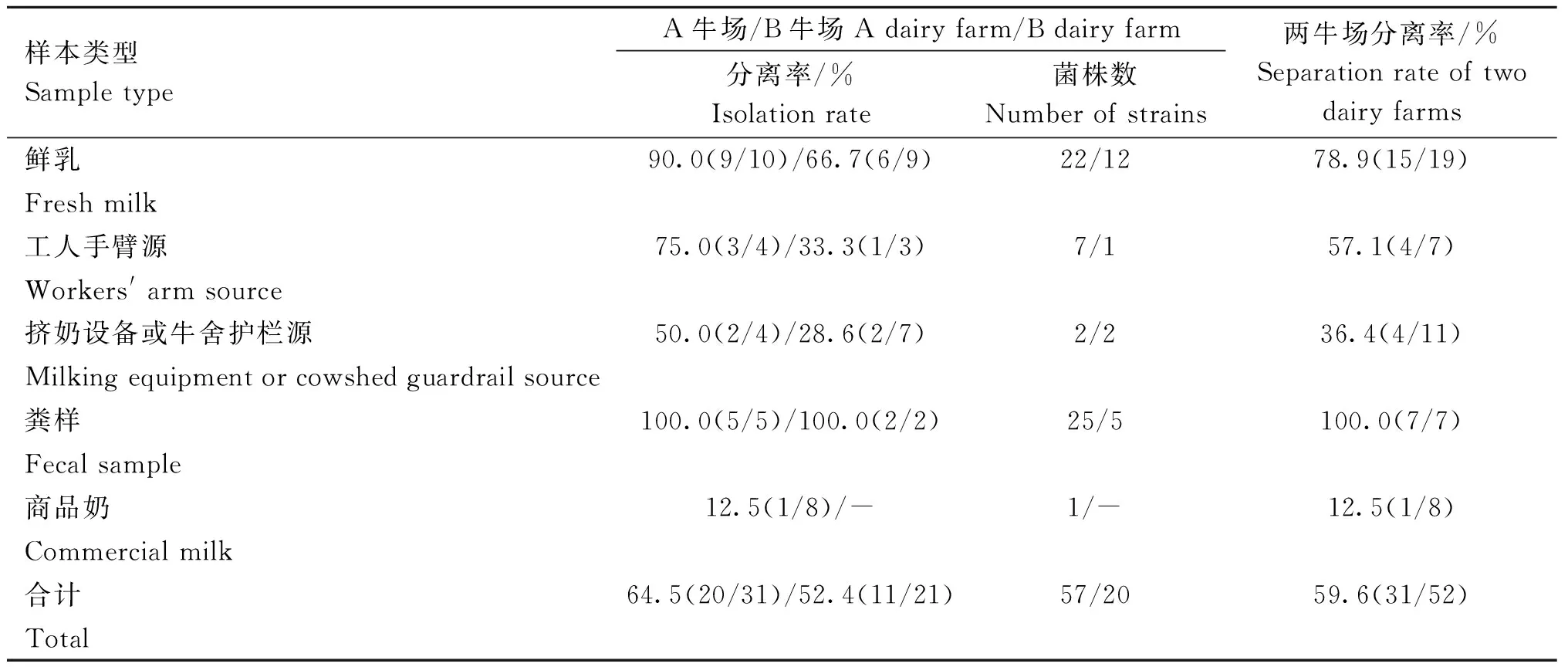
表3 不同来源样本细菌分离率Table 3 Bacterial isolation rate of samples from different sources
将疑似菌株的16S rRNA序列与NCBI数据库进行BLAST比对,鉴定结果如表4所示。肠杆菌科为49株,其它菌株24株,NCBI数据库中未鉴定的有4株。在鉴定的所有物种中,E.coli的检出率最高,为41.6%(32/77);其次为K.pneumoniae,检出率为16.9%(13/77)。阴沟肠杆菌(Enterobactercloacae,E.cloacae)的检出率为3.9%(3/77);产气肠杆菌(Enterobacteraerogenes,E.aerogenes)检出率为1.3%(1/77)。
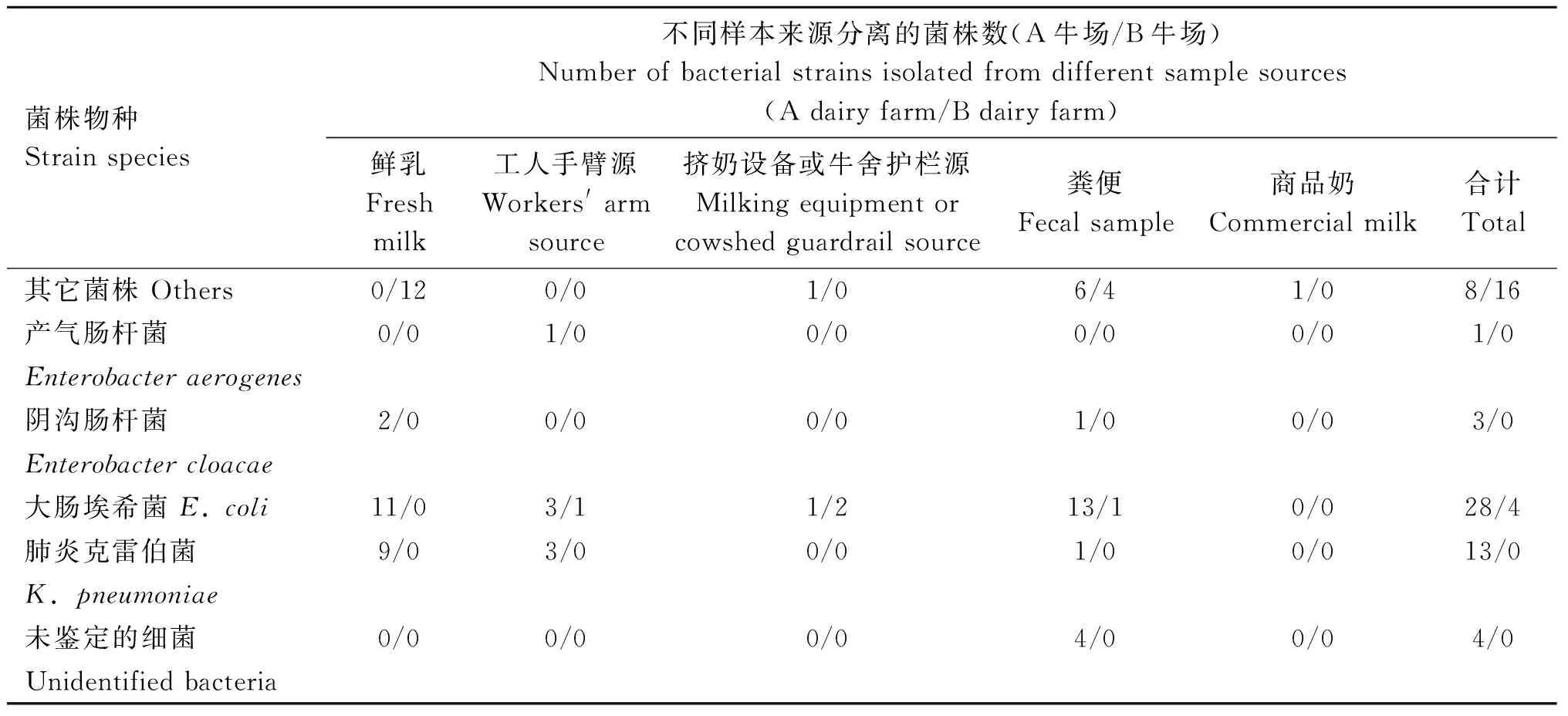
表4 不同来源样本细菌分离鉴定情况Table 4 Isolation and identification of bacteria from different sources
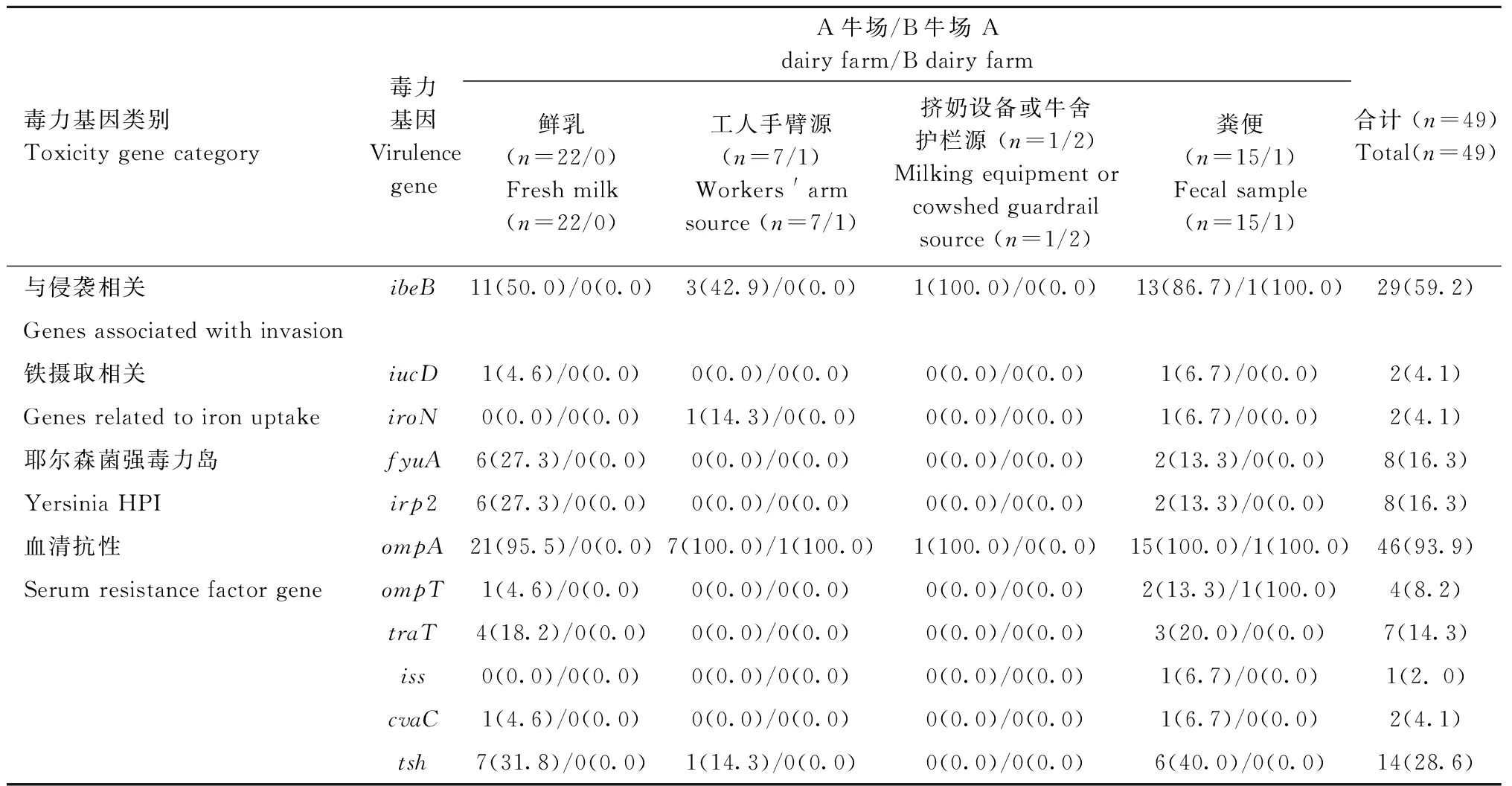
表5 毒力基因在不同样品来源分离株中的分布Table 5 Distribution of virulence genes in isolates from different sample sources
2.2 肠杆菌科细菌的毒力基因和质粒分型
毒力基因结果显示,ompA的检出率最高,为93.9%。A牛场鲜乳源菌株中ompA的检出率最高,为95.5%(21/22),未检出iroN和iss基因。A牛场工人手臂源菌株中,ompA的检出率最高,为100.0%(7/7),未检出iucD、fyuA、irp2、ompT、traT、iss和cvaC基因。A牛场挤奶设备或牛舍护栏源菌株中只检出了ibeB和ompA基因。A牛场粪源菌株中,ompA的检出率最高,为100%(15/15)。B牛场中工人手臂源菌株中只检出了ompA基因,粪源菌株中检出了ibeB、ompA和ompT基因。
质粒型检测结果显示(表6),K质粒的阳性率最高,为44.9%(22/49);其次为W(40.8%,20/49)、FIB(36.7%,18/49)、Y(18.4%,9/49)、FrepB(14.3%,7/49)、FIA(6.1%,3/49)和HI2(2.0%,1/49)质粒。本研究中未检测到HI1、I1、L/M、N、X、P、FIC、T、A/C、FIIS和B/O质粒。2.0%的菌株携带6个质粒,4.1%的菌株携带5个质粒,12.2%的菌株携带4个质粒,20.4%的菌株携带3个质粒,6.1%的菌株携带2个质粒,8.2%的菌株携带1个质粒,46.9%的菌株未携带所检测的质粒。携带质粒的菌株中,96.2%(25/26)为E.coli,3.8%(1/26)为E.aerogenes。
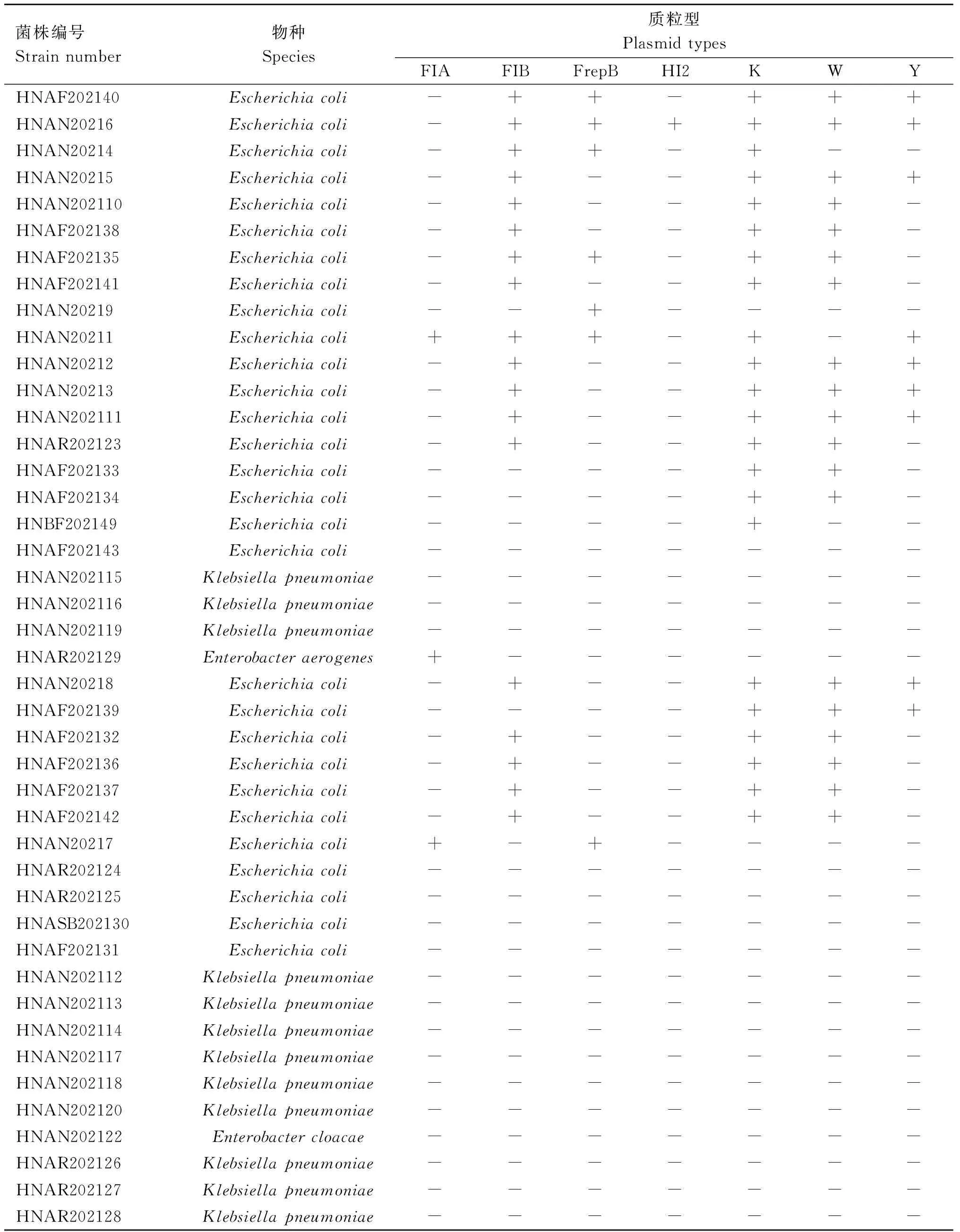
表6 分离菌株携带质粒型情况Table 6 plasmid type of isolated strains
2.3 菌株生物被膜定性
49株肠杆菌科细菌中有37株(75.5%,37/49)在CRA上有不同程度的黑色菌落出现,指示具有生物被膜形成能力,见图 1a。其中E.coli20株(62.5%,20/32),K.pneumoniae13株(100.0%,13/13),E.cloacae3株(100.0%,3/3),E.aerogenes1株(100.0%,1/1)。其余12株(24.5%,12/49)在CRA上为红色菌落,没有生物被膜形成能力,见图 1b,均为E.coli。

a. 刚果红染色阳性;b. .刚果红染色阴性A. Congo red-positive;b. Congo red-negative图1 刚果红琼脂上培养的菌株外观Fig.1 The appearance of strains cultured on Congo red agar
2.4 肠杆菌科分离株的ERIC-PCR分子分型
以条件位置差异容许度为1.5%,优化值为1.5%对ERIC-PCR指纹图谱进行分析,依据UPGMA方式进行聚类,以70%为标准划分[28],最终49株肠杆菌科菌株被划分为Ⅰ-ⅩⅦ共17型,Ⅵ型为优势型,共20株,其次为ⅩⅤ型(6株)、Ⅷ(3株)、Ⅸ(3株)、Ⅳ(2株)、Ⅹ(2株)、ⅩⅠ(2株)和ⅩⅢ(2株),剩余的9株菌分布在其它9种型中,聚类关系见图 2。Ⅵ型全部为E.coli,其中19株分离自A牛场(包括鲜乳、工人手臂源和粪源),1株分离自B牛场粪源,说明A牛场菌株的聚类关系较近。ⅩⅤ型全部为K.pneumoniae,均分离自A牛场(包括鲜乳和工人手臂源)。

图2 肠杆菌科细菌ERIC-PCR基因分型的聚类树图Fig.2 Cluster tree of ERIC-PCR genotype of Enterobacteriaceae
3 讨 论
一些致病性肠杆菌科细菌不仅给畜牧业发展造成危害,还威胁人类健康,而牛是致病性肠杆菌科细菌引起人类感染(牛肉、乳制品、牛粪便污染)的重要来源[29]。因此,了解牛养殖环境肠杆菌科细菌流行情况对动物和人类预防疾病和维护健康至关重要,海南省的养殖业欠发达,对养殖环境和商品奶中肠杆菌科细菌的研究较少,故本研究以养殖环境和商品奶的肠杆菌科细菌为研究对象。本研究中鲜乳样细菌的分离率较高,为78.9%,其中A牛场更是高达90.0%。分离率较高的原因可能是样本来源牛场中奶牛饲养密度和活动量较大,接触污染源多,未及时清洗或未彻底清洗乳头;除此之外,海南省气候湿润,气温高,也容易滋生细菌。本研究中工人手臂源样本和挤奶设备或牛舍护栏源细菌的分离率分别为57.1%和36.4%,可能是因为挤奶工人或挤奶过程中消毒不当,这会导致牛奶或其衍生物被污染,必须引起我们足够的重视,因为摄入受病原体污染的牛奶或其衍生物可能会导致严重的人类感染,提示应对食品行业的工人提出安全和耐药性的建议和持续教育,以防止食品污染和耐药菌株的出现。对饲养环境勤消毒,勤通风与勤打扫,以及及时正确的清洁手臂和挤奶设备等。商品奶的分离率为12.5%,主要的原因是运输过程中样本意外暴露,但未及时处理导致样本被污染。
自然界中E.coli分布广泛,故本研究中,E.coli检出率最高,为41.6%。该菌可以通过粪便和废水等途径排放到环境中,由食物链进入人体,但大多数E.coli都是动物肠道中的正常寄居菌,只有很小一部分才会在一定条件下引起疾病。K.pneumoniae是一种革兰阴性菌,也是机会性和环境性病原体,广泛存在于动物黏膜、水和土壤等环境中,不仅导致奶牛乳腺炎,还会引起医院医疗相关感染[30]。在本研究中,A牛场的鲜乳、工人手臂源和粪样中均能分离得到K.pneumoniae。随着消费鲜乳的趋势,A牛场的鲜乳和工人手臂上分离到该菌必须引起我们足够的重视,因为携带该菌的鲜乳被人类食用后可能会引起严重的感染,而手臂上存在该菌也有可能造成携带者的感染或者进一步的传播。
为进一步确定分离株的致病潜力,又对其毒力基因进行了检测。有研究显示,只要同时含有papC、iucD、irp2、tsh、vat、aatA、iss和cva8个基因中4个以上即为强毒株[28],本研究中来自A牛场的粪源E.coli菌株HNAF202140同时携带了所检测的11个毒力基因(ibeB、iucD、iroN、fyuA、irp2、ompA、ompT、traT、iss、cvaC、tsh),说明该菌株存在着较大的致病风险。A和B牛场毒力基因检测率最高的是ompA。研究报道ompA是不同毒力基因中的流行率最高,最常见的基因,它在生物膜形成、维持膜完整性和抗生素转运中发挥着重要作用[31],这会加强细菌的抗生素耐药性,不仅如此,该基因中的L3位点可能与K.pneumoniae的致病性有关[32],所以ompA在本研究中的高检出率需要引起我们高度重视。目前,已有一些关于基因敲除技术的研究表明,iucD和E.coli的致病力存在着某种联系[33]。贾青辉等[34]报道鸡源致病性E.coli中iucD的检出率为95.2%,而在本研究中iucD的检出率为4.1%,明显低于上述报道,说明海南省奶源和养殖环境中致病性菌株较少,向环境、人或其它动物传播致病风险的可能性较低。徐政平[35]报道的APEC中的iss的检出率为69.8%;在Manita等[36]的研究中,APEC分离株中ompT和iroN的检出率为100%;De Carli等[37]报道的APEC菌株中ompT和iroN的检出率分别为100%和98.8%。本研究中iss、ompT和iroN的检出率分别为2.0%、8.2%和4.1%,远远低于上述报道。Asai等[38]发现这5种毒力基因(iutA、hlyF、iss、iroN和ompT)与APEC的高致病力相关,而本研究中的E.coli并未确定为APEC,这可能是iss、ompT和iroN的检出率较低的原因。总体来看本研究中毒力基因总体检出率相较于其它报道低,可能是因为本研究样品来源于养殖环境和鲜乳样。
病原菌对抗菌素的耐药情况可以从调查菌株中质粒携带情况入手。PBRT具有较高的特异性和灵敏性,但该方法也存在着一定的局限性,只能检测18种常见的质粒,对于一些新型的或变异的质粒可能无法识别。本研究中质粒类型多样,其中Inc K、Inc W和Inc FIB为优势质粒,这些优势质粒也经常在动物源E.coli中发现[39-40]。有研究显示携带AMR基因的质粒主要在典型的Inc组质粒上[30]。其中Inc K质粒主要与blaCMY-2和blaCTX-M-14抗性基因在欧洲传播有关,尤其是西班牙和英国。而CMY型和CTX-M型β-内酰胺酶是E.coli中最常见的超广谱β-内酰胺酶(the extended-spectrum β-lactamases,ESBLs)家族,但是产生 CMY 和 CTX-M 的肠杆菌科的直接人畜共患食源性传播并不常见[41]。表明本研究中奶牛场的E.coli危害人类健康的可能不大。Inc W质粒被认为是最小的接合质粒,1980年代,在许多细菌中发现了Inc W质粒[42]。研究显示,Inc W质粒携带氯霉素、四环素、磺胺类、庆大霉素和甲氧苄啶等耐药基因(antimicrobial resistance genes,ARGs),也有研究显示携带blaKPC-2和blaVIM-1抗性基因[43-44],这两个抗性基因都与碳青霉烯类药物耐药性有关[43]。Inc F组质粒是大小在45~200 kb的低拷贝接合质粒,Inc F质粒上最常见的耐药基因有编码ESBLs、碳青霉烯酶、氨基糖苷类修饰酶的基因[45]。这些质粒不仅会对细菌的生存做出贡献,其介导的耐药机制也是抗生素耐药性增加的原因。
CRA是生物被膜定性常用的方法之一。本研究生物被膜定性检测结果显示49株肠杆菌科细菌中有37株指示具有生物被膜形成的能力,在E.coli、K.pneumoniae、E.cloacae、E.aerogenes中都能发现生物被膜形成的能力,而且E.coli中也有无生物被膜形成能力的菌株,表示生物被膜形成能力似乎不是菌种依赖性的。Staji等[46]发现,ERIC-PCR检测结果中同一来源分离菌株基因存在差异,不同来源也可有较近的亲缘关系。郑晓风等[47]用ERIC-PCR方法发现菌株间存在交叉传播。本研究ERIC-PCR检测结果显示49株肠杆菌科细菌被分为17型,参照我国华东地区的103株临床乳房炎E.coli被分为10型[48],可见本研究中肠杆菌科细菌广泛的DNA多样性。DNA多样性的原因可能是频繁使用抗生素导致的选择性压力引起的基因修饰。Ⅵ型菌株来源于鲜乳、粪便和工人手臂源,XⅤ型来源于鲜乳和工人手臂,说明粪便和工人手臂是鲜乳污染或者乳腺感染的主要来源,会进一步导致乳腺炎的发生。
4 结 论
本研究样品的总分离率为59.6%(31/52),共分离出77株菌,不同来源的样品均有分布,粪便检出率最高,为100.0%;其次为鲜乳,检出率78.9%。经鉴定疑似菌株覆盖了5个科11个种,主要为肠杆菌科。所分离肠杆菌科细菌毒力基因的检测中ompA的检出率最高,为93.9%,其余毒力基因均有不同程度的检出;质粒类型多样,携带3个质粒型的菌株较多,携带质粒的菌株中,96.2%为E.coli,其中75.5%指示有生物被膜形成能力;分子分型中Ⅵ型为优势型,全部为E.coli;其次是ⅩⅤ型,全部为K.pneumoniae。肠杆菌科细菌在奶源环境中广泛存在、生物特性多样,推测具有一定的致病力和耐药性。



